博文
【文献速递】变废为宝:利用机器学习从宏基因组中挖掘污泥中的抗菌肽
|
文章题目:Waste to resource: Mining antimicrobial peptides in sludge from metagenomes using machine learning
文章链接:https://doi.org/10.1016/j.envint.2024.108574
发表时间:2025-05-06
期刊:Environment International
影响因子:10.3
一、研究背景
耐抗生素细菌的出现对感染的治疗构成了巨大的威胁。抗菌肽(AMPs)是一类广泛存在于生物体中的短肽,被认为是传统抗生素的潜在替代品。AMPs的早期发现依赖于天然代谢物筛选,即对大量生物样品进行分离纯化,然后采用液相色谱-质谱法检测其有效成分。传统的方法既耗时又费力,而且不能确保肽的活性。因而组合化学和高通量筛选技术的出现为抗菌肽的研发开辟了一个新的研究领域。作为典型的工程环境,城市污水处理厂收集了大量的生活污水和工业污水,是抗生素耐药基因和多重耐药细菌的储存库。因此,通过对污泥的资源挖掘,有可能找到对致病菌有效的抗菌肽。基于此,本研究通过宏基因组学结合机器学习来挖掘污泥中的AMP。
为开发更多的抗菌肽来替代抗生素,研究人员利用宏基因组学结合机器学习从环境宏基因组中寻找抗菌肽,并成功获得了16,044,909个预测AMP。研究者比较了自然环境和人工环境中潜在抗菌肽的丰度,发现人工环境在提供抗菌肽方面也具有很大的潜力。此外,研究人员选择污泥作为典型的人工环境样本,并试图从中提取抗菌肽。通过宏蛋白质组分析和相关性分析,研究者从污泥中挖掘出27个候选AMP。通过化学合成方法成功合成了25个多肽,并通过实验验证了其中21个多肽对4种菌株具有抑菌活性。此项研究强调了从人工环境中挖掘新的抗菌肽的潜力,并证明了从污泥中挖掘抗菌肽的有效性。
二、技术路线
数据收集与预处理:
从公共数据库获取52,515个宏基因组组装基因组(MAGs),筛选出4,867个高质量MAGs(自然与工程环境,排除宿主相关)。
环境分类:自然(水体、土壤等) vs. 工程(污水厂、反应器等)。
AMP预测流程:
ORF预测:使用 EMBOSS getorf 工具从MAGs中提取非冗余小开放阅读框(sORFs, 5-50 AA)。
机器学习预测:采用集成模型(LSTM + Attention + BERT)预测AMP,阈值 >0.5 视为潜在AMP。
污泥AMP筛选与验证:
宏蛋白质组交叉验证:比对污泥宏蛋白质组数据,筛选表达肽段(>50%序列匹配)。
相关性分析:基于污泥宏基因组(66样本),计算AMP与微生物丰度的负相关性(p<0.05)。
实验验证:化学合成候选肽,测试对4种病原菌(B. subtilis, S. aureus, E. coli, P. aeruginosa)的最小抑菌浓度(MIC)。
生物信息学分析:
相似性评估:比对公共AMP数据库(CAMP, DRAMP等),计算序列相似性。
抗菌活性对比:与数据库AMP的MIC值比较。
三、主要研究结果
研究人员主要利用宏基因组学结合机器学习来挖掘微生物组中的AMP。并通过预测和分析自然环境和人工环境的宏基因组比较了不同环境中潜在AMP的丰度。此外,研究者还借助污泥宏蛋白质组和宏基因组进行后续的筛选。对从污泥中挖掘出的抗菌肽,采用化学合成的方法进行合成,并通过抗菌实验验证了其抗菌活性。
本项研究证明了基于机器学习的方法从污泥微生物群中挖掘AMP的能力。研究人员化学合成了筛选获得的抗菌肽,并证实其中至少84%具有抗菌作用。对于其他环境样本的宏基因组也可以使用相同的方法来挖掘AMP,或者使用类似的方法从“微生物暗物质”中挖掘隐藏肽信息。在人工环境中,由于微生物群落与人类密切相关,因此更有可能挖掘针对病原体的AMP。在自然环境中也有大量的AMP,但与人工环境相比开采可能更加困难。因此,有必要使用宏基因组和蛋白质组数据集和高通量筛选对环境中的肽进行挖掘。本研究中宏基因组学与机器学习相结合的方法强调了从人工环境中挖掘新的抗菌肽的潜力,并证明了从污泥中挖掘抗菌肽的有效性。
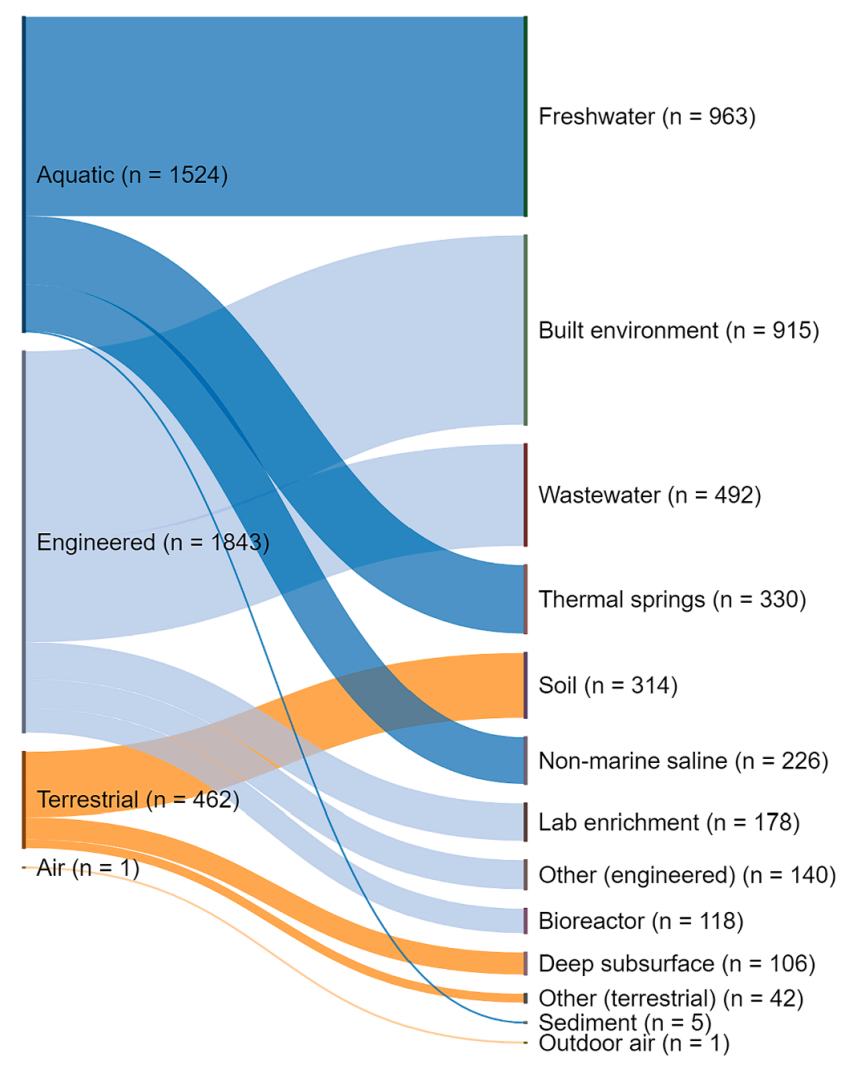
图 1 桑基图描绘了宏基因组组装的基因组的环境分布
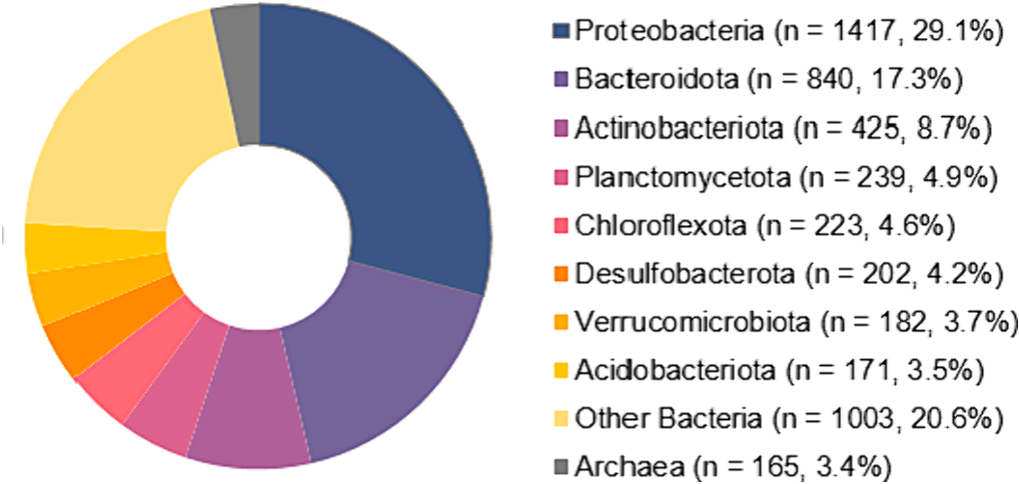
图 2 扇形图描绘了元基因组组装的基因组在门水平上的种群分布
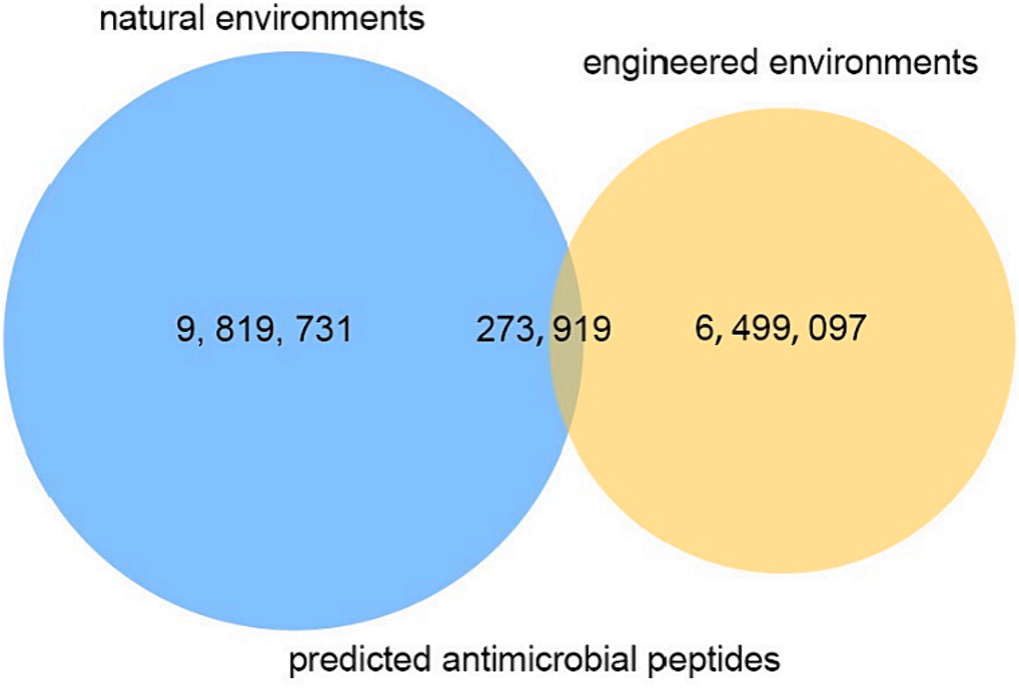
图 3 维恩图描绘了来自自然和工程环境的宏基因组组装基因组中预测的抗菌肽
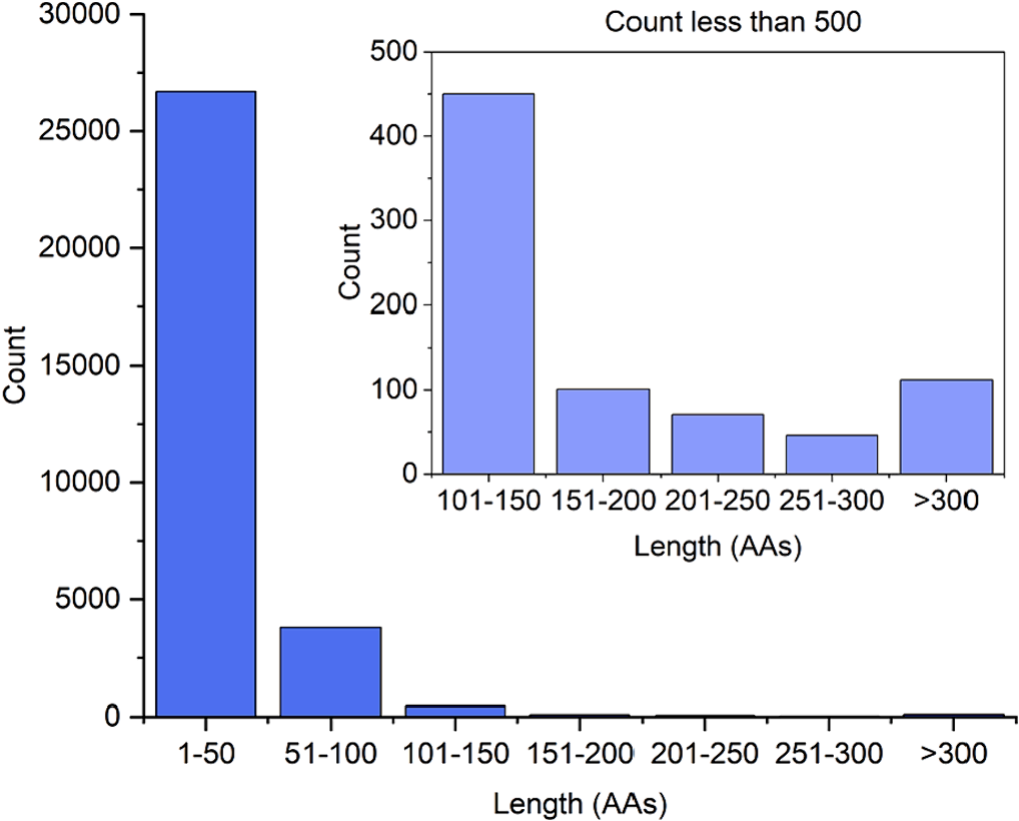
图 4 抗菌肽在CAMP、DRAMP、APD和ADAM数据库中的长度分布的条形图。
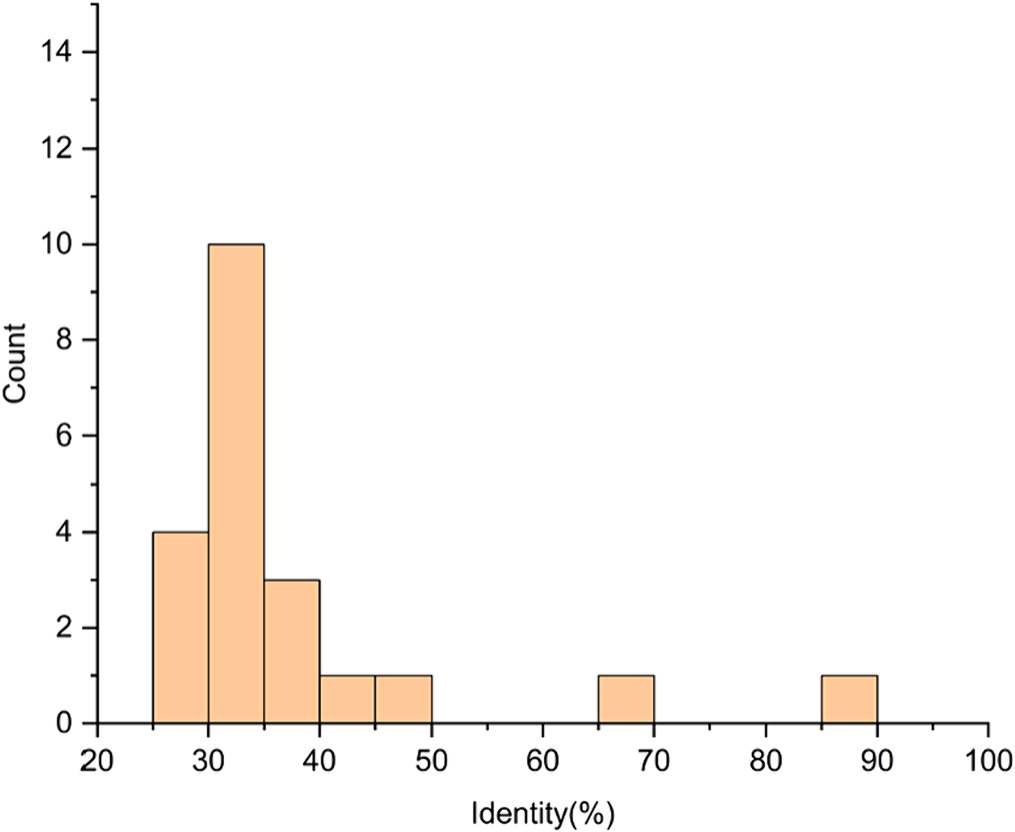
图 5 柱状图描述了研究中21个验证的抗菌肽与CAMP、DRAMP、APD和ADAM数据库的最大相似度分布
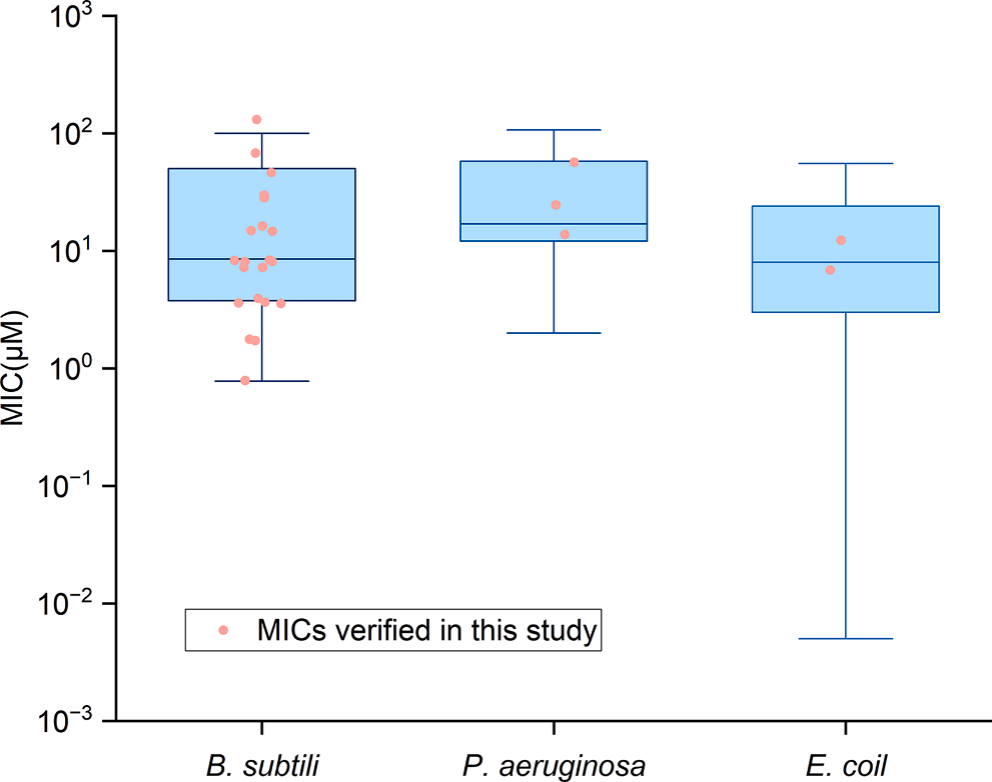
图 6 本实验通过参考CAMP、DRAMP和APD数据库中抗菌肽的MIC值筛选出抗菌肽的MIC范围
四、参考文献
Xu J, Xu X, Jiang Y, Fu Y, Shen C. Waste to resource: Mining antimicrobial peptides in sludge from metagenomes using machine learning. Environ Int. 2024 Apr;186:108574. doi: 10.1016/j.envint.2024.108574. Epub 2024 Mar 11. PMID: 38507933.
密码子云平台-小工具
登录密码子生信云平台(https://cloud.mimazi.net),免费畅享云流程和小工具一键化操作,助您快速完成数据分析。平台提供宏基因组抗菌肽预测和丰富的可视化工具,包括桑基图、环形饼图、韦恩图、柱状图、箱线图等,您也可以轻松创建类似文献中的精美图表。





https://wap.sciencenet.cn/blog-3447233-1495955.html
上一篇:Kaptive脂多糖血清型分类系统更新
下一篇:【文献速递】确定性和随机过程在构建陆地生态系统土壤细菌生态型中的不同作用