 精选
精选
一种基因编辑方法可对小鼠肠道内的细菌进行改造
数十年来,微生物学家一直面临一个难题:若要真正了解一种肠道细菌,就必须在其天然生态系统中对其进行研究;而若要对其基因进行改造,则必须将其从肠道中分离出来。将经过基因改造的细菌重新引入其自然环境难度极大——这种方法仅限于无菌动物模型,或需使用抗生素处理动物以促进工程菌定植,而这两种方式都会破坏肠道内原有的微生物群。此外,肠道微生物中仅有一小部分易于在培养环境中生长,且能够进行基因改造。若能在细菌的天然环境中直接对其进行编辑,便可规避这些限制,这一方向已成为极具前景的研究领域(参考文献1-3)。在本期杂志第696页,格尔辛格等人(参考文献4)介绍了“宏基因组编辑技术”(MetaEdit),该技术可在小鼠肠道环境中,将大片段DNA序列(即“DNA载体”)定向插入细菌基因组(见图)。
近年来,一些原位微生物组编辑方法通过温和噬菌体向目标细菌递送新DNA——温和噬菌体是一类感染细菌的病毒,感染后不会立即杀死宿主细菌(参考文献3、5)。另一些方法则基于噬菌粒递送DNA:噬菌粒是一种可被包装进噬菌体蛋白质外壳(衣壳)的质粒(参考文献2、6)。尽管这些方法可作用于近100%的目标细菌群体(参考文献2),但它们需要依赖能特异性识别目标细菌表面受体的噬菌体载体,才能实现DNA载体的递送。与之不同的是,MetaEdit技术利用细菌接合作用(细菌间相互交换遗传物质的过程)向小鼠肠道内的细菌递送DNA(参考文献1)。MetaEdit所使用的RP4接合机制可作用于多种革兰氏阴性菌和革兰氏阳性菌,且不易受细菌表面受体进化的影响(细菌表面受体进化可能导致噬菌体无法感染)。不过,该技术在肠道内直接编辑微生物基因组的递送效率,通常远低于其他方法。
由于复制型质粒可能会随时间推移而丢失(尤其当质粒无法为宿主细菌带来优势时),格尔辛格等人采用CRISPR相关转座酶(CAST)系统,将DNA载体整合到细菌基因组中,以确保其稳定且高效表达。由于接合作用的非特异性可能导致脱靶基因组编辑,研究人员利用宏基因组数据,通过计算设计出高特异性向导RNA(“megRNA”),确保DNA载体仅在目标细菌的特定基因组位置进行整合。此外,他们还筛选了一个质粒载体库,筛选出含有特定调控元件组合的载体——这些调控元件可确保目标DNA在拟杆菌科(Bacteroidaceae)细菌中高效表达,而拟杆菌科细菌在哺乳动物肠道中含量丰富。借助这一系统,格尔辛格等人构建了供体细菌,通过口服方式给小鼠给药,并成功将遗传载体转移到小鼠复杂肠道微生物群中的一种天然拟杆菌(Bacteroides)中。值得注意的是,研究人员还实现了对分节丝状菌(segmented filamentous bacteria)的基因改造——这类细菌参与免疫调节,但由于其会附着在肠道上皮细胞上,极难在培养环境中生长(参考文献7)。
利用MetaEdit工具对小鼠肠道细菌进行基因编辑
宏基因组编辑技术(MetaEdit)可在小鼠肠道内直接对细菌进行基因改造,包括此前无法进行基因编辑的不可培养细菌。通过调控特定营养物质的可利用性,可调节经基因改造细菌的丰度。
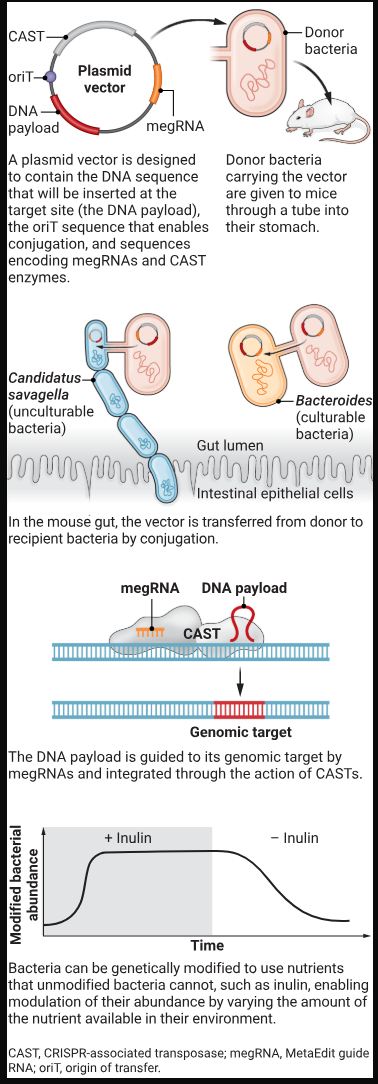
图示:K.霍洛斯基(K. HOLOSKI)/《科学》(SCIENCE)
即便DNA能完美递送、编辑具有特异性且基因组编辑结果稳定,若工程菌很快被野生型同类细菌竞争淘汰,这种编辑也将失去意义——而这正是工程菌的常见结局。格尔辛格等人观察到,经编辑的细菌通常在5-10天后从小鼠肠道中消失。为避免这一情况,研究人员向一种天然的多形拟杆菌(Bacteroides thetaiotaomicron)菌株中导入了一组基因,使该细菌能够代谢菊粉(一种未经编辑的同类细菌无法利用的碳源)。通过这种方式,工程菌可占据独特的代谢生态位,从而获得竞争优势。当给小鼠喂食富含菊粉的饲料时,工程菌大量繁殖,占多形拟杆菌总群体的比例达到约30%。这一策略与早期的一项研究思路一致:该研究显示,经改造后可利用紫菜聚糖(一种稀有的海藻多糖)的普通福赛斯坦纳菌(Phocaeicola vulgatus)菌株,在有紫菜聚糖存在的情况下,能够在新的小鼠肠道环境中定植(参考文献8)。然而,一项临床试验发现,经改造的普通福赛斯坦纳菌与其他肠道细菌之间的基因交换和重组事件,可能导致期望的治疗效果与利用补充多糖的能力脱节——这为未来的治疗方法提供了警示(参考文献9)。
展望未来,MetaEdit技术可用于在天然环境中系统探究细菌基因的功能:通过向导RNA库或载体库,开展高通量的功能缺失或功能获得研究。该技术还可能应用于治疗领域,例如:灭活细菌中不需要的基因(如毒力因子),或赋予肠道固有细菌在肠道内产生药物的能力。在一项相关研究中(参考文献10),研究人员在大肠杆菌中使用CAST系统,破坏了志贺毒素基因,同时插入了一个可表达纳米抗体(一种小型抗体)的基因——该纳米抗体可抑制未经改造的致病性大肠杆菌与肠道上皮细胞的结合。
目前,使用MetaEdit技术仍需克服一些障碍,例如:接合作用的效率、基因编辑对细菌宿主适应性的影响,以及设计目标物种特异性载体的需求。尽管如此,格尔辛格等人的研究为在微生物的天然生态位中直接对其基因组进行系统编辑,提供了一条极具前景的路径。
转载本文请联系原作者获取授权,同时请注明本文来自孙学军科学网博客。
链接地址:https://wap.sciencenet.cn/blog-41174-1510157.html?mobile=1
收藏

