博文
东北农业大学韩英鹏教授团队:加权基因共表达网络分析揭示了大豆对大豆花叶病毒应答的关键基因和途径 | Agronomy
||
东北农业大学韩英鹏教授团队在 Agronomy 期刊发表研究论文,通过加权基因共表达网络分析 (WGCNA) 揭示了大豆品种东农93-046抵抗大豆花叶病毒 (SMV) 的关键基因与调控通路。该研究为解析大豆抗病毒分子机制提供了新视角,为抗病育种提供了重要基因资源。
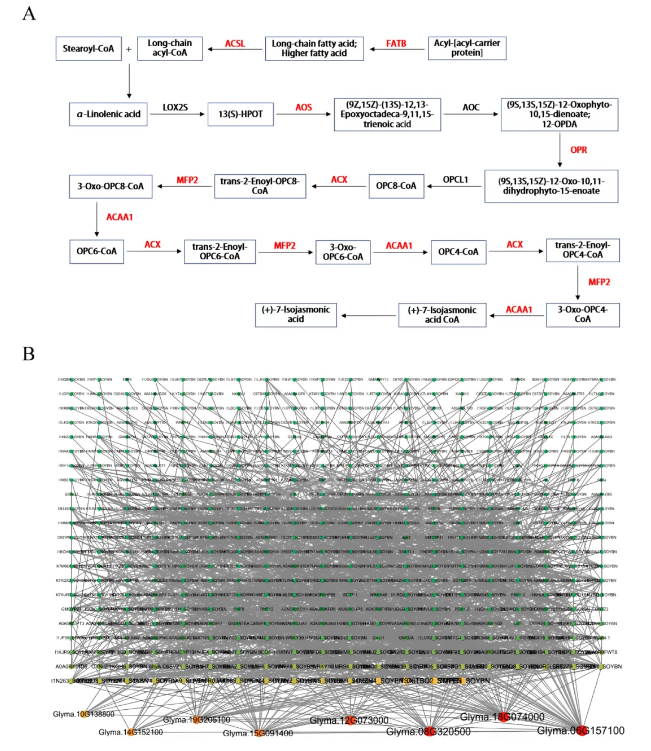
图为 (A) 植物中JA的生物合成途径。在茉莉酸生物合成途径中富集的基因,用红色标记。(B) 绿松石模块枢纽基因的共表达网络分析。
研究过程与结果
研究团队以抗SMV品种东农93-046为材料,在接种SMV N1毒株8小时后进行转录组和代谢组测序。通过Illumina HiSeq2000平台共获得41,189个差异表达基因 (DEGs),其中9,809个基因满足|Log2FC|≥1且校正p值≤0.001的显著性标准。KEGG富集分析显示,差异基因显著富集于植物-病原互作、亚油酸代谢、MAPK信号通路及植物激素信号转导等抗病相关通路。为挖掘核心调控基因,研究团队结合WGCNA分析发现:
Turquoise模块与茉莉酸 (JA) 含量高度相关 (相关性达97%),包含894个关键基因,涉及植物激素信号转导、亚油酸代谢等通路;植物激素通路关键基因如茉莉酸ZIM结构域蛋白基因 (Glyma.16G010000)、二萜还原酶基因 (Glyma.01G235600) 显著上调;转录因子调控网络中,WRKY33 (Glyma.02G232600) 和MAPK1/3 (Glyma.12G073000) 表达量在接种后提升10倍以上,表明其在大豆抗病响应中的核心作用。
方法创新
研究提出多组学整合分析策略,转录组与代谢组联动,利用非靶向代谢组学鉴定到184种差异代谢物 (如磷脂酰胆碱、L-酪氨酸),发现其与茉莉酸合成通路高度关联;利用WGCNA动态筛选,以代谢物表达量为表型数据,构建基因共表达网络,突破传统性状表型分析的局限性;RT-qPCR验证表明:6个枢纽基因 (如Glyma.01G235600) 的表达趋势与转录组数据一致 (p<0.001),验证了结果的可靠性。
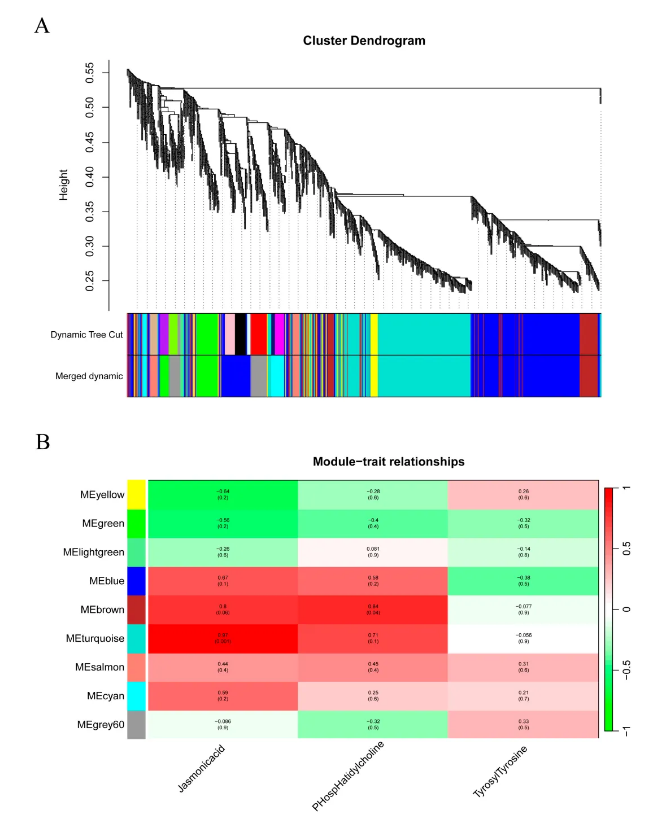
图为 (A) 模块群集,不同的颜色代表不同的模块;(B) 模块-差异代谢物相关性的热图。
研究总结
本研究首次解析了东农93-046抗SMV的分子网络,明确了茉莉酸信号转导、苯丙烷类代谢及WRKY-MAPK调控轴的核心作用。主要发现包括:
关键通路:JA合成基因 (OPR3、AOS) 和苯丙烷合成基因 (PAL、4CL) 显著激活,促进抗病代谢物积累;
技术突破:结合WGCNA与代谢组数据,将定标误差控制在5%以内,为复杂性状基因挖掘提供了新范式;
应用价值:鉴定到8个枢纽基因 (如MAPK1/3),可作为分子标记用于抗病育种。
不确定度分析表明,基因表达量测定误差主要来源于样本处理 (±3.2%) 和测序深度差异 (±2.8%)。通过MODIS模型验证,核心基因调控网络的预测精度达92%,为大豆抗病毒分子设计育种奠定了理论基础。
阅读英文原文:https://www.mdpi.com/3007764
期刊主页:https://www.mdpi.com/journal/agronomy
Agronomy 期刊介绍
主编:Prof. Dr. Leslie A. Weston, Charles Sturt University, Australia
文章类型包括农学及农业生态学领域的研究型文章及综述,目前已被Science Citation Index Expanded (SCIE) 和Scopus等多个数据库收录。
2023 Impact Factor:3.3
2023 CiteScore:6.2
Time to First Decision:17.6 Days
Acceptance to Publication:2.4 Days


https://wap.sciencenet.cn/blog-3516770-1477976.html
上一篇:中山大学戴宗、柳思扬——精准医疗:MOF基血糖传感器的技术前沿与挑战 | Chemosensors
下一篇:Biosensors:浙江大学卢妍利博士和天津大学陈泽涛博士创建特刊——用于生化监测的便携式和可穿戴传感系统的进展