博文
NCBI蛋白质BLAST检索数据库最新变更
|
1、NCBI蛋白数据库变更
自2025年8月起,ClusteredNR将取代传统的标准NR数据库,成为蛋白质BLAST搜索的默认数据库。这一变更旨在提升搜索效率和结果质量。当然,传统的NR数据库仍可通过手动选择继续使用。
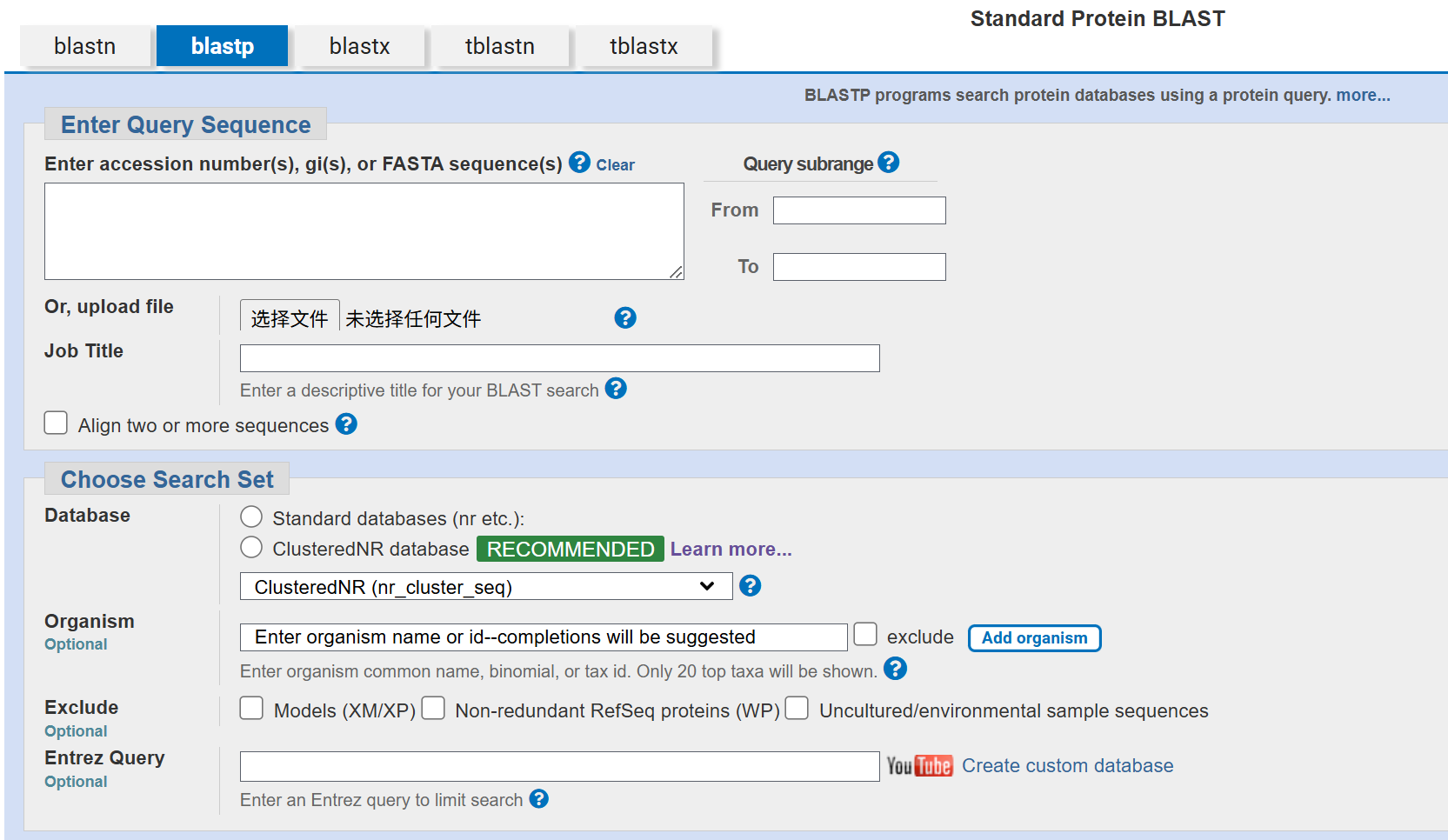
2、ClusteredNR的优势
更快的搜索速度:通过聚类减少数据量,加速分析流程。
减少冗余结果:每个聚类仅保留代表性序列(基于标题和功能筛选),避免重复相似序列的干扰。
更广的分类覆盖范围:结果中能体现更多物种的多样性信息(某些物种代表性过高,在蛋白质搜索中占据主导地位,例如示例中人类朊蛋白的搜索结果覆盖了132条序列的聚类)。
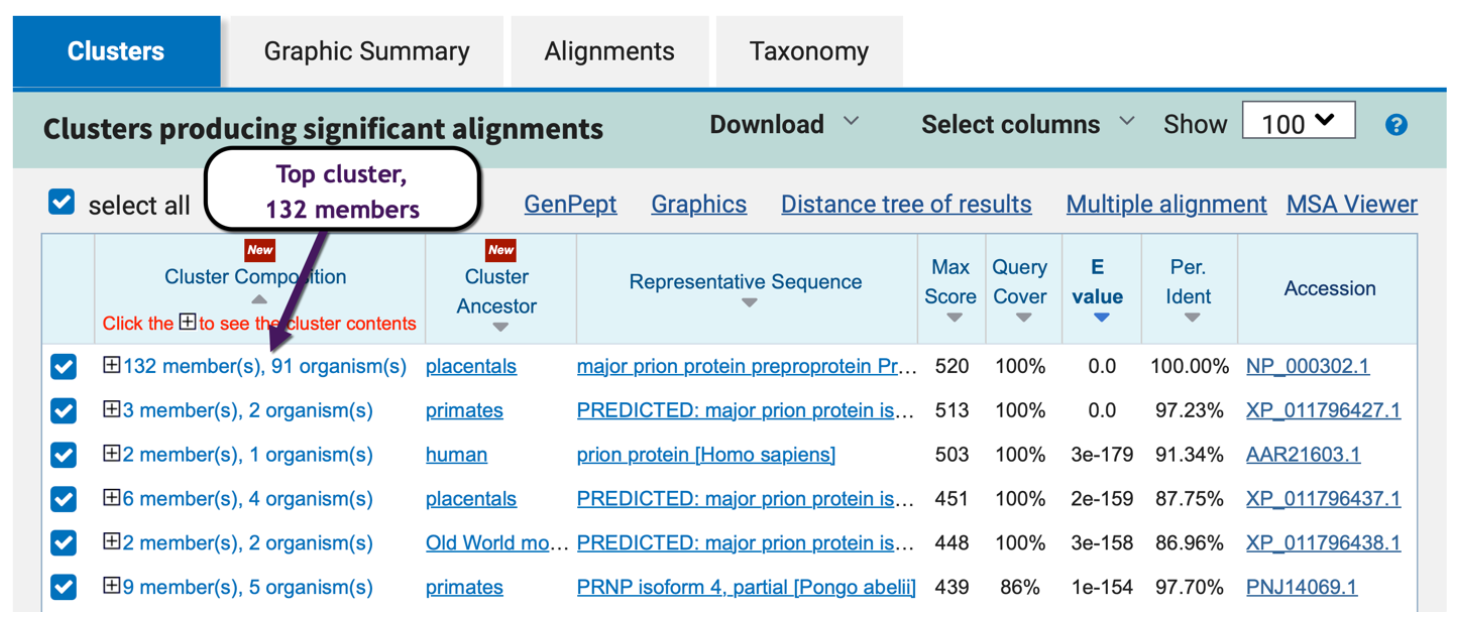
3、ClusteredNR的来源
ClusteredNR的本质是对标准NR数据库进行去冗余聚类,通过90%相似性+90%长度的阈值,将高度相似的蛋白质归为同一簇,并选取代表性序列优化 BLAST 搜索体验。
聚类方法:使用MMseqs2对NR数据库中的蛋白质序列进行聚类,保证序列相似性≥90%(即同一簇内的蛋白质序列彼此间至少90%相同)。长度≥90%的最长成员(确保短片段或截断序列不会影响聚类质量)。
代表性序列的选择:每个簇选取一个注释良好的代表性蛋白质,在BLAST结果中,仅显示该代表性序列的标题,以减少冗余信息。
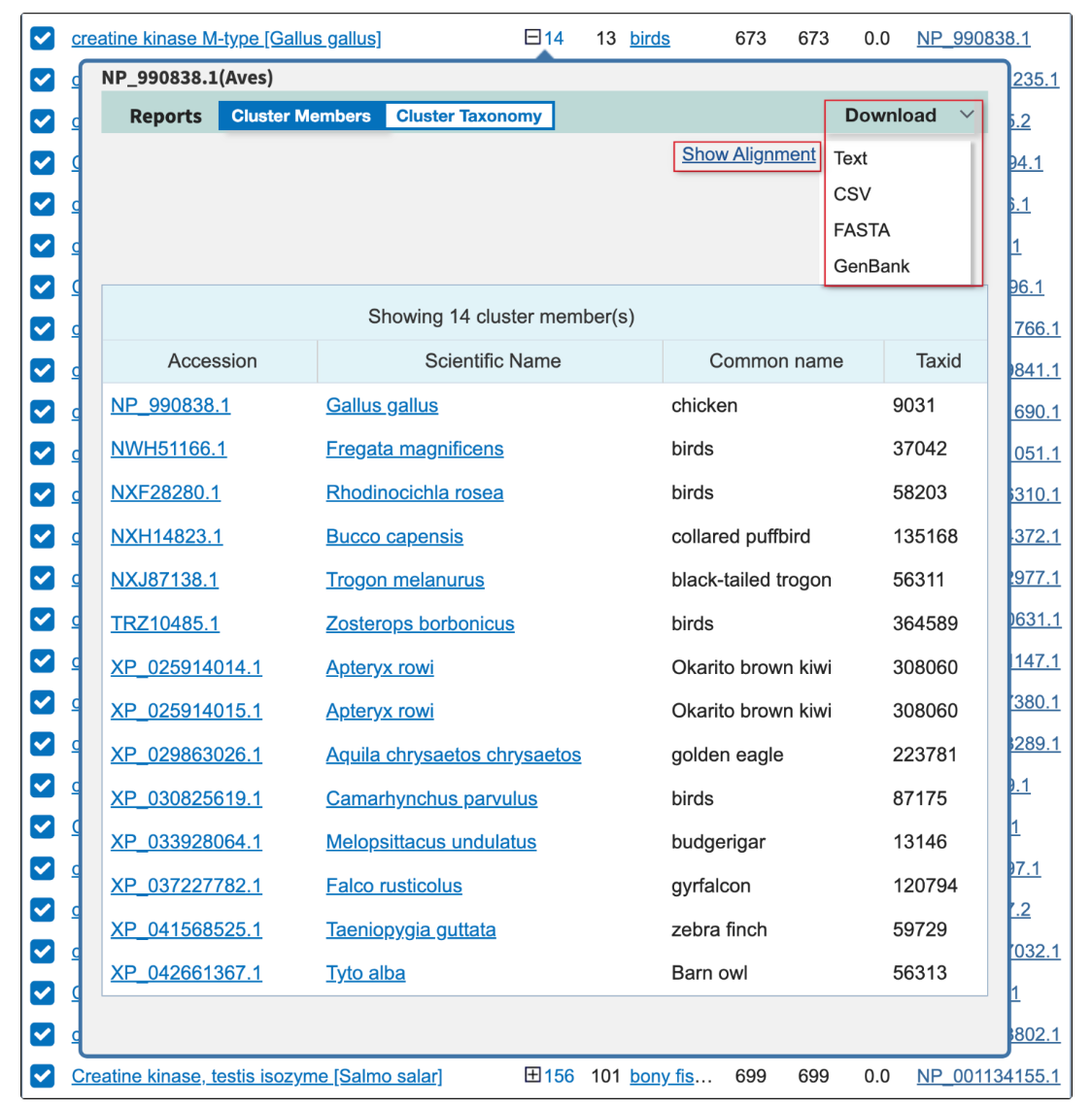
用户可以展开Cluster,查看或下载所有成员蛋白质的序列报告。
参考文件(NCBI):
1、Faster, Better Results for Protein BLAST Searches
2、New ClusteredNR database: faster searches and more informative BLAST results
我们将持续分享微生物组学研究和生信分析相关的专业技能资料。推荐课程请搜索“密码子学院”。课程问题或个性化分析需求,请联系小唯(微信号:winnerbio01)。
https://wap.sciencenet.cn/blog-3447233-1489949.html
上一篇:BLAST软件自定义输出表格样式
下一篇:新型ST6417 CR-hvKP的发现