博文
大肠埃希菌的分型和克隆传播
|
第一部分:大肠埃希菌的流行病学分型
1.1 系统发育分型(Phylogroup)
大肠埃希菌(Escherichia coli)通常使用phylogroup(称为亚特异性群体/亚种分型)来反映其种内的集群特征,它是根据多位点酶电泳检测到的酶编码基因的等位基因变异确定的。如图1所示,目前,E. coli可以分为9个主要phylogroups:A、B1、B2、C、D、E、F、G、H和5个隐进化支Escherichia clades I-V[1]。Escherichia fergusonii、Escherichia albertii由于和E. coli在系统发育上非常接近,可能会影响E. coli的判断。
2018年Clermon等人开发了基于chuA、yjaA、arpA以及DNA片段TspE4.C2的一种四重PCR模拟鉴定程序ClermonTyping(http://clermontyping.iame-research.center/),此程序允许使用给定菌株的fasta序列,将phylogroup分配给E. albertii、E. fergusonii、Escherichia clades I-V、以及E. coli sensu stricto (phylogroup A-F)[2]。
图1 Phylogenetic history of 72 Escherichia strains(https://doi.org/10.1038/s41579-020-0416-x)
1.2 多位点序列分型(MLST)
大肠埃希菌的ST方案包括STWU(Achtman, 7 loci)和STIP(Pasteur, 8 loci),两者采用的管家基因完全不同。在pubMLST数据库中,STWU有明确的STc(clonal complex, Cplx),它可以用于Clade/Lineage的划分,如图2和图3[3]所示。
图2 pubMLST database(https://pubmlst.org/bigsdb?db=pubmlst_escherichia_seqdef)
图3 Inferred phylogenetic relationships of 112 E. coli phylogroup G strains(doi:10.1111/1462-2920.14713)
1.3 血清型(Serotype)与致病型(Pathotype)
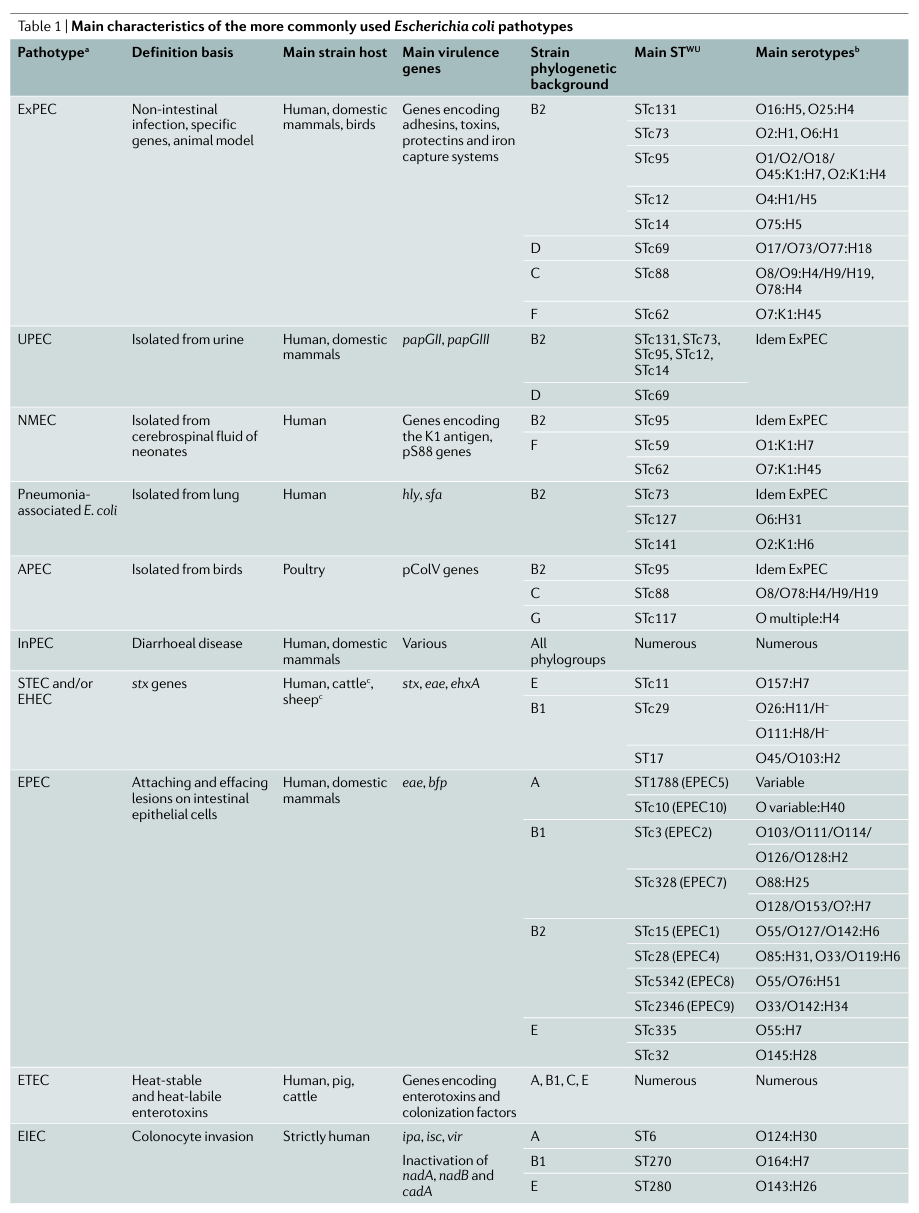
O抗原、H抗原、K抗原血清型(Serotype)也是大肠埃希菌非常重要的流行特征,它们与致病型(Pathotype)密切相关[1],包括肠致病性大肠杆菌(EPEC)、肠产毒性大肠杆菌(ETEC)、肠侵袭性大肠杆菌(EIEC)、肠出血性大肠杆菌(EHEC)、肠黏附性大肠杆菌(EAEC)和弥散粘附性大肠杆菌(DAEC)等,如O157: H7就是EHEC的代表。另外,fimH分型与大肠埃希菌的粘附和移动有关。这些内容常用于系统发育树注释(见图3、图4)。
图4 Example phylogenetic history of Escherichia coli strains of three main sequence types according to the Warwick University (WU) scheme(https://doi.org/10.1038/s41579-020-0416-x)
第二部分:细菌克隆的通用SNP界定
Salipante, S. J. 等人(2015)探索了细菌全基因组测序 (WGS) 作为菌株分子分型的替代方法[4]。文中写道:真正构成“细菌克隆”或“克隆爆发”的问题出奇地复杂,菌株可能在个体患者的感染过程中发生变化,因此,遗传不同但关系非常密切的分离株可能反映了共同的起源。目前,还没有确定的阈值来定义识别分离株属于爆发的全基因组多态性的数量。各种研究估计,大多数细菌的基因组以每年大约 2 到 10 个的速度积累SNP,具体取决于生物体。因此,根据为 PFGE 开发的现有分类框架,作者建议通过以下三项定义对基于 WGS 的菌株比较做一般性的解释: ① SNP≤3,认为菌株“基因组上无法区分”;② SNP≤12,认为菌株“密切相关”;③ SNP≥13,认为“不相关”。另外,分子流行病学调查需要将该分类与额外的信息和背景相结合,特别是分离株收集的时空信息,以确定潜在传播事件的合理性。
第三部分:大肠埃希菌克隆的SNP阈值
1、大肠埃希菌克隆与ST的关系
Denamur, E.等人(2021)在综述[1]中写道,克隆由来自共同祖先、无法区分或高度相近的分离株组成。目前,MLST是定义大肠埃希菌菌株的常用方法,分离株的ST通常被视为代表某一克隆。然而对于EHEC,研究人员仍然坚持用血清型命名。图4(原文图2)分别表示了STWU131、STWU95和STWU117的克隆/ST,并通过STIP来表现树的逐步分化,这意味着克隆的定义取决于ST的群体结构。
2、文献中的SNP阈值依据
① 一项基于丹麦 127 个全基因组测序分离株单克隆 (ST410) 的全国流行病学研究,以≤10 个SNP确定了 5 个可能的区域性爆发。
② 一项以MLST来区分产ESBL大肠埃希菌流行病学“相关”和“无关”分离株的研究,将从同一患者获得、属于同一 MLST 并在 30 天内培养的分离株定义为流行病学相关。
③ 一项使用cgMLST 调查德国医院血液科和肿瘤科患者之间大肠埃希菌传播的研究,将“密切相关”的分离株定义为最多具有 10 个等位基因差异。
3、大肠埃希菌克隆的阈值参考
由于没有统一的参考标准,Ludden, C.等人(2021, Lancet Microbe)[5]通过比较来自同一宿主分离株的SNP距离并将其与流行病学数据相结合,建立了一个基于基因组的SNP阈值,来推断研究人群中的大肠埃希菌传播。研究发现来自同一患者、相同ST的分离株,其核心基因组成对比较最大多样性为 17 个 SNP,且这些样本具有很强的流行病学联系(同一隔间、房间或病房,同一时间或在 7 天内)。
图5 Histogram of maximum pairwise SNP distance (n=92) -同一患者鉴定出至少两个相同ST分离株时的SNP差异(doi: 10.1016/S2666-5247(21)00117-8)
基于17个SNP的阈值定义,作者分析了患者间的传播网络,如下图所示,同一时间或在 7 天内进入同一隔间、房间或病房的被归类为强流行病学联系;同一病房的入院间隔超过7天或进入研究医院不同病房的被归类为弱流行病学联系;两者都不是,则没有流行病学联系。
图6 Transmission network of E. coli STs shared by patients based on 17 SNP cut-off(补充材料,doi: 10.1016/S2666-5247(21)00117-8)
参考文献
[1] Denamur E, Clermont O, Bonacorsi S, et al. The population genetics of pathogenic Escherichia coli [J]. Nature Reviews Microbiology, 2020, 19(1): 37-54.
[2] Beghain J, Bridier-Nahmias A, Le Nagard H, et al. ClermonTyping: an easy-to-use and accurate in silico method for Escherichia genus strain phylotyping [J]. Microb Genom, 2018, 4(7).
[3] Clermont O, Dixit O V A, Vangchhia B, et al. Characterization and rapid identification of phylogroup G in Escherichia coli, a lineage with high virulence and antibiotic resistance potential [J]. Environmental Microbiology, 2019, 21(8): 3107-17.
[4] Salipante S J, Sengupta D J, Cummings L A, et al. Application of Whole-Genome Sequencing for Bacterial Strain Typing in Molecular Epidemiology [J]. Journal of Clinical Microbiology, 2015, 53(4): 1072-9.
[5] Ludden C, Coll F, Gouliouris T, et al. Defining nosocomial transmission of Escherichia coli and antimicrobial resistance genes: a genomic surveillance study [J]. The Lancet Microbe, 2021, 2(9): e472-e80.
零基础做生信
唯那生物推出的生信云平台服务(https://cloud.mimazi.net)提供了一些与SNP分析、分子分型相关的小工具。用户无需安装软件或配置环境,仅需注册一个账号,找到以下免费小工具,提交对应文件,即可便捷地完成SNP和分型相关的分析,快速获取并下载对应的结果。大肠埃希菌亚群和血清型鉴定可以参考往期文章《实用组学小工具——大肠埃希菌亚群分类和鉴定》和《实用组学小工具——大肠埃希菌血清型分析SerotypeFinder和ECTyper》。SNP分析小工具包括以下内容:
1、基于多序列比对结果分析SNP/Indel
2、基于多序列比对文件生成SNP距离矩阵(snp-dists)
3、基于FastA比对文件提取SNP位点(SNP_site)
4、微生物核心基因组比对和 SNP(单核苷酸多态性)检测(Parsnp软件)
https://wap.sciencenet.cn/blog-3447233-1502058.html
上一篇:实用组学小工具——耐药基因注释(CARD-RGI)
下一篇:细菌基因组注释 | 第二站:高精度注释工具Bakta