博文
PSI-BLAST使用
|
PSI-BLAST 即Position-Specific Iterated (PSI)-BLAST,是一种更加高灵敏的Blastp程序,对于发现远亲物种的相似蛋白或某个蛋白家族的新成员非常有效,适用于使用标准的Blastp比对失败时,或比对的结果仅仅是一些假基因或推测的基因序列时("hypothetical protein" or "similar to...")。
PSI-BLAST使用方法
1. 访问NCBI-BLAST 在线比对网址(https://blast.ncbi.nlm.nih.gov/Blast.cgi),选择 Protein BLAST
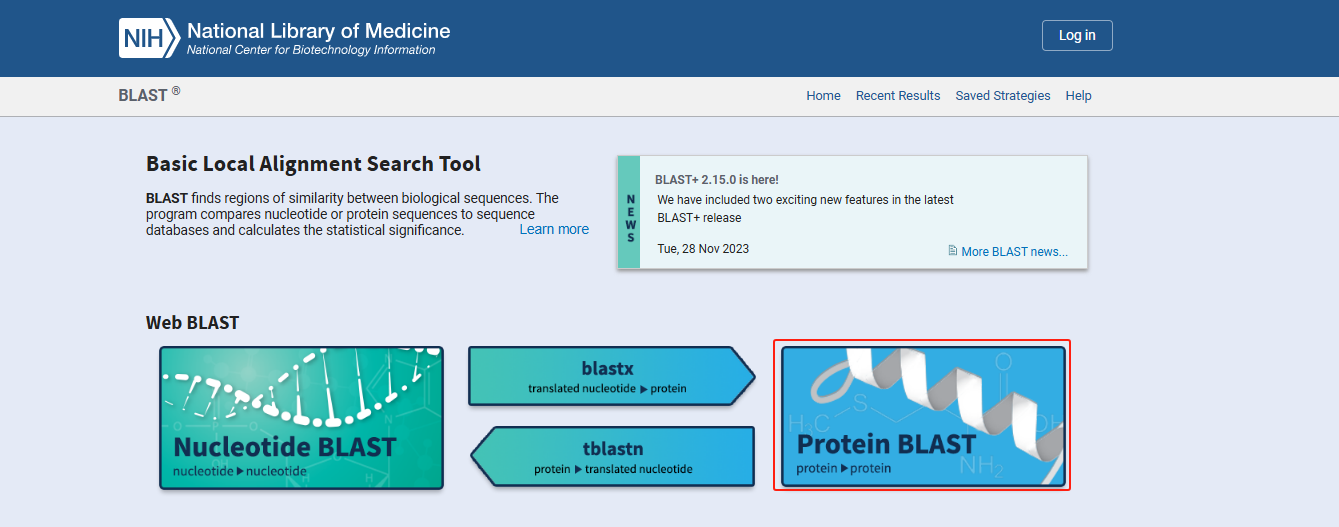
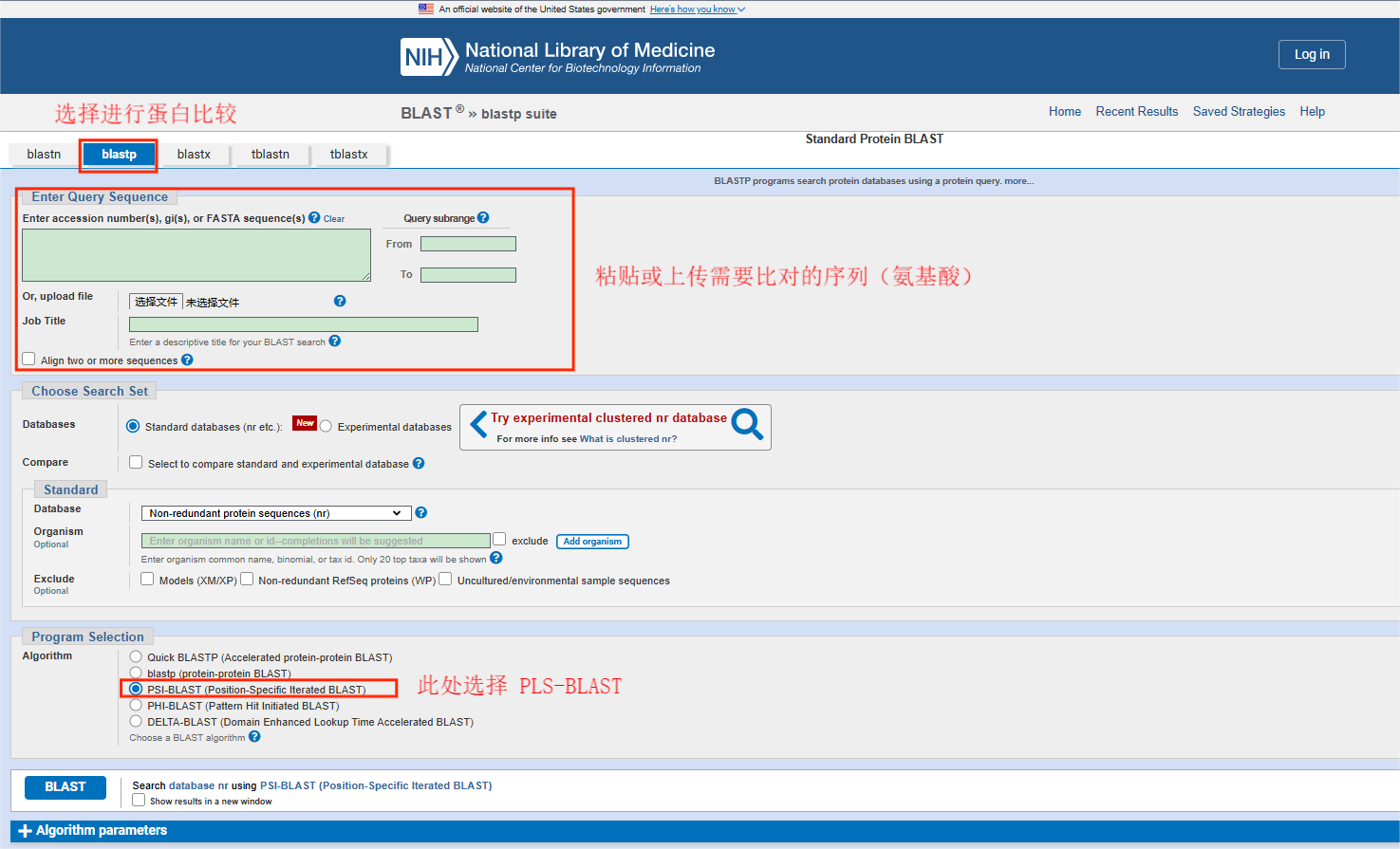
2. 粘贴或上传需要比对的序列之后,Program Selection 选择 PSI-BLAST,运行blast。
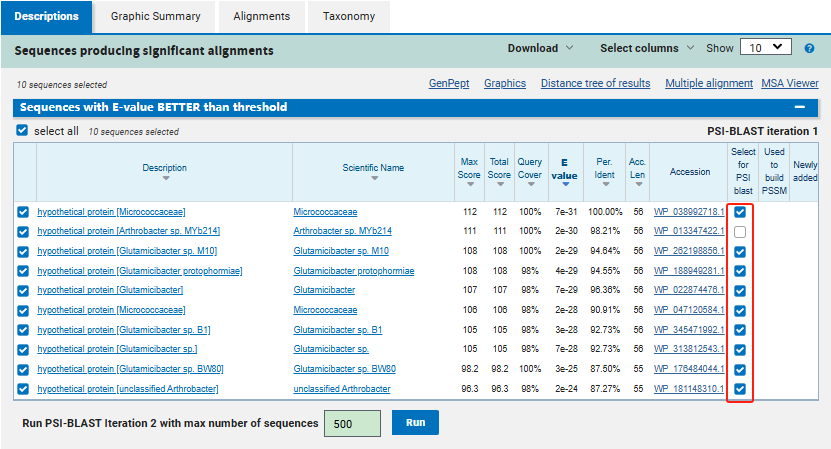
3. 勾选所需的序列,点击运行,生成PSSM打分矩阵。
4. 运行后会根据打分矩阵将第一轮中遗漏的部分(黄色)抓取回来。

5. 反复多次比对,直到新的结果不再出现或者程序明确指出不会再有新的结果出现时,可以停止比对循环。
我们将持续分享微生物组学研究和生信分析相关的专业技能资料。推荐课程请搜索“密码子学院”。课程问题或个性化分析需求,请联系小唯(微信号:winnerbio01)。
https://wap.sciencenet.cn/blog-3447233-1494197.html
上一篇:文献速递 | 乳酸通过PTS-CRP促进荚膜多糖合成,加剧肺炎克雷伯菌肝脓肿侵袭性
下一篇:【文献速递】人类呼吸道微生物组和抗生素耐药基因在健康人群中的传播
扫一扫,分享此博文