 精选
精选
地球生物祖先靠氢气为生!
阿斯加德古菌中海姆达尔古菌外真核生物的深层起源
本研究是华东师范大学团队的工作。
Zhang J, Feng X, Li M, Liu Y, Liu M, Hou LJ, Dong HP. Deep origin of eukaryotes outside Heimdallarchaeia within Asgardarchaeota. Nature. 2025 May 7.
摘要
对阿斯加德古菌的形态学、生理学和基因组学的研究,为了解真核生物的进化历史提供了有价值的见解[1,2,3]。先前的一项研究表明,真核生物嵌套在海姆达尔古菌中[4],但其在阿斯加德古菌中的准确系统发育位置仍然存在争议[4,5]。这一争议使得理解早期真核生物祖先的代谢特征和时间尺度变得复杂。在这里,我们生成了223个之前未被记录的阿斯加德古菌宏基因组组装的近乎完整的基因组。我们在属水平或更高水平上鉴定出16个新的谱系,这极大地扩展了已知的阿斯加德古菌的系统发育多样性。通过对这个扩展的基因组数据集进行复杂的系统发育基因组分析(涉及多个标记集),我们推断真核生物在所有采样的海姆达尔古菌多样化之前就已经进化,而非与海姆达尔古菌中的霍达古菌目分支在一起。真核生物位置的这种差异可能是由于之前未被充分认识的尼奥德古菌目基因组的嵌合性质,我们发现其由阿斯加德古菌和TACK古菌(阿斯加德古菌的姐妹门类)的序列组成。通过祖先重建和分子定年,我们推断最后一个阿斯加德古菌和真核生物的共同祖先出现在大氧化事件之前,并且可能是一种依赖氢气的厌氧产乙酸菌。我们的发现支持真核生物起源的氢气假说,该假说认为真核生物起源于消耗氢气的古菌宿主与产生氢气的原线粒体的融合。
真核细胞的起源长期以来一直是个谜[6]。阿斯加德古菌比其他古菌含有更多编码真核生物特征蛋白(ESP)的基因,被认为是真核生物最接近的亲属[4,5,7,8,9]。第一个被分离和培养的阿斯加德古菌(“Candidatus Prometheoarchaeum (Ca. P.) syntrophicum” MK-D1,属于洛基古菌门)显示,这种古菌具有长的分支突起,并且能够通过互营方式降解氨基酸[1]。这些形态和代谢特征为真核生物起源的过程提供了新的见解。例如,人们认为细胞突起可能介导细胞间的相互作用,导致吞噬其伙伴细胞并逐渐形成线粒体。随后,第二个阿斯加德古菌(“Candidatus Lokiarchaeum (Ca. L.) ossiferum”)最近被富集,冷冻电镜断层扫描和免疫染色显示存在类似肌动蛋白的细胞骨架丝以及Lokiactin的表达[2]。据推断,Lokiactin可能作为阿斯加德古菌细胞的支架,并在维持细胞形态、细胞分裂和分子运输中发挥作用,类似于真核细胞中的肌动蛋白丝[10,11]。这些结果和假设暗示真核生物的核谱系从古菌中进化而来。
尽管在阿斯加德古菌的纯培养或富集物中已经鉴定出一些类似真核生物的特征,但由于菌株数量有限,推断早期真核生物祖先的确切系统发育位置、时间尺度和代谢特征仍然具有挑战性。相比之下,基于大量古菌基因组数据的系统发育基因组分析可以弥补这些不足[12]。通过使用超树法和合并法,对古菌和真核生物中超过3000个基因家族进行分析,海姆达尔古菌门被认为是真核生物最接近的亲属[13]。然而,该研究仅使用了六个阿斯加德古菌基因组,未能代表目前扩展的阿斯加德古菌的多样性。最近,两项研究基于对阿斯加德古菌的扩展基因组采样,得出了关于真核生物与阿斯加德古菌之间关系的不一致结果。一项研究表明,真核生物更有可能从阿斯加德古菌中分支出来,作为海姆达尔古菌门 - 悟空古菌分支的姐妹群,或者是古菌中更深层次的分支[5],而另一项研究则提出真核生物在海姆达尔古菌门中作为霍达古菌目的姐妹谱系分支[4]。前者仅使用了29个串联标记来推断系统发育,而后者使用非核糖体蛋白作为标记,并广泛排除比对中的位点。真核生物与古菌之间的确切关系仍有待进一步阐明。
在这里,我们展示了223个新的阿斯加德古菌基因组,包括鉴定出的16个额外的目、科或属水平的谱系,这些谱系是从中国沿海湿地的14个位点生成的宏基因组样本中获得的,这显著扩展了阿斯加德古菌的多样性。通过利用复杂的系统发育基因组方法(包括对序列比对进行重新编码、在最大似然法(ML)和贝叶斯推断中使用复杂的位点异质进化模型以及减少速率异质性)分析扩展的阿斯加德古菌基因组采样,我们有力地将真核生物置于阿斯加德古菌中,作为海姆达尔古菌的姐妹分支。通过应用祖先重建和分子定年,描绘了最后一个阿斯加德古菌和真核生物共同祖先(LAECA)的时间尺度和代谢特征,发现其与先前的研究有显著差异[1,4]。
阿斯加德古菌多样性的扩展
阿斯加德古菌基因组多样性的增加有助于解决真核生物与阿斯加德古菌之间的进化关系[4,5,7]。为此,我们从中国的盐沼和红树林湿地收集了40个沉积物样本,并进行了宏基因组测序(补充表1和补充图1a)。在对支架进行从头组装和分箱后,我们重建了11878个宏基因组组装的基因组(MAGs)(补充图1b)。在这些MAGs中,223个属于阿斯加德古菌,完整性大于70%,污染率小于10%(补充表2和补充图1c)。这些MAGs与395个公开可用的高质量基因组相结合。通过使用dRep[14]在物种水平上选择代表(平均核苷酸同一性(ANI)至少为95%),我们最终获得了一组411个阿斯加德古菌基因组,其中136个来自本研究(补充表2)。其中,两个来自培养的阿斯加德古菌(Ca. P. syntrophicum和Ca. L. ossiferum),其余基因组是从环境样本中回收的MAGs(图1)。这些基因组的平均完整性为85.3%,平均污染率为3.6%。

图1:在GTDB r207中对53个串联古菌标记(GTDB.ar53)的系统发育基因组分析。
使用IQ-TREE在LG + C60 + F + G + PMSF模型下推断的最大似然树,基于一组579个古菌分类群(411个阿斯加德古菌、51个DPANN古菌、47个广古菌和70个TACK古菌代表)。树中仅显示阿斯加德古菌谱系。两个培养的阿斯加德古菌菌株用红星突出显示。本研究中新鉴定的基因组在最外环中用彩色条表示。自展支持值至少为95%用黑点表示。比例尺表示每个位点的平均预期替换数。
为了解决这些进化枝的系统发育位置问题,我们基于基因组分类数据库(GTDB)中一组53个古菌特异性标记蛋白(GTDB.ar53)推断了一个最大似然树,所涉及的古菌基因组在系统发育上具有多样性(411个阿斯加德古菌、51个DPANN(Diapherotrites、Parvarchaeota、Aenigmarchaeota、Nanoarchaeota和Nanohaloarchaeota)古菌、47个广古菌和70个TACK(Thaumarchaeota、Aigarchaeota、Crenarchaeota、Korarchaeota)古菌代表,补充表3)。该树证实了十个类水平的阿斯加德古菌谱系的存在(图1)。基于相对进化距离[16],我们在奥丁古菌门和海姆达尔古菌门中鉴定出两个假定的目水平的谱系(相对进化距离小于0.53),暂时命名为杨戬古菌目和文忠古菌目。此外,在西芙古菌门、霍达古菌目、赫莫德古菌门和洛基古菌门中鉴定出四个新的科水平进化枝(由六个MAGs代表)和十个新的属水平进化枝(由18个MAGs代表)。在十个阿斯加德古菌纲中的六个纲中,我们展示了108个物种水平的新基因组(图1)。目前,最近提出的尼奥德古菌目在古菌系统发育中的位置存在激烈争议[4,17,18]。最近,在使用新的替代标记(NM57)推断的树中,它被置于海姆达尔古菌门内,与格尔德古菌目、卡里古菌科和海姆达尔古菌科形成一个进化枝[4]。我们从先前的研究中选择了12组常用的标记蛋白(补充表4)[4,5,7,8,13,19,20,21,22,23],这些蛋白在大多数古菌进化枝中是保守的(方法部分)。基于一组包括扩展的阿斯加德古菌基因组采样的579个古菌代表,在复杂的进化模型(LG + C60 + F + G + PMSF)下对这些标记集进行系统发育基因组分析。这些结果表明,除了在NM57树中,尼奥德古菌目被置于TACK超门内,并且在其他11棵树中作为Korarchaeota的姐妹谱系分支,且具有较高的支持度(补充图2)。鉴于在先前的系统发育基因组分析中,尼奥德古菌目经常与真核生物分支在一起[4,18],确定不同树中尼奥德古菌目在古菌中拓扑结构冲突的潜在因素至关重要。
重新审视尼奥德古菌目的位置
Eme等人[4]认为,在RP56树中尼奥德古菌目和Korarchaeota的单系性可能是由于RP56核糖体标记蛋白序列中的高组成相似性,这与这两个谱系的嗜热生活方式有关。基于这一假设,他们提出了一个新的标记数据集,该数据集由57个古菌来源的蛋白组成,不包括核糖体蛋白,用于推断古菌的系统发育。NM57标记是从Petitjean等人[21]鉴定的一组200个古菌保守标记蛋白(NM200)中选择的,其中大多数是参与代谢和细胞过程的功能蛋白。我们观察到,尽管没有核糖体蛋白,NM200数据集的最大似然树将尼奥德古菌目置于TACK超门内,作为Korarchaeota的姐妹进化枝(补充图2k)。这表明尼奥德古菌目和Korarchaeota的聚类不一定与核糖体蛋白相关。不一致的拓扑结构可能归因于两个数据集比对中氨基酸位点的数量和性质的差异。与NM57拓扑结构相比,具有更多氨基酸位点的NM200树更有可能代表尼奥德古菌目与其他古菌之间的真实进化关系。
在系统发育基因组分析中,标记蛋白在不同古菌门或进化枝中的均匀分布对于阐明古菌进化枝之间的系统发育关系至关重要。为此,我们整合了先前研究中的12组常用标记蛋白(补充表4),并选择了67个在所有采样的古菌基因组中保守的标记(S67)(在每个古菌进化枝的至少60%的代表中鉴定到;方法部分)。S67标记集由39个核糖体蛋白和28个参与不同细胞活动的功能蛋白组成(补充表4)。在复杂的进化模型下对S67数据集进行的系统发育基因组分析证实,尼奥德古菌目是Korarchaeota的姐妹谱系,并且以高支持度(自展值至少为95)解析了阿斯加德古菌的所有节点(扩展数据图1)。
鉴于大量数据的串联可能会增强非系统发育信号,并导致具有高支持度的人为系统发育树,接下来我们通过去除突变饱和或速率偏差位点,评估数据排除对系统发育推断的影响[24,25,26,27]。我们追踪了在使用复杂位点异质进化模型从S67比对推断的树中,(1)尼奥德古菌目和Korarchaeota的单系性,或(2)尼奥德古菌目和海姆达尔古菌门的单系性的自展支持度的演变,同时逐步去除快速进化位点。在去除80%或更多的快速进化位点之前,尼奥德古菌目和Korarchaeota的分组一直得到强烈支持,而尼奥德古菌目和海姆达尔古菌门的单系性从未得到支持(扩展数据图2a)。系统发育信号的显著丧失可能是去除80%或更多快速进化位点后支持度下降的原因。
然而,观察到从NM57比对中逐步排除快速进化位点也强烈支持尼奥德古菌目和海姆达尔古菌门的分组(扩展数据图2b)。为了确定这种效应的原因,我们使用CAT[28,29]和MMseqs2(参考文献[30,31])分类工具,确定了十个尼奥德古菌目代表的contigs的分类特征。尽管这两种工具使用不同的算法,但它们产生了可比的结果。在每个基因组中,大约24%到60%的contigs(图2a,b)被归类为TACK古菌,这些contigs总共占基因组大小的22%到51%(补充图3)。相比之下,3%到35%的contigs被归类为阿斯加德古菌,其总长度占基因组大小的1%到34%(补充图3)。结果表明,这些尼奥德古菌目的MAGs可能含有高水平的污染,这源于错误的分箱,或者,TACK古菌和阿斯加德古菌之间发生了广泛的水平基因转移(HGT),特别是转移到尼奥德古菌目。随后,我们使用读取招募技术,在来自相同采样位置的几个宏基因组中检查了这些MAGs的contigs的分布模式[4,32,33]。我们发现,十个MAGs中的四个的contigs被至少两个样本的读取映射。基于它们在不同宏基因组中的序列组成和差异覆盖模式,四个MAGs中的每个的contigs被划分为两到四个不同的簇(图2c,d)。对于B7_G17_GCA_029856635,其两个较大簇中的contigs被分别用于基于NM57标记推断最大似然系统发育。一个簇中的contigs被置于Korarchaeota的姐妹谱系,而另一个簇中的contigs在阿斯加德古菌内与阿斯加德古菌分支(扩展数据图3)。结果表明,这些尼奥德古菌目的MAGs可能代表来自两个或三个不同群体的嵌合组装体。与其他数据集相比,NM57数据集可能包含更强大的系统发育信号,支持这些MAGs的阿斯加德部分,这可能是因为NM57标记是基于它们在阿斯加德古菌中的分布选择的[4]。尼奥德古菌目的分类特征及其使用含核糖体蛋白的标记集推断的系统发育树(图2,补充图2和4以及补充讨论)表明,这些MAGs的大部分似乎与TACK古菌相关。在系统发育树重建中包含这些嵌合体可能会影响真核生物在古菌中的进化位置。

图2:尼奥德古菌目基因组中contigs/支架的分类特征和聚类
a,b:基于十个尼奥德古菌目代表的计数,分配给热变形菌门(以前称为TACK超门)或阿斯加德古菌的contigs/支架的百分比;使用CAT(a)或MMseqs2(b)工具确定contigs/支架的分类。c,d:基于它们在不同宏基因组中的序列组成和差异平均覆盖度,尼奥德古菌目B7_G17和B20_G9(c)以及B62_G16和S143_49(d)基因组中contigs/支架的层次聚类。
真核生物在海姆达尔古菌门外的出现
我们扩展的阿斯加德古菌基因组有助于解决真核生物在古菌中的位置问题。为此,我们将14个常用的真核生物分类群[4,5,7](补充表3)添加到S67数据集中,生成了一个超级矩阵:ES67。ES67数据集的最大似然树将真核生物置于TACK超门内,作为尼奥德古菌目和Korarchaeota的姐妹进化枝(补充图5a),这在先前的研究中从未被观察到[4,17]。这可能是由于尼奥德古菌目基因组中的阿斯加德古菌序列与真核生物序列之间的高组成相似性造成的。为了减轻远缘外类群对真核生物位置的影响,我们从ES67数据集中去除DPANN、广古菌、Korarchaeota和尼奥德古菌目序列,同时保留50个TACK古菌代表作为外类群;或者,省略所有外类群序列。得到的超级矩阵分别命名为tES67和AsES67。tES67数据集的最大似然分析表明,真核生物与海姆达尔古菌分支,具有高支持度(海姆达尔古菌姐妹分支)(图3a和补充图5b)。基于AsES67数据集的阿斯加德古菌和真核生物的无根最大似然系统发育树也强烈支持海姆达尔古菌姐妹分支的拓扑结构(补充图5c)。
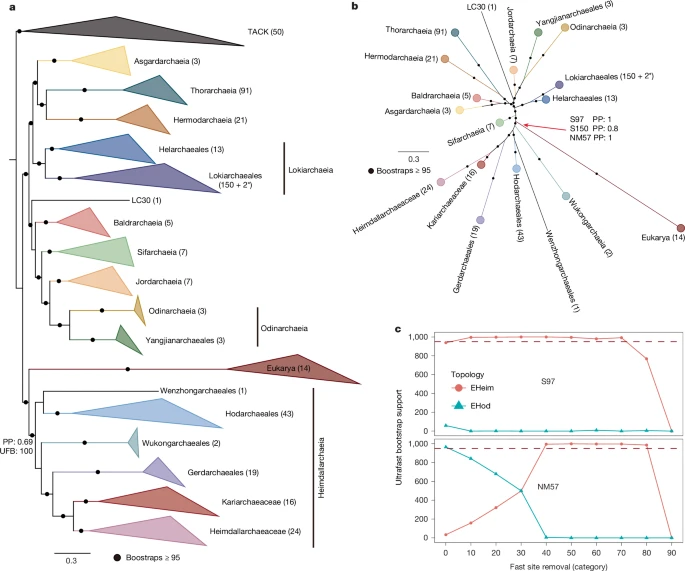
图3:几组串联标记蛋白的系统发育基因组分析,显示真核生物相对于基因组采样的阿斯加德古菌的位置
a:基于461个古菌分类群和14个真核生物的67个串联标记蛋白的最大似然系统发育分析(使用IQ-TREE在LG + C60 + F + G + PMSF模型下推断),使用50个TACK古菌作为外类群(tES67比对;13,348个位点)。洛基古菌目下方的数字代表150个MAGs和两个培养的阿斯加德菌株的基因组。同时,比对进行了SR4重编码,并使用CAT + GTR模型进行贝叶斯推断(补充图6a;两条链;50,000代)。显示了对真核生物节点和最接近的阿斯加德亲属的后验概率(PP)支持(0.69)。b:基于411个阿斯加德古菌分类群和14个真核生物的97个串联标记蛋白的最大似然系统发育分析(使用IQ-TREE在LG + C60 + F + G + PMSF模型下推断)(S97比对;20,067个位点)。此树是无根的。此外,使用CAT + GTR模型对SR4重编码的S97、S150和NM57超级矩阵进行贝叶斯推断(补充图7和8b)。显示了对真核生物节点和最接近的阿斯加德亲属的PP支持(S97的PP = 1,S150的PP = 0.8,NM57的PP = 1)。c:从S97和NM57数据集推断的系统发育树中,真核生物与海姆达尔古菌(海姆达尔古菌姐妹分支,EHeim)或霍达古菌目(霍达古菌目姐妹分支,EHod)分组的自展支持度的演变,同时逐步去除快速进化位点。
基于tES67和AsES67数据集,我们通过逐步去除两个数据集的快速进化位点,检查了速率异质性对真核生物位置的影响。随着异质性梯度的降低,计算最大似然系统发育树,以评估对海姆达尔古菌姐妹分支以及真核生物和霍达古菌目的单系性(霍达古菌目姐妹分支)的支持度。从未观察到对霍达古菌目姐妹分支的支持,而对海姆达尔古菌姐妹分支的支持随着速率异质性的降低而波动(扩展数据图4)。我们推断,对海姆达尔古菌姐妹分支支持度的波动可能归因于系统发育信号的丧失与速率异质性水平之间的动态关系。尽管对SR4重编码的tES67和AsES67数据集的贝叶斯推断显示出海姆达尔古菌姐妹分支的拓扑结构,但支持度较弱(后验概率(PP)支持值分别为0.69和0.5)(图3a和补充图6)。结果表明,S67标记集不包含足够的系统发育信号来解析真核生物和阿斯加德进化枝之间的节点。
为了解决这个问题,我们放宽了标记选择标准。基于扩展的阿斯加德基因组中标记蛋白的保守性,我们从先前报道的在古菌中保守的标记蛋白中选择了另外两个标记集[4,5,7,13,19,21]。第一个标记集由97个标记(S97)组成,这些标记在每个阿斯加德进化枝的至少60%的代表中被发现,而第二个标记集包括150个标记(S150),这些标记在至少80%的阿斯加德古菌基因组中被鉴定出来(补充表4)。从未处理的S97数据集及其基于阿斯加德古菌和真核生物分类群的排除快速进化位点的子集推断的最大似然树,为海姆达尔古菌姐妹分支拓扑结构提供了显著支持(自展支持度大于95)(图3b,c)。相比之下,从未处理的S150数据集推断的最大似然树强烈支持真核生物和霍达古菌目的聚类(扩展数据图5a)。鉴于S150超级矩阵中包含的大量缺失数据可能会使位点进化速率的估计产生偏差,我们还对S150超级矩阵应用了逐个位点去饱和策略。我们观察到,在去除40%的快速进化位点后,对霍达古菌目姐妹分支的支持被对海姆达尔古菌姐妹分支的支持所取代(扩展数据图5b)。真核生物与海姆达尔古菌的分组也通过对SR4重编码的S97和S150数据集的贝叶斯推断得到证实(PP值分别为1.0和0.8;图3b,补充图7)。
在Eme等人的研究[4]中,基于NM57标记集,真核生物被置于海姆达尔古菌内,作为霍达古菌目的姐妹谱系。我们使用扩展的阿斯加德古菌和真核生物对NM57数据集重新进行了系统发育基因组分析。尽管未处理的NM57的最大似然树支持霍达古菌目姐妹分支拓扑结构(补充图8a),但随着快速进化位点的逐步减少,对霍达古菌目姐妹分支的支持度急剧下降,而对海姆达尔古菌姐妹分支的支持度显著上升(图3c)。对SR4重编码的NM57数据集的贝叶斯推断也确定地将真核生物置于海姆达尔古菌之外(PP = 1.0)(补充图8b)。结果表明,先前报道的真核生物与阿斯加德古菌之间的关系[4]可能是人为造成的。使用支持海姆达尔古菌姐妹分支拓扑结构的垂直进化标记进行的额外测试在补充信息中进行了描述(补充表4和补充图9-12)。此外,我们检查了在扩展的阿斯加德古菌采样中ESP的分布,发现先前描述的霍达古菌目特有的ESP也存在于其他阿斯加德进化枝中(补充结果和讨论以及补充图13-16)。
综上所述,我们的系统发育分析表明,真核生物可能在所有采样的海姆达尔古菌多样化之前就已经进化。这些结果与Liu等人先前报道的使用29个标记树推断的情景之一一致[5],但对最近提出的霍达古菌目姐妹假说[4]提出了挑战。与Liu等人的研究[5]相比,我们的结果更可靠,因为它们是通过使用扩展的阿斯加德古菌基因组采样,结合排除外类群、去除快速进化位点以及应用复杂的位点异质进化模型和多个标记集得到的。我们的发现有助于推断真核生物祖先的性质。
最后一个阿斯加德古菌和真核生物共同祖先(LAECA)的时间尺度和特征
我们使用MCMCTree[35],通过宽松的分子钟模型来估计阿斯加德门内关键节点的分歧时间。在分子定年分析中,应用了古菌根年龄范围为42.9亿至38亿年前(Ga),其他化石校准采用均匀年龄先验(补充表5)。最后一个阿斯加德古菌共同祖先(LAsCA)出现在约33.8亿年前(置信区间(CI),37.2亿至30.6亿年前)的太古代(补充图17)。海姆达尔古菌和洛基古菌共同祖先的年龄估计约为31.6亿年前(CI,35.4亿至28.2亿年前),处于中太古代。作为真核生物的姐妹谱系,冠状海姆达尔古菌在31.2亿至22.6亿年前分歧,早于大氧化事件[36]。结果表明,真核生物的主干谱系在大氧化事件之前进化,这与Betts等人报道的主干真核生物的时间尺度基本一致[3]。最近,基于ATP合酶的进化历史,有人提出真核生物在26.7亿至21.9亿年前从海姆达尔古菌内的霍达古菌目分歧出来[37]。这在时间上与我们对海姆达尔古菌和真核生物分歧的估计部分重叠。
我们使用概率基因树 - 物种树协调方法[38](合并似然估计(ALE))来推断阿斯加德古菌物种树关键节点的祖先事件和基因内容。为了完善代谢重建,选择了上述完整性超过80%且污染率小于5%的古菌基因组子集进行ALE分析。该子集包括235个阿斯加德古菌、47个广古菌和60个TACK古菌,平均完整性为88.5%,平均污染率为2.7%(补充表2和3)。在物种树中,从LAsCA到海姆达尔古菌祖先的进化过程中,祖先基因内容逐渐增加(补充图18)。沿着进化路线,基因丢失率也逐渐增加,这意味着在海姆达尔古菌辐射之前发生了大量的基因获得。
接下来,我们基于ALE报告的节点中基因的存在概率(补充表6)以及阿斯加德古菌基因组中的基因频率(补充表7),重建了阿斯加德古菌关键祖先的代谢特征。最近对两个培养的阿斯加德古菌,即Ca. P. syntrophicum和Ca. L. ossiferum的研究表明,它们具有通过互营方式厌氧降解氨基酸的能力[1,2]。基于这些生理特征,推断LAsCA可能是一种降解氨基酸的厌氧菌,产生氢气和脂肪酸作为副产物[39]。然而,我们的分析表明,这两个培养的阿斯加德菌株及其三个密切相关的MAGs缺乏古菌Wood-Ljungdahl途径(WLP)的H4MPT甲基分支的大多数关键基因[40],但编码细菌WLP的H4F甲基分支中的三个关键酶:甲酰四氢叶酸合成酶(fhs)、亚甲基四氢叶酸脱氢酶(folD)和亚甲基四氢叶酸还原酶(metF)(补充图19)。这个部分的H4F分支可以将甘氨酸、丝氨酸和组氨酸转化为甲酸,介导种间电子转移[1](补充图20)。相比之下,推断LAsCA含有完整的古菌WLP,但缺乏细菌WLP中的Fhs(图4a,补充表6和7以及补充图19和20)。它还编码各种NiFe氢化酶(补充表6和补充图21)。这些结果表明,LAsCA是一种依赖氢气的化能自养生物。太古代海洋中丰富的二氧化碳[41]和蛇纹石化产生的海洋氢气[42]将为这些生物提供充足的底物。

图4:关键阿斯加德古菌祖先的代谢重建以及海姆达尔古菌谱系中Wood-Ljungdahl途径(WLP)的分布
a. 从阿斯加德共同祖先向洛基古菌和海姆达尔古菌祖先的转变。基于ALE结果,推断最后一个阿斯加德古菌共同祖先(LAsCA)是一种依赖氢气的化能自养生物。推断所有四个祖先中都存在古菌的Wood-Ljungdahl途径(WLP),因此,在最后一个阿斯加德古菌和真核生物共同祖先(LAECA)中也可能存在该途径。预测在海姆达尔古菌祖先中存在与产乙酸相关的四个基因(pta、ack、acs和acd),这表明LAECA可能是一种依赖氢气的厌氧产乙酸菌。b. 海姆达尔古菌中WLP关键酶的分布。作为海姆达尔古菌内的基础分支,霍达古菌目拥有完整的WLP,这支持了海姆达尔古菌祖先中存在该途径。在从霍达古菌目向海姆达尔古菌科和卡里古菌科的转变过程中,WLP的这些关键酶似乎逐渐缺失。实心圆表示在该进化枝至少一半的基因组中检测到该基因。半实心圆表示在该进化枝不到一半的基因组中检测到该基因。EMP:糖酵解途径(Embden–Meyerhof–Parnas);NOPPP:非氧化戊糖磷酸途径;RuMP:核酮糖单磷酸途径;sp/np-ED:半磷酸化/非磷酸化的恩特纳-杜多罗夫途径;TCA循环:柠檬酸循环;AA:氨基酸代谢;β:β-氧化;H4F:四氢叶酸甲基分支;H4MPT:四氢甲烷喋呤甲基分支;Fdh:甲酸脱氢酶;Mvh/Hdr:F420非还原性氢化酶和异二硫化物还原酶复合物;Pyr:丙酮酸;Ech:能量转换氢化酶;F420:辅酶F420还原性氢化酶;Mcr:甲基辅酶M还原酶;Nucl:核苷酸;H:海姆达尔古菌科;K:卡里古菌科;G:格尔德古菌目;Hod:霍达古菌目;W:悟空古菌;fwd:甲酰甲烷呋喃脱氢酶;ftr:甲酰甲烷呋喃-四氢甲烷喋呤N-甲酰基转移酶;mch:亚甲基四氢甲烷喋呤环化水解酶;mtd:亚甲基四氢甲烷喋呤脱氢酶;mer:5,10-亚甲基四氢甲烷喋呤还原酶;cdh:乙酰辅酶A脱羰基酶/合成酶,CODH/ACS复合物。
洛基古菌门包括海拉古菌目和洛基古菌目。发现海拉古菌目的所有成员(13个宏基因组组装基因组,MAGs)都编码一种类似于在氧化丁烷的古菌中发现的甲基辅酶M还原酶样酶(补充表7)。ALE分析推断在洛基古菌祖先中存在甲基辅酶M还原酶样酶(补充表6),这表明该祖先具有通过将脂肪酸的β-氧化与逆向WLP耦合来厌氧降解碳氢化合物的潜力(图4a和补充图20)。
与Eme等人的结果相反[4],我们的推断表明海姆达尔古菌和霍达古菌目祖先也拥有完整的古菌WLP途径(图4a,补充图20和补充表6)。我们发现,在Eme等人[4]使用的海姆达尔古菌的MAGs(包括三个霍达古菌目的基因组)中,编码古菌WLP中几种关键酶的基因缺失。然而,我们展示了22个霍达古菌目的新基因组,代表了两个新科和七个新属,其中几个基因组或谱系含有完整的古菌WLP(图4b)。作为海姆达尔古菌中的基础分支,霍达古菌目中古菌WLP的存在支持了冠状海姆达尔古菌的自养生活方式。除了WLP之外,推断冠状海姆达尔古菌编码磷酸转乙酰酶(pta)和乙酸激酶(ack),这些酶是将乙酰辅酶A转化为乙酸所必需的,以及一种能量转换氢化酶(ech),它有可能作为离子转运化学渗透偶联位点,还有ATP合酶(补充表6和7以及补充图20)。基因频率分析表明,ack基因仅在海姆达尔古菌的成员中发现(补充图21a)。这些数据支持了海姆达尔古菌祖先为依赖氢气的厌氧产乙酸菌的推断,正如先前的一项研究所示[45]。
作为冠状海姆达尔古菌的祖先,LAECA也可能是一种依赖氢气的产乙酸古菌。我们的代谢证据支持氢气假说,该假说认为真核生物的起源最初是由消耗氢气的古菌宿主与在缺氧条件下生存的产氢气的原线粒体之间的互营伙伴关系介导的,原线粒体是兼性需氧菌[46,47]。我们在38个霍达古菌目成员和16个卡里古菌科成员中鉴定出不完整的细胞色素bd-I泛醇氧化酶和不完整的细胞色素o泛醇氧化酶(补充图21b和补充表7)。然而,推断在海姆达尔古菌祖先中仅存在细胞色素o泛醇氧化酶的两个亚基(补充图20和补充表6),这些亚基可能是通过与细菌伙伴的水平基因转移获得的。这些不完整的复合物可能缺乏将电子运输或将分子氧还原为水的能力。推断在海姆达尔古菌祖先中存在对氧气极其敏感的酶,如乙酰辅酶A脱羰基酶/合成酶(cdh)、2-氧代酸:铁氧还蛋白氧化还原酶(kor、vor和por)和NiFe氢化酶(补充图20和补充表6和7),再结合当时缺氧的深海环境[46],表明该祖先一定是在厌氧条件下生长的。
总之,我们使用复杂的系统发育基因组方法,并结合对阿斯加德古菌的显著扩展采样,重新评估了阿斯加德古菌、尼奥德古菌目和真核生物之间的关系。我们揭示了尼奥德古菌目的基因组可能代表主要由TACK古菌和阿斯加德古菌序列组成的嵌合组装体。这些奇特的组装基因组会显著影响真核生物在古菌中的系统发育位置。在排除外类群并使用多个标记集后,真核生物被可靠地置于阿斯加德古菌内,作为海姆达尔古菌的姐妹分支,而不是嵌套在与霍达古菌目分支的海姆达尔古菌内。祖先重建推断真核生物起源的宿主谱系是一种依赖氢气的厌氧化能自养生物。我们的发现纠正了现有知识,并填补了真核生物早期进化过程中的一些空白。海姆达尔古菌多样性的进一步扩展及其分离将有助于完善真核生物起源的进化重建[48]。
转载本文请联系原作者获取授权,同时请注明本文来自孙学军科学网博客。
链接地址:https://wap.sciencenet.cn/blog-41174-1485528.html?mobile=1
收藏

