 精选
精选
CRISPR技术对寻找抗癌药研究的影响
自 2012 年作为基因编辑工具被发现以来,CRISPR-Cas9 一直被吹捧为药物发现的转折点。事实上,基于 CRISPR 的技术具有前所未有的能力,可以通过系统测试靶向基因改变的细胞效应来询问疾病的机制基础 (1)。癌症特有的疾病标志,例如不受控制的细胞增殖和耐药性,很容易用于大规模平行 CRISPR 筛选。在这些筛选中,携带向导 RNA 的单个细胞也充当目标 DNA 编辑的条形码,相对于同一测试池中的其他细胞,可能会获得竞争优势或减弱的增殖优势。这允许在单个实验中扰动数千个基因。在癌细胞系 (CCL) 和癌症衍生组织中生成的遗传图谱现在支持新药开发的合理化以非凡的速度和经济性,并可能缩短从实验室到床边的时间。
与通常必需的基因相反,识别环境依赖性必需的必需基因(在确定的癌症类型或遗传背景中细胞增殖所必需的)是确定避免化疗中常见毒性的药物靶标的关键步骤 (2)。系统地挖掘基因组以寻找此类治疗机会的最大努力是癌症依赖图 (DepMap),它利用多种癌症类型的 >1000 个 CCL 来确定 17,645 个基因中每个基因的 CRISPR 介导的扰动(多次导致基因“敲除”)后的单突变体适应度 (SMF) 测量值 (3)。结果是基因× CCL 矩阵为 ~1.75e7FITNESS 测量可用于识别最佳癌细胞增殖所需的先前未预料到的基因。这个开放且不断增长的数据矩阵不断被挖掘,并已成为新发现的缓存。例如,发现有丝分裂驱动蛋白样蛋白 KIF18A 在非整倍体癌症中是必需的 (4, 5),而 Werner 解旋酶在 DNA 错配修复通路受损的癌细胞中是必需的 (6)。这两种蛋白质现在是几种候选药物的靶标。
当针对给定双突变体(在相同基因中包含两个突变或两个不同基因的癌细胞)观察到的适应度表型偏离基于两个单突变表型之和的预期表型 (7) 时,就会发生遗传相互作用 (GI)。如果双突变体的增殖效果优于预期,则称该基因对表现出阳性 GI。相反,阴性 GI 与增殖缺陷相关,该缺陷比单基因突变效应预期的更严重。范围从合成疾病(细胞活力降低)到最极端的情况,合成致死(细胞死亡)。合成致死 GI 代表了潜在靶标的开采不足的来源,并且基于使用 CCL 中的大规模单基因扰动筛选构建的癌症遗传依赖图谱赢得了关注和投资。此外,这些机会有可能支持创造具有更大治疗窗口(具有可耐受毒性的有效药物剂量范围)的药物,假设与在癌细胞中观察到的 GI 相比,正常组织的健康对此类 GI 的依赖较小。
GI 概念可以扩展到给定表型的许多突变相互作用,因此即使借助 CRISPR 技术,也能迅速超越人类细胞中任何系统实验方法的限制。这阻碍了对癌症中通常发现的基因突变的复杂相互作用进行建模的能力。与此同时,推理或机器学习方法可以帮助填补这些空白。事实上,此类努力的目标是使用机器学习模型创建个性化治疗,这些模型引用 CCL 表型数据库(如 DepMap)和从肿瘤中检索的基因组序列信息来预测药物反应 (8)。虽然 GI 在肿瘤学领域通常与基于癌细胞适应性和增殖的合成致死性相关联,但 GI 的证据出现在专注于其他适应性表型(如胚胎和成人发育)的遗传观察中。
CRISPR 支持药物靶点发现
基于 CRISPR 的癌细胞系筛选和表型读数(如细胞生长)可以促进发现可能成为治疗靶点的遗传相互作用。这些数据集和其他信息的机器学习可用于模拟药物对肿瘤微环境的影响,并更好地预测对患者的影响。
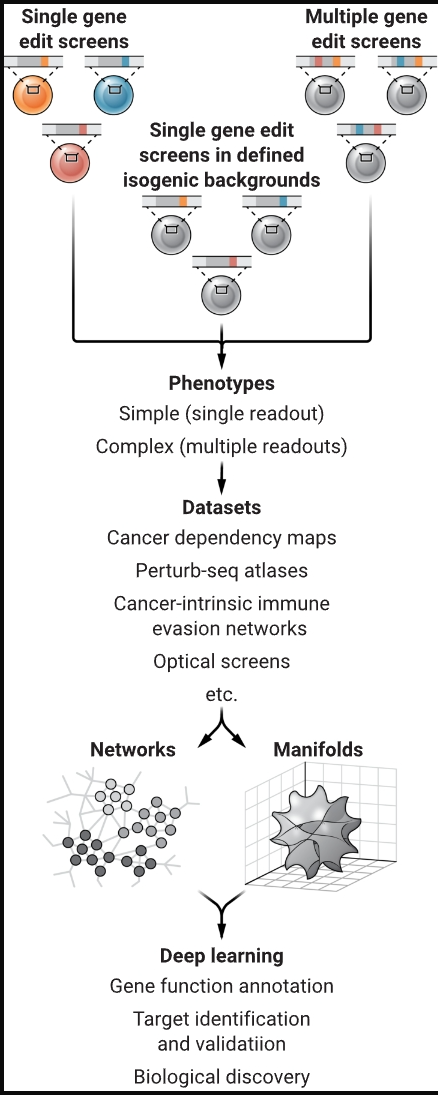
制图:N. BURGESS/SCIENCE
GI 也可以使用串联基因 CRISPR 扰动技术进行分类,该技术可以识别单基因消除方法无法识别的潜在药物靶标 (9, 10)。这些筛选解决了重要的生物学问题,包括 CRISPR 介导的 DNA 损伤反应对筛选结果解释的不良影响;冗余基因 (旁系同源物) 的作用,可能使癌细胞能够承受任何一个旁系同源基因的丢失;以及发现可用作治疗靶点的 GI。使用这种串联基因扰动筛选出现的药物开发新机会包括 DUSP4/6(双特异性磷酸酶 4 和 6)酶,这些酶被证明可以共同促进突变 NRAS(神经母细胞瘤 ras 病毒癌基因同源物)或 BRAF(B-Raf 原癌基因,丝氨酸/苏氨酸激酶)的致癌活性 (9).鉴于在整个基因组中生成数据矩阵需要大量的组合双基因扰动排列,靶向有限数量特定基因类别(如转录因子或磷酸化酶)的 CRISPR 文库应该有助于实现组合基因扰动事件的饱和水平,从而提高这些具有挑战性的筛选中的基因覆盖率。
识别 GI 的另一种方法是构建携带癌症相关突变的同基因(否则遗传相同)细胞系,并使用全基因组扰动文库筛选它们以推断 GI。这种方法的优点是可以精确定义查询(一种经常与癌症相关的突变),并在实验室之间共享细胞系以进行后续研究。一个缺点是,感兴趣的突变可能会产生意想不到的选择压力,随着时间的推移促进细胞群遗传漂变,从而使细胞系成为不太可靠的疾病表型报告基因。此外,在其自然病程中从未发生过癌症相关突变的细胞可能无法忠实地模拟疾病的病理生理学。这种同基因方法是率先使用基于全基因组 RNA 干扰的筛选,通过靶向信使 RNA 进行降解来沉默基因表达。初步研究侧重于一对 KRAS 野生型和突变型人结直肠细胞系,并恢复了 PLK1(polo 样激酶 1)等有丝分裂调节基因在 KRAS 信号转导中的作用 (11)。最近,这种方法已被采用,使用混合全基因组 CRISPR 筛选来识别与脂肪酸代谢相关的 GI,这一过程在癌症中通常不受控制,并且正在作为治疗靶标积极研究 (12)。因此,具有既定查询的 CRISPR 筛选结果可以解释为适应性依赖性,这可能有利于识别癌症脆弱性,但可能产生与两个或多个基因的急性同时扰动不同的结果。
双链断裂 (DSB) 是高细胞毒性损伤,如果不修复,单个 DSB 就足以导致细胞致死 (13)。因此,基于 CRISPR 的基因编辑必然会引发触发修复的 DNA 损伤信号。然而,也会发生修复错误事件,导致染色体畸变,例如易位、大缺失和单个或多个染色体的碎裂(称为染色体碎裂的过程)。CRISPR 干扰 (CRISPRi) 等 CRISPR 技术需要将核酸酶缺陷型 Cas9 变体融合到 Krüppel 相关盒 (KRAB) 阻遏蛋白结构域,从而能够在不引起 DSB 的情况下进行靶向基因转录抑制 (14)。此外,现在可以以可编程方式切割 RNA 的 CRISPR 相关 (Cas) 酶(如 Cas13d)已被部署以降低 RNA 水平的基因表达,从而避免不需要的基因组变化。这种替代 CRISPR 方法的另一个好处是对基因的部分功能丧失效应,与使用经典 CRISPR 方法实现的完全基因消融相比,它可以更忠实地模拟药物治疗结果。
类器官捕获了在 CCL 中经常丢失的完整肿瘤的重要特征。它们由自我更新的细胞产生,这些细胞保留了不断构建次级细胞类型的能力。因此,在类器官中使用 CRISPR 选择性消耗潜在抗癌靶标可以产生复杂的表型,这些表型可以更保真地捕捉具有完整细胞结构的肿瘤中靶标禁用的影响。同样,应用于类器官(或三维细胞培养中的癌细胞系)的大规模平行遗传筛选可以提供一种在更多生理条件下识别支持癌细胞行为的基因的方法。因此,使用类器官进行此类筛选有可能识别在典型 CCL 培养条件下未报告的基因。然而,在这些更具挑战性的生长环境中执行基因组规模筛选的困难(即,使用细胞外基质支持,更专业的培养基配方)限制了联盟生成与 CCL 类似的综合遗传数据库的机会。然而,单独体外培养的癌组织(CCL 或类器官)并未报告靶向在肿瘤和正常宿主细胞界面起作用的基因的治疗剂的影响(例如检查点抑制剂诱导的免疫细胞杀死癌细胞)。这需要寻找新的方法来评估肿瘤-微环境相互作用,并考虑筛选包含其他遗传信息的数据。在体内执行的 CRISPR 筛选也被证明是揭示关键癌症进展调节因子的有效方法,这些调节因子不一定存在于癌症中,而是存在于宿主细胞中 (1)。
CRISPR 能够快速、系统地核算癌细胞中的选择性遗传脆弱性(见图)——无论是以癌症类型依赖性和合成致死相互作用的形式——都激励了药物开发人员将新的遗传观察结果与在患者中产生类似选择性反应特征的治疗药物相匹配。因此,代表非典型或难以用药的靶标的基因,例如细胞类型决定转录因子,由于对此类基因在癌症中的功能有更大的信心,人们加倍努力开发药物。然而,嵌入未来药物开发的 CRISPR 平台还可以通过实现对靶上和脱靶效应的大规模平行基准测试来识别意想不到的机会或威胁,从而加快基因到药物的时间表 (15)。此外,碱基和引物编辑技术的出现 (1) 能够系统地评估编码经过验证的癌基因和肿瘤抑制因子的整个基因序列中针对靶向碱基对替换的药物活性,从而告知癌症中潜在的先天性和必需的耐药性突变,以及看到患者对给定药物的反应的可能性。虽然这里没有讨论,但基于 CRISPR 的疗法也可以用于解决以前难以用药的靶点,这些靶点不适合更传统的基于小分子的方法。因此,即使 CRISPR 生成的基因功能目录正在缩短识别潜在药物靶点的时间,但 CRISPR 衍生技术的持续发展对于将这些新发现的知识转化为下一代药物至关重要。
转载本文请联系原作者获取授权,同时请注明本文来自孙学军科学网博客。
链接地址:https://wap.sciencenet.cn/blog-41174-1456943.html?mobile=1
收藏

