博文
超越基准:迈向数据集特异性单细胞RNA-seq管道性能的预测模型
||
超越基准:迈向数据集特异性单细胞RNA-seq管道性能的预测模型
单细胞RNA测序(scRNA-seq)通过在单细胞分辨率下实现转录组范围内的基因表达定量,已经彻底改变了生物医学。scRNA-seq数据的分析通常是复杂的,需要多个相互作用的组件,如细胞过滤、归一化、降维和聚类,这些组件的选择可能影响下游方法的结果。为了满足这一需求,计算方法的开发激增,截至2021年底,开发了1000多个工具,仅为细胞聚类就开发了270多个工具。
随着这个大型工具集的开发,可以应用于给定的scRNA-seq数据集的可能管道的组合数量,其中给定的管道由每个步骤的方法组合以及各自的参数选择来定义。考虑一个不切实际的简单例子:如果有3个分析步骤(例如,过滤,归一化,聚类),每个步骤有4种计算方法,每种方法有2种可能的参数组合,那么有(4x2)3 = 512种可能的管道。考虑到步骤、方法和参数的可能性要大得多,在实践中,可应用于scRNA-seq数据的合理管道的数量可能高达数千甚至数百万。因此,这导致了一个重要的问题:我们如何为我们的数据集选择“最佳”的管道?
为了解决这个问题,该领域在很大程度上依赖于基准研究。例如,众多论文对scRNA-seq 工作流程的多个阶段进行了综合评估,包括聚类、伪时间排序、降维、数据集整合、数据输入和基因选择。这些阶段可以组合在框架中,如pipeComp,将多个管道步骤集成在一起,对组合进行基准测试。虽然这种基准研究非常有价值,但它们可能受到两个原因的限制。首先,极其庞大的管道组合数量意味着不可能对所有方法组合进行详尽的基准测试,因此不同阶段之间的相互作用可能会影响性能。其次,管道性能通常是特定于数据集的,这意味着平均性能最好的管道可能不是给定数据集的最佳选择。
这种可能管道的组合数量并不是单细胞分析所独有的。在监督机器学习(ML)中,试图优化可能的数据集、处理管道、算法和超参数选择导致了自动化机器学习(AutoML)的兴起。AutoML算法试图使用先进的统计技术,如贝叶斯优化,自动选择最优的超参数组合。重要的是,以前的AutoML工作已经表明,与其考虑一组详尽的可能管道,不如在一个大但固定的子集上进行测试,然后从其中推荐一个就足够了。然而,在监督机器学习背景下,在一个持有数据集上优化预测模型准确性的目标不容易适用于单细胞分析。相比之下,大多数单细胞管道是无监督的,并且在AutoML和无监督基因组分析的交叉点上开发的方法很少。
在这里,Fang等人研究了AutoML方法是否可以适应并应用于scRNA-seq分析管道的优化,以便为给定的数据集推荐一个分析管道。为此,作者们将288个scRNA-seq聚类管道应用于86个数据集,其中数据集产生24,768个独特的聚类输出,并通过聚类纯度和基因集富集指标来量化每个数据集的性能。因此,作者们创建了单细胞管道预测(SCIPIO-86),这是单细胞管道性能的第一个数据集。然后,作者们开发了一组有监督的机器学习模型,使用管道和数据集特征的组合来预测给定数据集上给定管道的性能(图1A)。作者们研究了针对数据集和管道定制的预测准确性,并将这些预测与仅使用管道特征作为输入特征的预测进行了比较。他们进一步研究了这种预测的相关性,通过将86个已识别的集群与现有细胞标签的重叠程度与作者们的模型预测的管道的变化相关联,以确定其表现是否良好。最后,作者们研究了scRNA-seq数据集的哪些特征使其更容易预测哪些管道将适用于它。该研究为scRNA-seq分析管道推荐系统的开发提供了基础,并且训练数据可在zenodo.org/records/10342364上公开获取,以鼓励该领域的进一步方法开发。
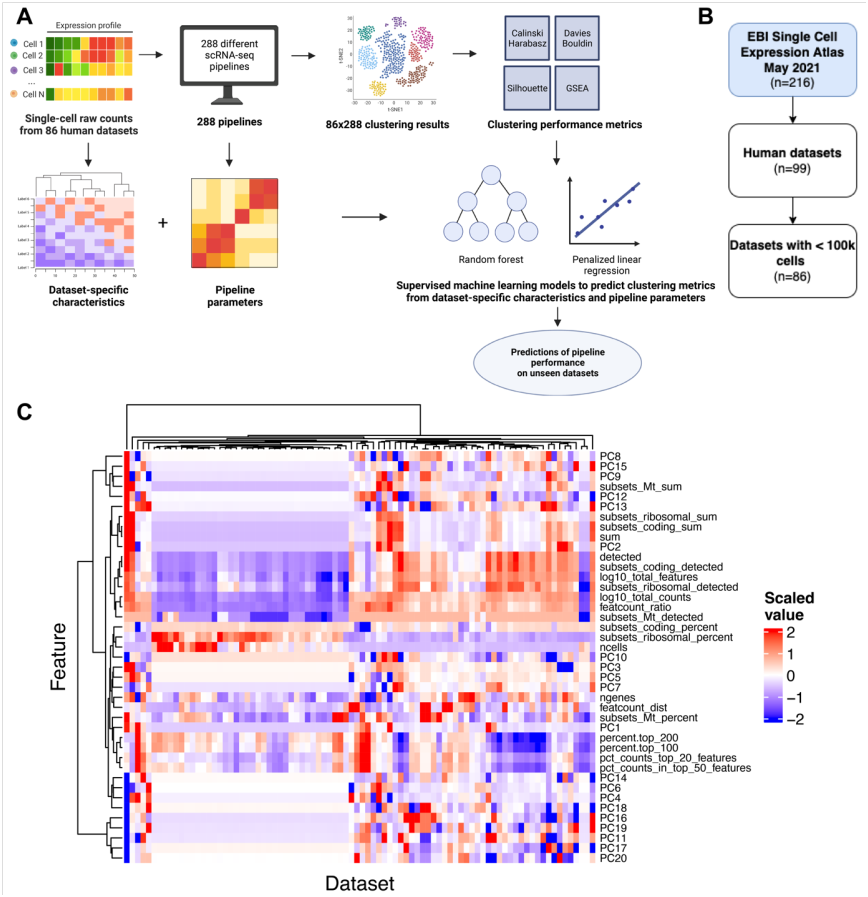
图1 机器学习工作流程概述。A在每个数据集上运行288个聚类管道,每个管道的都用4个无监督指标进行量化。然后计算数据集和管道的特定特征,并将其作为监督机器学习模型的输入,以预测度量值。B 本研究选择了截至2021年5月EBI单细胞表达图谱中包含<100k个细胞的86个人类数据集。C 86个数据集的特征,用作特定数据集管道性能预测模型的输入。这些包括经常用于细胞水平质量控制指标的中位数(例如,线粒体计数的百分比),以及每个数据集共同分解的平均表达值的主成分。每个特征在训练集中按标准正态分布进行缩放。使用训练集中每个特征缩放前的均值和方差对测试集中的对应特征进行缩放,以防止训练-测试泄漏
参考文献
[1] Cindy Fang, Alina Selega, Kieran R Campbell. Beyond benchmarking: towards predictive models of dataset-specific single-cell RNA-seq pipeline performance. bioRxiv 2024.01.02.572650; doi: https://doi.org/10.1101/2024.01.02.572650
以往推荐如下:
5. EMT标记物数据库:EMTome
8. RNA与疾病关系数据库:RNADisease v4.0
9. RNA修饰关联的读出、擦除、写入蛋白靶标数据库:RM2Target
13. 利用药物转录组图谱探索中药药理活性成分平台:ITCM
19. 基因组、药物基因组和免疫基因组水平基因集癌症分析平台:GSCA
22. 研究资源识别门户:RRID
24. HMDD 4.0:miRNA-疾病实验验证关系数据库
25. LncRNADisease v3.0:lncRNA-疾病关系数据库更新版
26. ncRNADrug:与耐药和药物靶向相关的实验验证和预测ncRNA
28. RMBase v3.0:RNA修饰的景观、机制和功能
29. CancerProteome:破译癌症中蛋白质组景观资源
30. CROST:空间转录组综合数据库

https://wap.sciencenet.cn/blog-571917-1419396.html
上一篇:TimeTalk:破译早期胚胎发育过程中的细胞通讯
下一篇:DeepCCI:从单细胞RNA测序数据中识别细胞-细胞相互作用的深度学习框架