博文
2018 MSB 破译合成人类肠道微生物组群落中的微生物相互作用
||
原文链接:Deciphering microbial interactions in synthetic human gut microbiome communities - PMC (nih.gov)

摘要
人类肠道微生物群的组装和稳定性所受的生态学影响仍未得到解决。我们开发了一种可通用的模型指导框架,通过对低阶群落的时间分辨测量来预测高维群落。这种方法被用于解密多样化人类肠道微生物组合成群落中的微生物相互作用。我们发现,与高阶相互作用相比,成对相互作用是多物种群落动态的主要驱动力。推断出的生态网络呈现出高比例的负向相互作用和频繁的正向相互作用。在该网络中发现了生态驱动因素和响应性受体物种。我们的模型证明,普遍存在的正负相互作用拓扑结构可以通过实施负反馈循环来平衡单物种适应性水平的差异,从而实现稳健的共存。我们的研究表明,负交互作用可产生初始物种比例的历史依赖性反应,而这种反应往往并非源于双稳态性。对细胞外代谢物的测量阐明了单物种的代谢能力和微生物相互作用的潜在分子基础。总之,这些方法确定了与人类相关的主要肠道物种的生态作用,并阐明了微生物群落的设计原则。

图1 合成人类肠道微生物组菌群高通量表征的实验设计
A 12成员合成生态学的系统发育树,涵盖肠道微生物组的主要门,包括放线菌门、拟杆菌门、厚壁菌门和变形菌门。使用通过PhylSift获得的单拷贝标记基因的串联比对进行系统发育分析。最大似然树是使用默认选项生成的。比例尺表示排列中每个位置的替换数量。
B 本研究的实验设计示意图。基于600 nm(OD600)处的吸光度测量,使用大约1:1或19:1的初始比例将物种组合到微量滴定板中,使用液体处理机器人操作。大约每12小时收集一次样本,用于多重16S rRNA基因测序(黑圈)。使用与Illumina平台兼容的双索引引物,使用V3–V4区域的多重16S rRNA基因测序测量相对丰度(堆叠条形图,右下角)。通过使用1:20稀释将等分的群落转移到新鲜培养基中,以大约24个间隔(紫色条,顶部)进行连续转移。同时,对单种和联合体进行时间分辨OD600测量。
表1 基于先前文献的研究中使用的物种表以及与人类疾病的关联。向上或向下的箭头分别表示正关联或负关联。
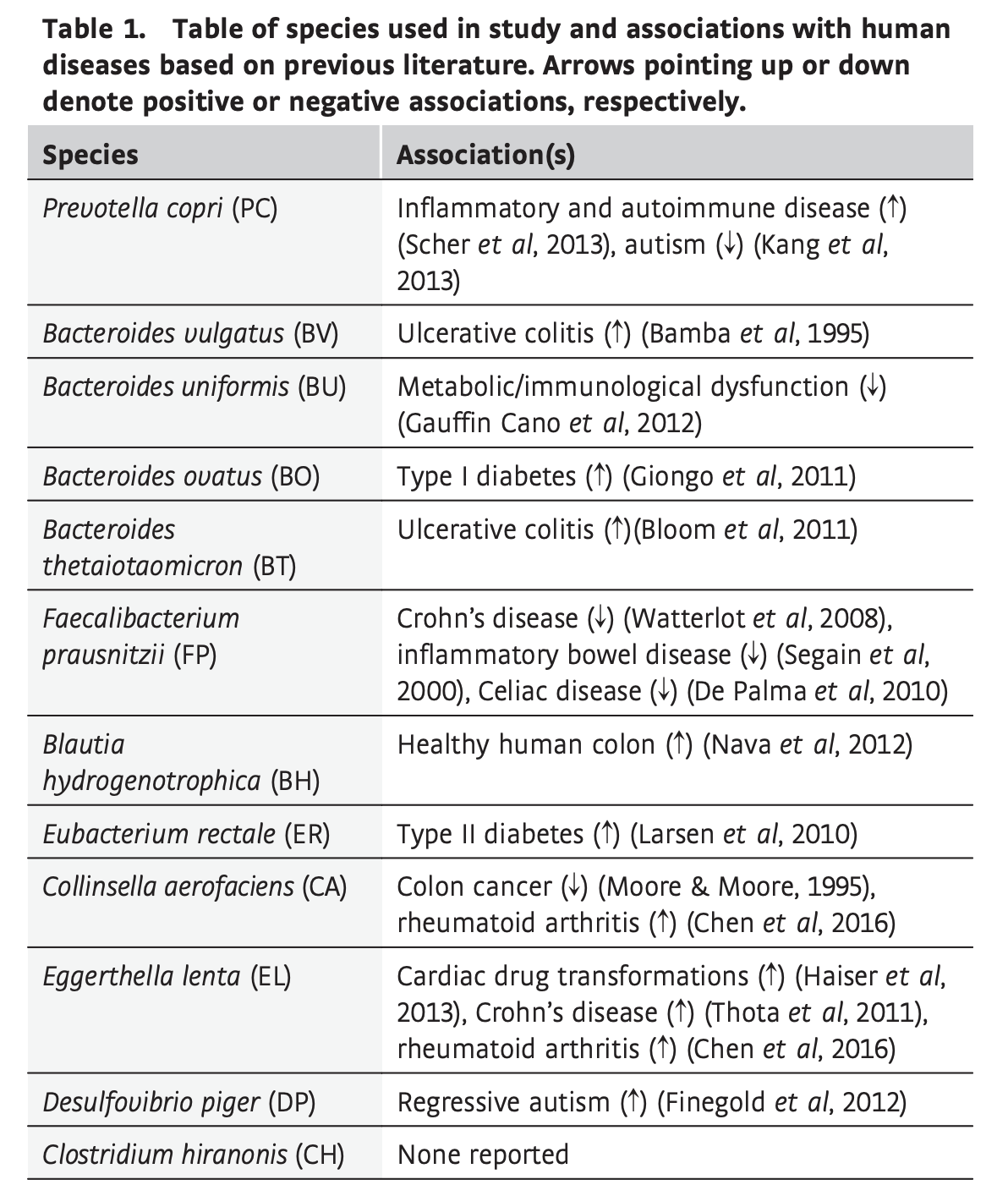
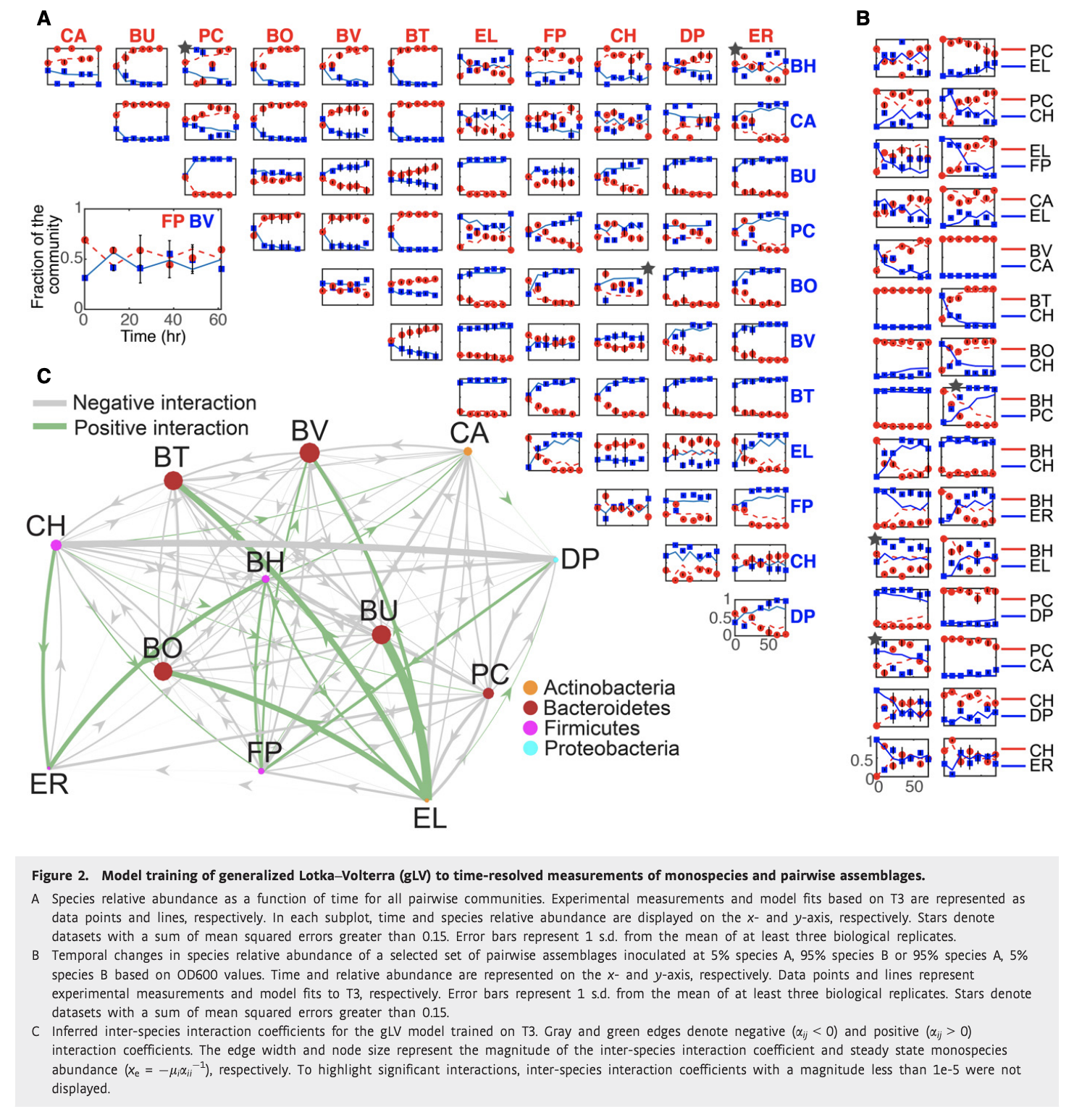
图2 广义Lotka–Volterra(gLV)对单物种和成对组合的时间分辨测量的模型训练
A 所有成对群落的物种相对丰度作为时间的函数。基于T3的实验测量和模型拟合分别表示为数据点和数据线。在每个小图中,时间和物种的相对丰度分别显示在x轴和y轴上。星形表示均方误差之和大于0.15的数据集。误差条表示至少三个生物重复的平均值的1 s.d。
B 基于OD600值,以5%物种A、95%物种B或95%物种A、5%物种B接种的一组选定成对组合的物种相对丰度的时间变化。时间和相对丰度分别表示在x轴和y轴上。数据点和线分别表示实验测量值和T3的模型拟合。误差条表示至少三个生物重复的平均值的1 s.d。星形表示均方误差之和大于0.15的数据集。
C 在T3上训练的gLV模型的推断物种间相互作用系数。灰色和绿色边表示负(αij<0)和正(αij>0)相互作用系数。边缘宽度和节点大小分别表示物种间相互作用系数和稳态单物种丰度(x e=-μiαii−1)的大小。为了强调显著的相互作用,没有显示大小小于1e-5的物种间相互作用系数。
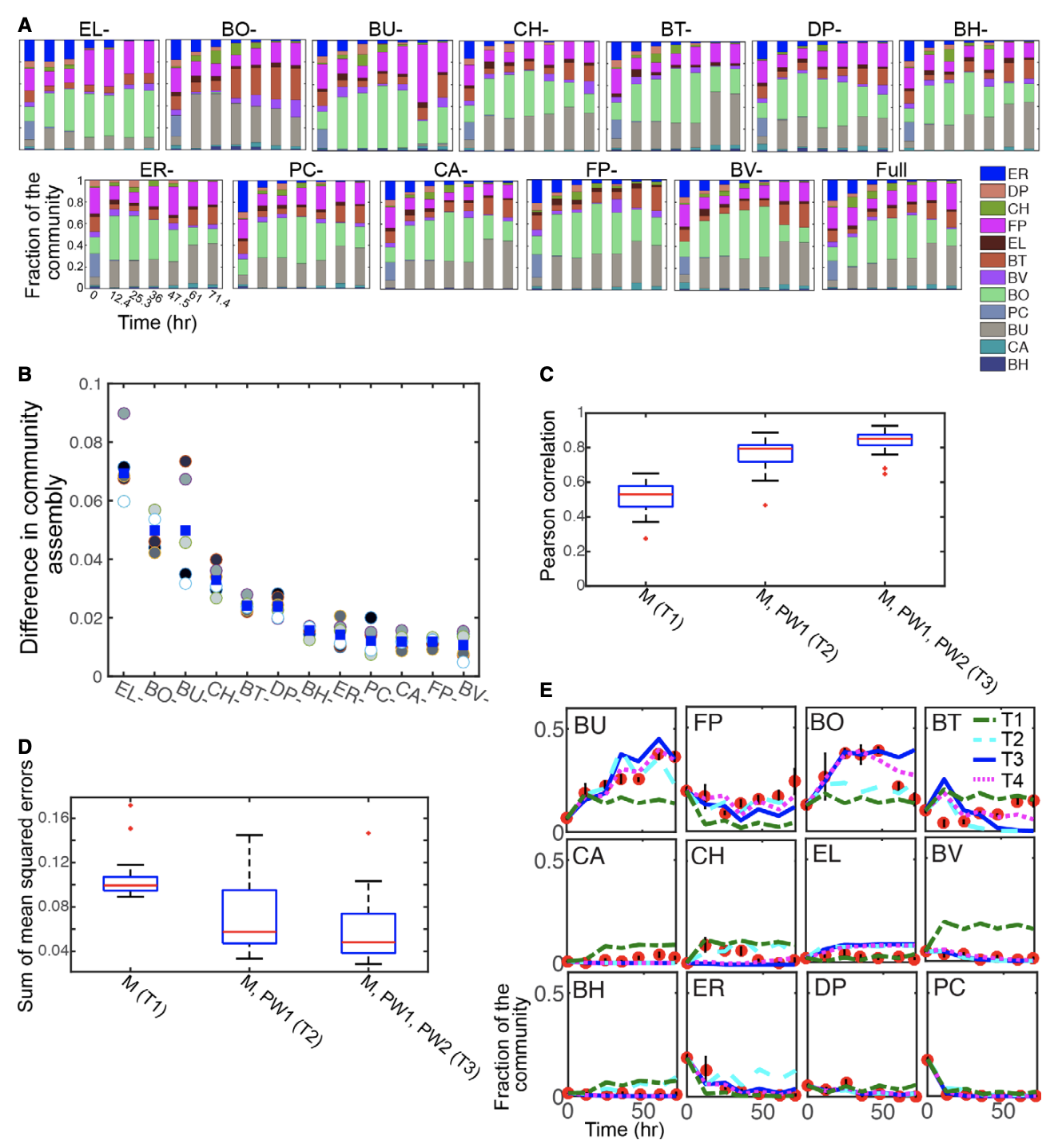

图3 参数化Lotka-Volterra模型对信息丰富的多物种群落的时间序列数据的验证
A 所有11个和12个成员(完整)多物种群落的堆积条形图。每个子图上方的文字表示群落中缺失的生物体。时间和相对丰度分别表示在每个子图的x轴和y轴上。颜色代表群落中的不同生物。
B 单一物种敲除群落的群落组装差异。群落组装的差异计算为![]()
其中i和j分别表示种类和时间点。X代表缺乏生物体X的单一物种敲除群落。𝑣表示全(12个成员)和单物种敲除群落中共享物种集的重整化相对丰度。在这里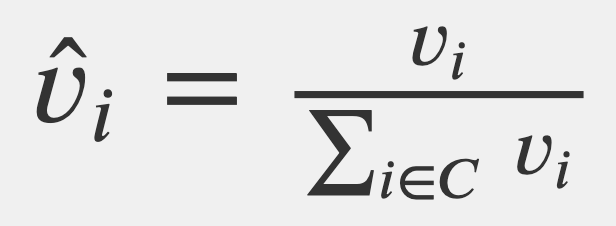 其中,C表示完整和单一物种敲除群落中11个共享生物的集合。数据点分别表示生物重复(n=6,圆形)和生物重复的平均值(蓝色正方形)。
其中,C表示完整和单一物种敲除群落中11个共享生物的集合。数据点分别表示生物重复(n=6,圆形)和生物重复的平均值(蓝色正方形)。
C 三个模型训练集(左)的Pearson相关系数的箱形图,包括T1:单种(M);T2:M和成对1(PW1);以及T3:M、PW1和成对2(PW2)。在每个框上,红线表示中值,框的边缘是第25和第75个百分位数,须状延伸到最极端的数据点,异常值绘制为红十字。
D 在T1、T2或T3训练集上训练的多物种群落的时间分辨测量的模型预测的均方误差之和的方框图。在每个框上,红线表示中值,框的边缘是第25和第75个百分位数,须状延伸到最极端的数据点,异常值绘制为红十字。
E 在T1–T4上训练的模型预测与整个群落中每个生物体的实验数据作为时间函数的比较。在每个亚图上,x轴和y轴分别代表每个物种的时间和相对丰度。数据点(红色)和线表示基于T1–T4的实验数据和模型预测。物种名称显示在每个子图的左上角。误差条表示六个生物重复的平均值的1 s.d。
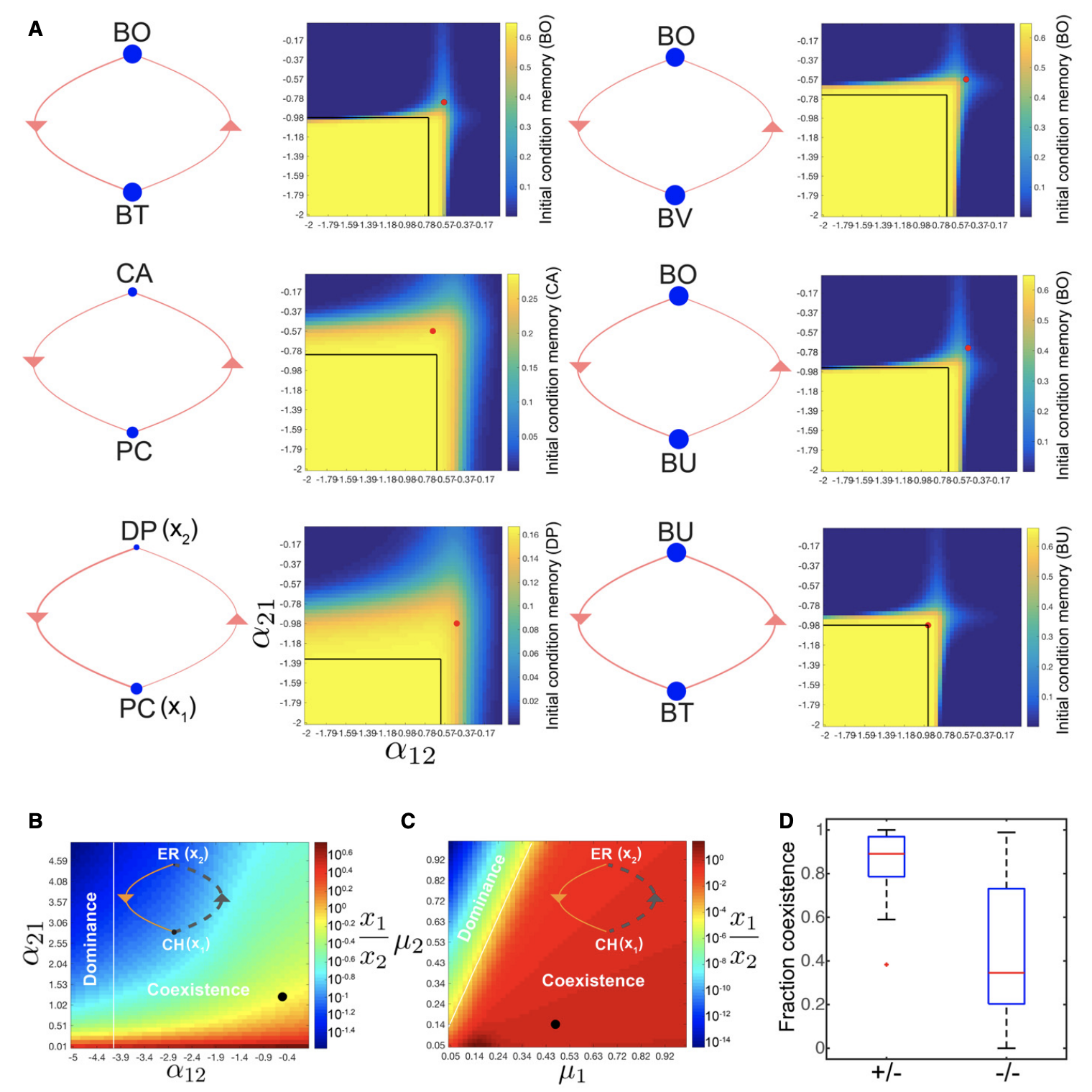

图4 成对群落基序中的历史依赖性和稳健物种共存
A 由于缓慢弛豫到平衡的历史依赖性可以通过负的物种间相互作用来增强。对六个成对群落的模型分析,这些群落在实验上表现出历史依赖性行为(附录图S5B),并在T3上训练的gLV模型中通过相互抑制相互作用进行耦合。网络拓扑结构(左)和一系列物种间相互作用系数值的历史相关响应热图(右)。网络图的线宽和节点大小分别表示物种间相互作用系数和稳态单物种丰度的大小。热图显示了使用两个初始条件模拟的群落在72小时时物种绝对丰度差异的绝对值:x1=0.0158,x2=0.0008或x1=0.0008,x2=0.0158图1B.1B.Thx1=0.0158,x2=0.0008或x1=0.0008,x2=0.0158使用图1B中的系列转移实验设计.黑框表示模型中双稳态的参数范围。圆圈(红色)指示基于训练集T3推断出的参数。
B 正和负相互作用的耦合可以对模型参数的变化表现出稳健的物种共存。网络图(插图)表示CH(X1)和ER(X2)之间推断的物种间相互作用的大小、符号和方向。虚线(灰色)和实线(橙色)分别表示正的和负的相互作用。线宽和节点大小分别表示单物种的物种间相互作用系数和稳态丰度的大小。x 1(CH)与x 2(ER)之比在72小时时作为物种间相互作用系数α12和α21的函数的热图。模拟的初始条件是x 1o=0.0008,x 2o=0.0158。圆圈(黑色)表示在T3上训练的gLV模型的CH、ER联合体的推断参数值。
C 在广泛的生长速率参数值范围内(μ1和μ2),72小时时x 1与x 2比率的热图(右)。模拟的初始条件是x 1o=0.0008,x 2o=0.0158。该线(白色)概述了稳定状态下共存和单物种优势的参数制度。圆圈(黑色)表示CH、ER联合体的推断参数值。
D 在T3上训练的gLV模型的所有推断的正/负(+/−)或双向负(−/-)网络的一系列模拟生长速率参数中,参数空间的部分呈现物种共存的箱形图。评估了2500个生长速率参数组合,范围从0.05到1 h−1。在每个框上,红线表示中值,框的边缘是第25和第75个百分位数,胡须延伸到算法认为不是异常值的最极端数据点,异常值绘制为红十字。

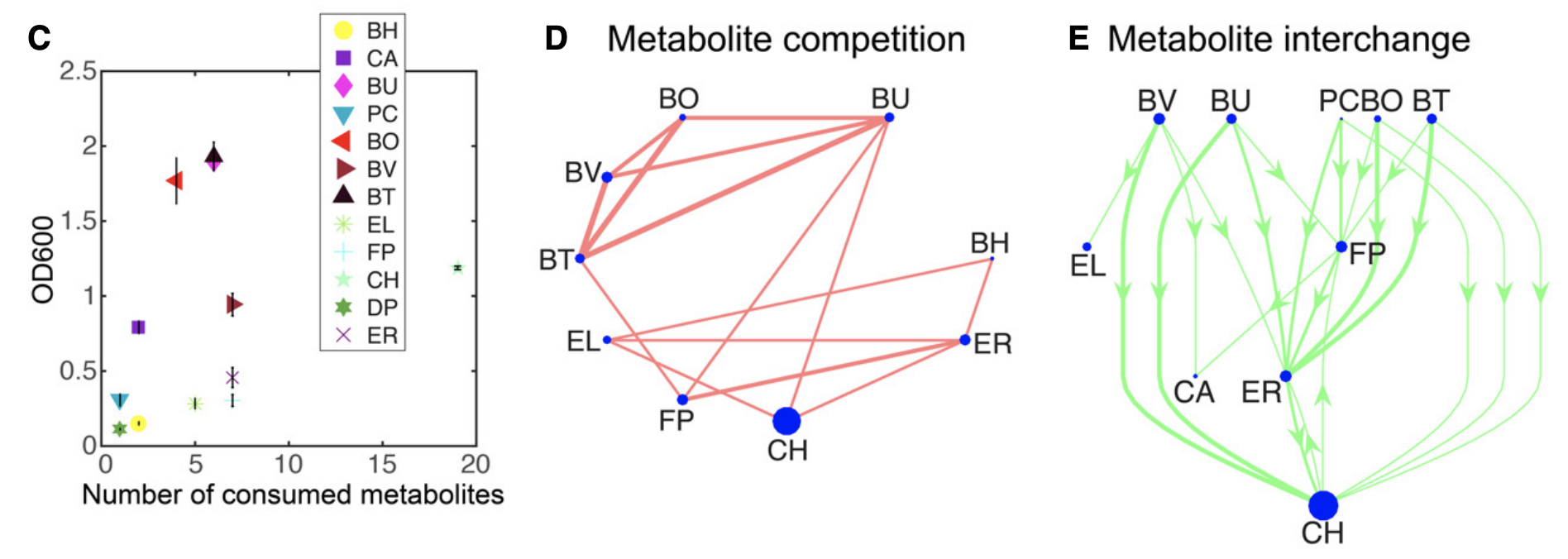

图5 主要代谢物的外代谢组学分析阐明了单物种的代谢能力
A 与实验开始时每种代谢物的丰度相比,减少了至少两倍的代谢物的物种(左)和代谢物(右)的二分网络。颜色表示网络中包含许多重叠交互的模块。使用BiMat将网络划分为模块(Flores等人,2015)。黑体代谢产物被耗尽,并由不同的生物体分泌。
B 与实验开始时相比,丰度增加了至少两倍的代谢物的物种(左)和代谢物(右)的二分网络。粗体突出显示的代谢产物被不同物种耗尽或分泌。颜色表示网络中包含许多重叠交互的模块。使用BiMat将网络划分为模块(Flores等人,2015)。
C 与实验开始相比减少了至少两倍的消耗代谢物数量与相应时间点单种培养物OD600值的散点图。误差条表示三个生物重复的平均值的1 s.d。
D 预测的资源利用互动网络。每个边缘代表至少两种共同消耗的代谢物,其减少了至少两倍,并且边缘宽度与共同消耗的代谢产物的数量成比例。节点大小与每个物种消耗的代谢物总数成比例。
E 基于双重阈值的预测代谢物交换网络,代表不同生物体分泌或利用的代谢物。箭头从来源物种指向消费生物。节点大小和线宽分别与分泌的代谢物总数和预测的代谢物相互作用的数量成比例。网络顶部和底部的物种分别主要是生产者或消费者。
https://wap.sciencenet.cn/blog-2675068-1415656.html
上一篇:2012 CB 竞争而非合作主导可培养微生物物种之间的相互作用
下一篇:2013 ARG 微生物群落社会互作的基因型观点