

2024年4月19日-21日,第一届Green Carbon青年编委会生物方向研讨会暨“合成生物技术与绿色生物制造——高版本底盘工程”学术沙龙在青岛召开。山东大学王海龙教授应邀出席并作题为“微生物基因编辑技术与细菌底盘构建”的邀请报告。
微生物基因编辑技术与细菌底盘构建
背景介绍
随着测序技术不断进步,微生物基因组数据成指数型爆发增长,其中蕴含着众多生物合成资源,亟待有效开发。基因编辑是基因功能解析和应用的重要手段,然而目前仍有很多基因由于长度过大和序列独特而难以克隆和改造。本次邀请报告中,王海龙教授系统介绍了其团队在开发微生物基因编辑技术和利用该技术进行细菌底盘构建方面的研究进展。王海龙教授团队开发了靶向抓取大片段复杂DNA的ExoCET技术和对复杂序列进行无痕定点改造的RedEx基因编辑技术,并应用上述技术构建了生产高值化合物的微生物底盘。这些技术为微生物基因资源开发和应用研究提供了强有力的工具,未来有望在多个合成生物技术领域发挥重要作用。

王海龙教授在“合成生物技术与绿色生物制造——高版本底盘工程”学术沙龙上作邀请报告
报告内容
大片段DNA直接克隆技术:ExoCET直接克隆技术实现>100kb DNA的一步靶向抓取
随着基因组测序技术的发展,研究人员在微生物基因组上发现了许多具有高研究和应用价值的生物合成途径。然而,由于很多微生物的遗传操作十分困难,在易于操作的宿主中进行异源表达成为研究这些生物合成途径的有效方法。面对编码基因(簇)庞大且序列复杂的生物合成途径时,经典的DNA克隆和修饰方法耗时费力,难以达到理想效果。
工欲善其事,必先利其器。王海龙教授团队长期致力于DNA直接克隆技术的研发,早在1998年王海龙教授的导师张友明教授就开发出Red/ET技术。该技术不受DNA分子大小和限制性酶切位点的制约,可以从大肠杆菌染色体上将任意大小的基因组DNA片段克隆到载体上,是基因工程领域的重要里程碑。为了突破Red/ET技术对物种的限制,张友明教授团队开发了RecET直接克隆技术。通过利用噬菌体5’-3’核酸外切酶(RecE)和单链DNA退火蛋白(RecT),在大肠杆菌细胞内将经限制性酶切的基因组片段与末端设计有同源臂的线性载体实现同源重组。基于RecET重组酶建立的DNA直接克隆技术能从纯化的细菌基因组上将50 kb以内的DNA片段直接捕获到表达载体上。RecET直接克隆技术极大地便利了微生物生物合成途径的克隆和异源表达研究。
此前,RecET直接克隆技术难以克隆> 50 kb的DNA片段,其原因在于该技术要求克隆载体和目的DNA片段必须同时进入一个大肠杆菌细胞内并相遇才能发生重组。王海龙教授团队在传统RecET技术的基础上增加一步体外重组步骤,发展形成新的ExoCET直接克隆技术,提高了克隆载体和目的DNA片段的结合概率,成功实现了大于100 kb细菌基因组DNA的靶向抓取;同时,应用ExoCET技术在克隆十几kb长度的DNA片段时,其效率相比传统的RecET直接克隆技术提高了80倍。与传统的大片段DNA克隆技术相比,ExoCET技术不仅实现了> 100 kb超大DNA片段的一步靶向抓取,而且将克隆周期从传统的基因组文库随机抓取策略的3个月缩减到3天(图1)。
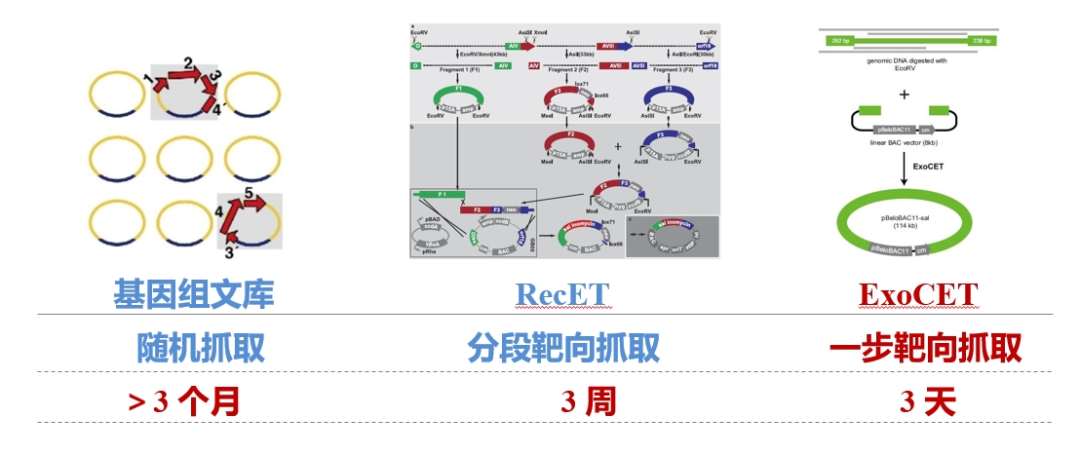
图1. 3种大片段DNA克隆技术
多操纵子人工基因簇技术:聚酮生物农药多杀菌素基因簇重构及异源表达
应用ExoCET直接克隆技术,王海龙教授团队进行了聚酮生物农药多杀菌素基因簇重构及异源表达研究。多杀菌素是一种广谱、低毒的绿色农作物杀虫剂,具有很高的应用价值;但其天然生产宿主刺糖多孢菌遗传操作难度大,且合成多杀菌素的基因簇长达80 kb,包含23个基因,采用传统方法难以成功克隆。应用ExoCET直接克隆技术,研究团队成功实现了对长达80 kb的多杀菌素基因簇的一步靶向抓取,并将其导入链霉菌底盘中成功实现异源表达。ExoCET直接克隆技术不仅能从基因组上抓取大片段,还能实现线性DNA片段的高效拼接。基于此特点,王海龙教授团队应用该技术对多杀菌素合成基因簇进行了表达优化。首先将基因簇上全部23个基因按照功能分成5个调控模块,进而应用ExoCET技术替换所有基因的启动子、核糖体结合位点和起始密码子,构建了多杀菌素的多操纵子人工基因簇,将多杀菌素在链霉菌底盘中的产量提高了328倍,实现了从微克水平到毫克水平的巨大提升(图2)。
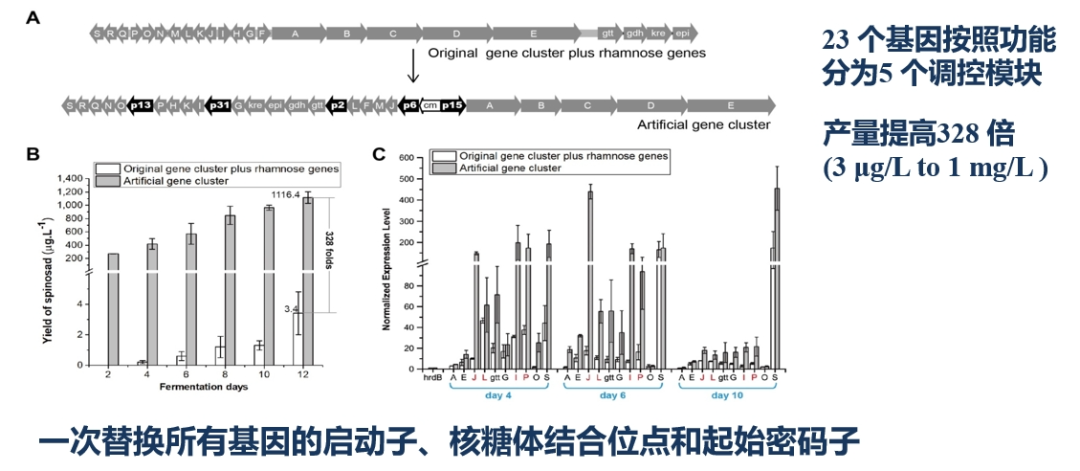
图2. 聚酮生物农药多杀菌素基因簇重构及异源表达
对表达多杀菌素合成人工基因簇的工程菌株发酵产物进行色谱分析后,研究团队发现产物中存在大量甲基化不完全的多杀菌素低活性副产物,占总量的50%左右。通过对多杀菌素合成通路的基因进行分析推测,该现象可能是由福乐糖胺转移酶和甲基转移酶的表达比例失衡造成的。针对这一问题,研究团队对多杀菌素的人工合成基因簇进一步优化,将副产物含量降低90%以上,而多杀菌素的产量则提高了5倍(图3)。
上述案例证明了ExoCET直接克隆技术在大片段DNA靶向抓取和多片段大型人工合成基因簇重构的高效性,展现出巨大的合成生物技术应用潜力。
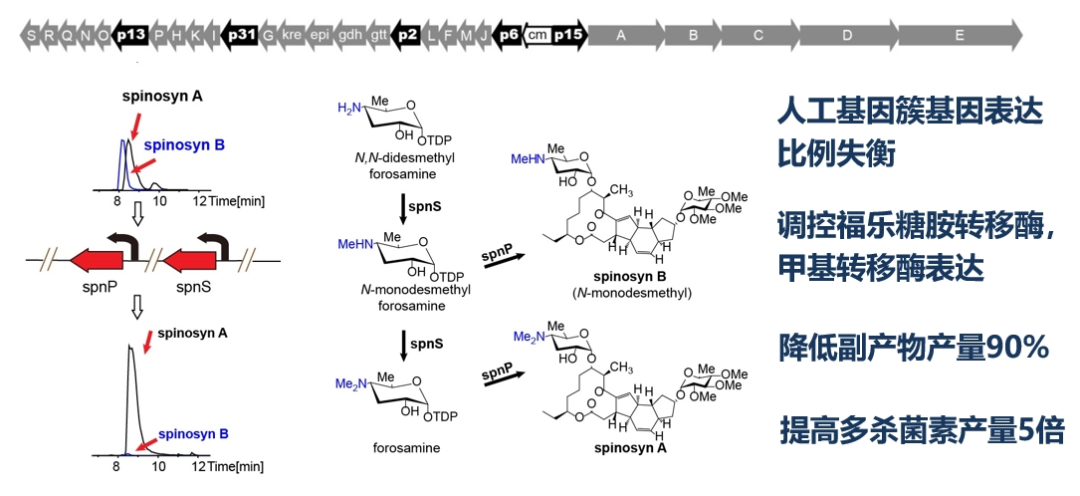
图3. 多杀菌素的人工合成基因簇优化改造
RedEx无痕定点突变技术:聚酮合酶结构域改造与化合物结构衍生
实现多杀菌素的异源高效合成后,王海龙教授团队进一步展开了多杀菌素衍生化合物丁烯基多杀菌素的合成研究。丁烯基多杀菌素杀虫活性更高、杀虫谱更广;其与多杀菌素合成的基因簇相似度极高,唯一的差别就是丁烯基多杀菌素基因簇中第一个聚酮合酶多了5个结构域,导致最终合成的产物多了两个碳原子,经成环后形成丁烯基多杀菌素。如果将这5个结构域添加到多杀菌素基因簇的第一个聚酮合酶中,就有可能实现丁烯基多杀菌素的合成;然而,聚酮合酶中功能相同的结构域导致其编码基因中存在着大量的重复序列,导致其无痕定点改造极其困难。
针对上述问题,王海龙教授团队开发了不受重复序列限制的RedEx无痕定点突变技术,成功对多杀菌素聚酮合酶基因进行异源结构域插入,从而实现丁烯基多杀菌素合成(图4)。在此基础上,为进一步提高丁烯基多杀菌素产量,团队利用RedEx无痕定点突变技术对基因簇中其他基因表达进行优化后,丁烯基多杀菌素总产量可提高至出发菌株水平的40倍。
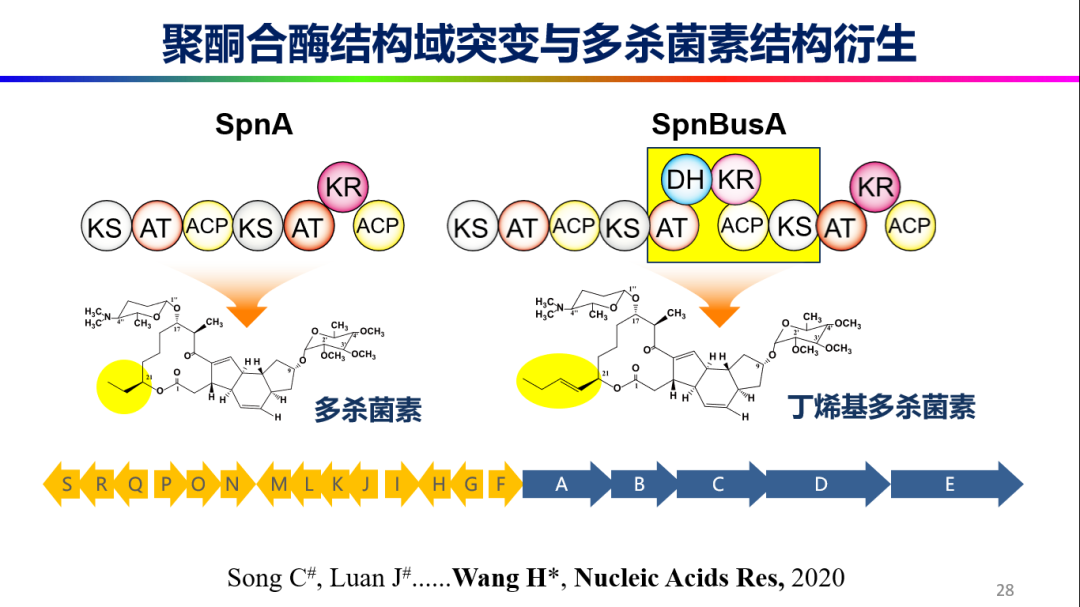
图4. 利用RedEx技术改造聚酮合酶结构域进行多杀菌素结构衍生
RedEx无痕定点突变的技术内核包括由Redαβ蛋白介导的体内同源重组、由CcdB毒蛋白介导的反向筛选、由核酸外切酶介导的体外同源重组。基于上述过程,可以高效地在大型复杂基因簇中进行点突变和结构域/模块替换等编辑操作。王海龙教授介绍了利用该技术实现聚酮合酶结构域改造与化合物结构衍生的典型案例,该技术有望在天然药物的合成生物学人工优化方面发挥重要作用。
专家介绍

王海龙 教授
王海龙,教授,博士生导师,Green Carbon期刊青年编委。现任山东大学微生物技术国家重点实验室副主任、微生物技术研究院副院长;入选山东省泰山学者(青年专家)(期满考核优秀)、山东大学杰出中青年学者(第一层次);获山东省青年科技奖、教育部自然科学二等奖(第二位)、山东大学优秀博士学位论文指导奖;主持国家优秀青年科学基金、山东省自然科学杰出青年基金、国家重点研发计划子课题、山东省自然科学基金重大基础研究等项目。主要从事微生物基因编辑与药物合成生物学研究,以第一或通讯作者在Nature Protocols、Nucleic Acids Research、Trends in Biotechnology、Journal of Advanced Research、Microbial Cell Factories、Analytical Chemistry、ACS Synthetic Biology等期刊发表论文18篇,授权欧洲发明专利1项,中国发明专利4项。
原文链接
王海龙教授与Green Carbon丨微生物基因编辑技术与细菌底盘构建
Green Carbon
期刊官网:Green Carbon官网
投稿网址:Green Carbon投稿
公众号:Green Carbon公众号

转载本文请联系原作者获取授权,同时请注明本文来自GreenCarbon科学网博客。
链接地址:https://wap.sciencenet.cn/blog-3620330-1496857.html?mobile=1
收藏

