过去二十年间,人类基因组研究虽取得显著进展,但其数据分布呈现出明显的区域性失衡。现有研究中超过80%的遗传数据集中于欧美人群及其他人口基数较大的洲际族群。相比之下,撒哈拉以南非洲、拉丁美洲、南亚、东南亚山地及太平洋岛国等地理隔离族群的基因组多样性研究及其群体精细遗传演化历史的解析仍显不足。这种研究偏倚不仅限制了我们对人类遗传多样性的系统认知,还导致了临床精准诊疗和疾病风险预测等领域显著的健康公平差距。
近日,广东省毒品实验技术中心刘超院士研究团队、四川大学考古科学中心/四川大学华西罕见病研究院何光林研究团队和重庆医科大学唐任宽研究团队联合在hLife发表了题为“Mighty oaks from little acorns: High-quality genomes of underrepresented populations enhance health equity in precision medicine”的文章(图1),从参考基因组序列完整性和群体遗传多样性两个维度出发,系统综述了测序技术的进步、参考基因组构建策略优化、大规模人群队列研究等在解析基因组复杂区域、鉴定人群特异性变异、重构人类群体演化历史等方面的重要进展。并进一步提出了若干未来研究方向,包括构建多元化族群参考基因组、完善社区参与式研究范式与数据主权保障体系,以及开发跨族群基因组资源整合分析工具链。

图1 论文标题及作者信息
以长读长测序、端粒到端粒(Telomere-to-telomere, T2T)组装、图形化参考基因组比对及高精度全基因组多类型遗传变异的精准分型为代表的技术革新,显著提升了人类基因组的解析深度(图2B)。然而,传统线性参考基因组在解析复杂结构变异方面存在固有局限。早期GRCh38版本中约8%复杂区域尚未被解析,这阻碍了对基因组结构与功能关联的系统研究。T2T组装技术的突破不仅实现了基因组序列的完整覆盖,还通过整合多维表观遗传信息,拓展了功能注释的维度。然而,基于单一遗传背景构建的参考基因组难以全面反映全球人群间的遗传异质性。国际人类泛基因组计划、H3Africa联盟及GenomeAsia 100K等项目采用长、短读长混合测序策略,在代表性不足的群体中发现了大量特异性遗传变异。例如,东南亚高地族群的代谢适应相关基因及非洲狩猎采集人群的免疫调控基因等(图2B)。
针对澳大利亚原住民、巴布亚高地人群、美洲土著及太平洋岛民等地理隔离群体的基因组研究表明,其独特的遗传多样性模式既受到复杂群体演化事件(如起源、迁徙及混合)的影响,也与古人类基因渐渗模式及环境适应性演化密切相关。这些发现不仅修正了传统人类演化模型,还揭示了跨大陆迁徙过程中基因流动的复杂动力学机制。值得注意的是,当前国际基因组计划对有效/普查群体规模偏小且地理隔离群体(如新几内亚高地部落、安达曼原住民)的系统研究仍存在显著缺口。这导致疾病风险预测模型存在族群特异性偏差,具体表现为群体特异性性状的遗传调控基础不明、罕见变异功能注释的不确定性以及药物代谢个体差异预测的局限性。这一困境的根源在于缺乏多维协同优化的研究策略,具体包括:(1)社区参与式研究框架的不足(如H3Africa的社区共研模式);(2)数据主权管理体系与共享机制的不完善,难以保障中、低收入国家(Low- and middle-income countries, LMICs)对其本土遗传资源的主导权;(3)基于泛基因组及多中心数据整合的分析算法的缺乏,无法消除当前以欧裔数据为中心的计算偏差。已有研究证实,提升基因组数据的多样性可显著增强临床转化的普适性。
未来研究应致力于构建更具包容性的全球基因组学研究体系(图2C)。在交叉学科国际合作框架下,需开展高质量的人类基因组研究,具体措施包括:(1)通过能力建设提升LMICs的本土研究水平,采用文化重视且社区参与的研究方法建立与土著群体的长效合作机制;(2)加强基础设施投入,支持LMICs开展自主基因组测序与生物信息学分析;(3)针对现有T2T/泛基因组分析工具的族群偏差,研发适用于多元遗传背景的人类参考基因组,并开发适用于科研和临床的计算方法。
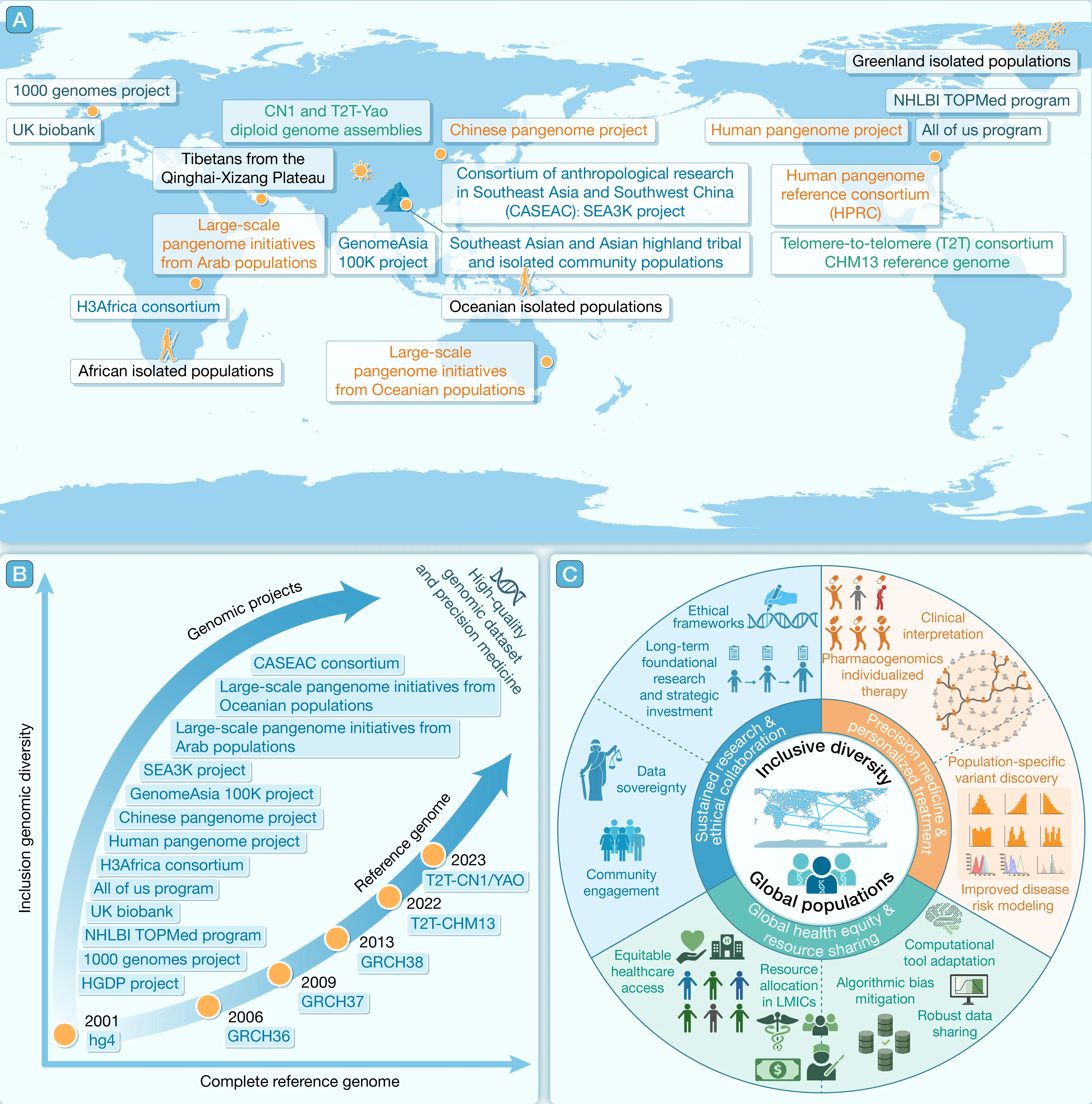
图2 全球基因组研究现状与未来方向
综上所述,当前大人群基因组学队列研究和群体遗传学研究的地域不平衡性制约了人类遗传多样性图谱的完整性。尽管T2T组装等新技术提升了参考基因组的质量,但基于单一遗传背景的参考基因组体系仍无法满足全球精准医学的需求。因此,亟需通过构建族群匹配的T2T泛基因组、完善伦理治理框架及开发跨族群分析工具等举措,推动基因组医学的公平发展。
作者简介
王萌鸽 副教授
第一作者
机构:重庆医科大学基础医学院法医系
研究方向:法医遗传学、古基因组学和群体演化遗传学相关研究
罗林焘 硕士研究生
第一作者
机构:重庆医科大学基础医学院法医系
研究方向:法医遗传学及群体演化遗传学相关研究
何光林 副研究员
第一作者
通讯作者
机构:四川大学考古科学中心/四川大学华西医院罕见病研究院
研究方向:古基因组学、法医基因组学与群体演化遗传学研究
唐任宽 教授
通讯作者
机构:重庆医科大学基础医学院法医系
研究方向:法医遗传学、法医病理学及法医临床学相关研究
刘超 中国工程院院士
通讯作者
机构:广东省毒品实验技术中心
研究方向:法医遗传学、法医病理学、法医微生物学及法医毒理相关研究
引用格式:Wang M, Luo L, Yeh H-Y, et al. Mighty oaks from little acorns: High-quality genomes of underrepresented populations enhance health equity in precision medicine. hLife 2025; https://doi.org/10.1016/j.hlife.2025.05.014.
期刊简介
hLife 由高福院士、董晨院士和Jules A. Hoffmann教授(2011诺奖获得者)领衔,是中国科学院微生物研究所主办,中国生物工程学会,浙江大学陈廷骅大健康学院,西湖大学医学院,上海市免疫治疗创新研究院和广州霍夫曼免疫研究所联合支持,与国际出版商爱思唯尔合作的健康科学领域综合性英文期刊。
hLife 聚焦健康科学领域的前沿进展,旨在促进基础研究与临床应用的融合发展。期刊发表与医学相关各研究领域最新成果,学科领域包括(但不限于)病原生物学、流行病学、生理学、免疫学、结构生物学、疾病监测、肿瘤、药物、疫苗和健康政策等。
hLife是一本金色开放获取期刊,月刊出版;2022年成功入选“中国科技期刊卓越行动计划高起点新刊”;2023年11月正式创刊;2024年5月被DOAJ收录;2024年8月被Scopus收录;2024年10月入选“首都科技期刊卓越行动计划——重点英文科技期刊支持项目”;2025年6月入选“2025年度支持高水平国际科技期刊建设-强刊提升”项目。
2026年前hLife接收的稿件免收文章处理费(APC)。
期刊网址:
https://www.sciencedirect.com/journal/hlife
转载本文请联系原作者获取授权,同时请注明本文来自闫群科学网博客。
链接地址:https://wap.sciencenet.cn/blog-3552961-1493026.html?mobile=1
收藏

