博文
Biology:环境DNA技术用于监测干旱区内陆河流底栖无脊椎动物的适用性
||
底栖无脊椎动物是水生态系统的重要组成部分,其群落结构和多样性可以反映河流生态状况。环境DNA (eDNA) 宏条形码技术由于具有非侵入性、省时以及成本低等优势,已被运用在监测底栖无脊椎动物多样性和水生态系统方面。在中国西北部新疆境内,乌伦古河是一条重要的干旱区内陆河流,其极端水文特征 (降雨量小,蒸发量大) 以及断流频发会导致形态学操作困难,现有的基于形态学的生物监测频率难以满足生态预警需求,因此迫切需要验证新的监测技术如eDNA宏条形码在此干旱区内陆河流的适用性。
来自塔里木大学生命科学与技术学院宋勇副教授、陈生熬教授团队在 Biology 期刊发表了相关的研究。在乌伦古河流域内6个位点采集了底栖无脊椎动物形态学和eDNA水样滤膜样本,使用Mantel.test和NMDS分析检验两种方法得到群落的相关性,计算Shannon指数评估生态状况,分析评估结果的相关性。
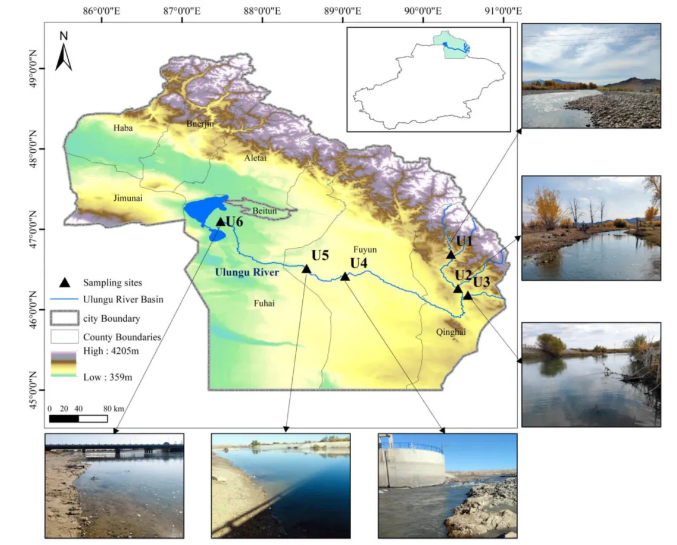
图1. 乌伦古河流域eDNA和形态学样本收集位点分布图。
研究发现,eDNA方法并未检测到比形态学更丰富的物种,但检测到更多体型小、传统形态学难以采集和识别的种类,包括扁形动物门,海绵动物门和刺胞动物门。eDNA方法在每个位点检测到的门级数量都高于形态学,且两种方法科级及以下分类水平共享分类单元数较少,在属级仅共享5个分类单元。对于EPT种类,eDNA元编码仅检测到蜉蝣目与毛翅目种类,并未检测出襀翅目。此外eDNA方法检测到的节肢动物物种丰富度低于形态学方法,而软体动物物种丰富度高于形态学,环节动物丰富度显著高于形态学 (Mann-Whitney U test:Arthropoda:Z = -1.615,p = 0.106;Annelida:Z = -2.569,p = 0.010;Mollusca:Z = -0.763,p = 0.445)。但两种方法监测到的物种丰富度差距最大的是节肢动物门。从相对丰度来看,eDNA整体上对环节动物门有更高的检测效率。
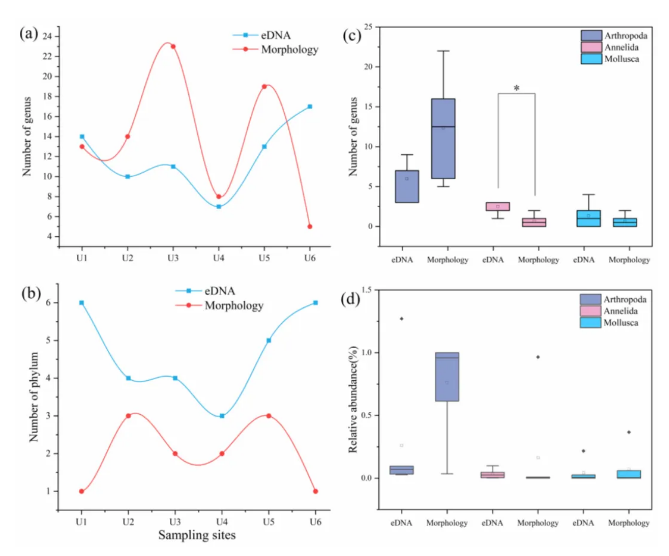
图2. 通过eDNA和形态学方法在属 (a) 和门水平 (b) 上检测到的每个位点的底栖无脊椎动物丰富度,以及两种方法检测到的共享门的属级分类单元数量 (c) 和相对丰度 (d)。
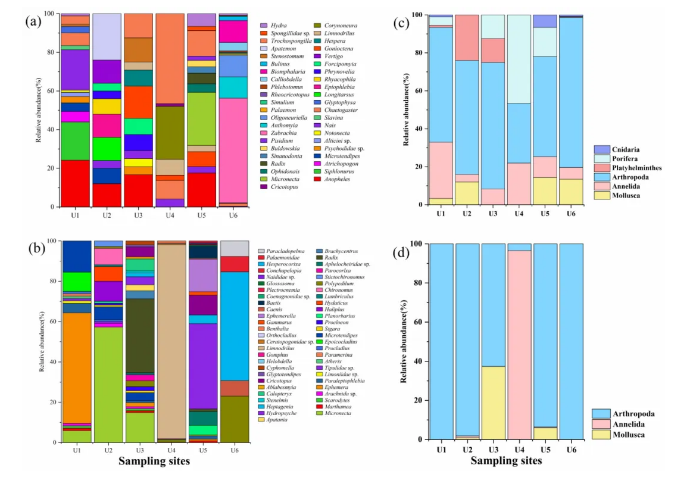
图3. 在各个位点,采用eDNA方法 (a、c) 或形态学方法 (b、d) 的属水平 (a、b) 和门水平 (c、d) 的底栖无脊椎动物的相对丰度 (%)。
经Mantel.test和NMDS分析发现两种方法监测到的群落之间具极显著差异 (PERMANOVA,p = 0.0056),表明使用两种方法对群落结构的监测一致性较低。在监测底栖无脊椎动物群落时,采用水样eDNA元编码方法作为一种独立方法进行监测存在不可靠性。
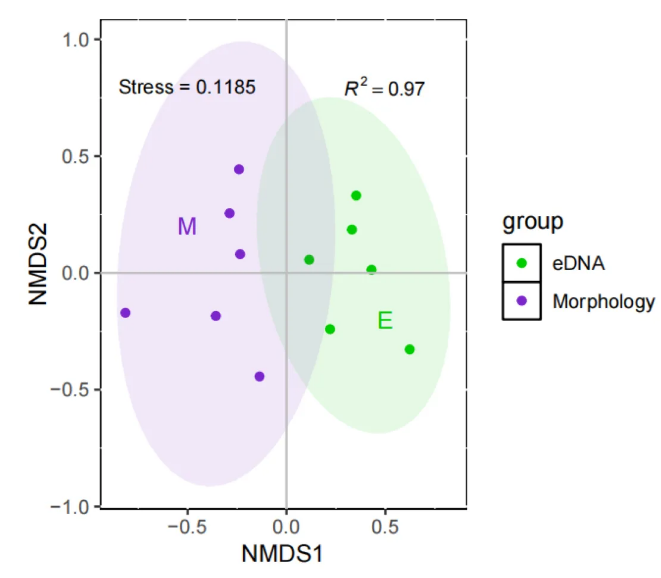
图4. eDNA和形态学方法监测到的底栖无脊椎动物不同群落的NMDS分析。
cor.test结果表明两种方法多样性及生态评估结果无显著相关性 (p = 0.055),综合对比发现,两种方法平均生态等级一致,然而具体到各位点上的比较,eDNA方法的生态评估结果整体上优于传统形态学。根据现场采样情况判断,eDNA方法的生态评估结果不能代表准确、真实的生态状况。因此,在干旱区河流中,eDNA生态评估需结合形态学进行验证,避免单一技术误判。
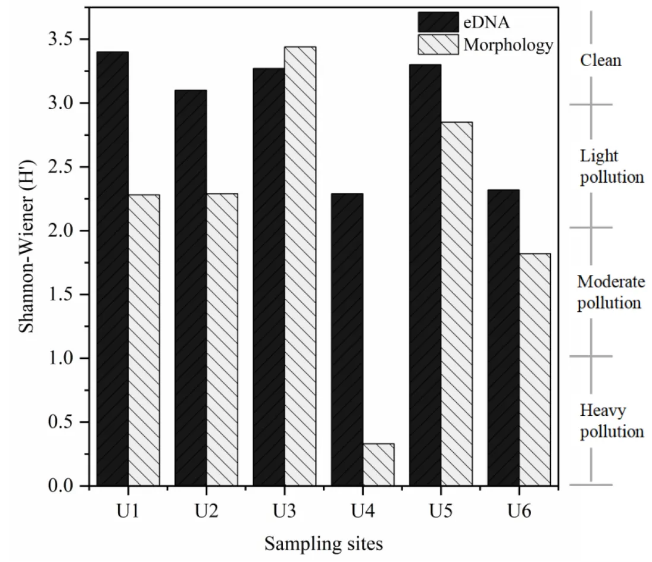
图5. eDNA和形态学方法在各个位点的Shannon指数及生态状况评估等级。
从水样获取eDNA的方法与形态学方法对干旱区河流底栖无脊椎动物群落和多样性的监测一致性低,但两者结合使用无疑能提供更高精度、更准确的生态监测结果。未来需针对eDNA采样的关键环节、降解率的控制、环境对eDNA结果的影响机制、数据的利用率及指数开发等方面进行更多的研究,以期为生态研究领域提供更环保有效的监测手段,为生态决策提供更可靠的支持,更充分的应对全球环境变化挑战。
原文出自 Biology 期刊:https://www.mdpi.com/3267004
期刊主页:https://www.mdpi.com/journal/biology
Biology 期刊介绍
主编:Jukka Finne, University of Helsinki, Finland;Andrés Moya, University of Valencia and CSIC, Spain
期刊主要涵盖细胞生物学、发育生物学、进化生物学、生物化学与分子生物学、微生物学等所有生物领域。期刊被Scopus, SCIE (Web of Science), PubMed等重要数据库收录。
2023 Impact Factor:3.6 (JCR Q1*)
2023 CiteScore:5.7 (Q1**)
Time to First Decision:16.4 Days
Acceptance to Publication:2.5 Days
* JCR Q1 at "Biology"
** CiteScore Q1 at "General Agricultural and Biological Sciences"

https://wap.sciencenet.cn/blog-3516770-1488453.html
上一篇:Applied Sciences:山东大学张同伟教授和浙江大学陈惠芳教授合作建立特刊——海洋声学前沿进展
下一篇:IJMS:2025年杰出青年学者奖提名火热进行中!