Prokka是一个适用于原核生物的基因组自动注释工具,由墨尔本大学生物信息学家 Torsten Seemann 开发。Prokka协调了一套现有的软件工具,可以对原核基因组和宏基因组进行快速高效的功能注释。
一、如何使用Prokka?
Prokka的使用要求:
1、准备基因组序列文件(FASTA格式);
2、安装 Prokka 及其依赖工具(如 BLAST+、HMMER)。
Prokka的运行命令:
prokka --outdir my_output --genus Escherichia --species coli genome.fasta
Prokka的输出结果:
Prokka 会在指定输出目录中生成多种格式的注释文件,包括 GFF、GenBank 和蛋白序列文件,方便后续分析或可视化。
本地安装和使用的具体操作方法可以参见发布的另一篇文章《Prokka安装和使用教程——快速原核基因组注释》
不过,需要注意的是,Prokka的运行环境依赖于Linux系统,这对于许多没有相关操作经验的用户来说,设置和使用门槛较高,常常成为科研工作中的一道“技术壁垒”。
二、零基础做生信
为了解决这一难题,我们推出了密码子·生信云平台 “Prokka原核基因组注释” 免费小工具!
零门槛体验:无需本地安装Linux环境,无需复杂配置,打开网页即可一键上传数据,轻松运行Prokka进行基因组注释分析。
高效安全:云端计算资源强大,数据处理速度快,保障数据安全与隐私。
友好界面:操作界面简洁直观,适合各类用户,无需编程基础。
专业支持:提供详细的操作指南和技术支持,助力您的科研工作顺利进行。
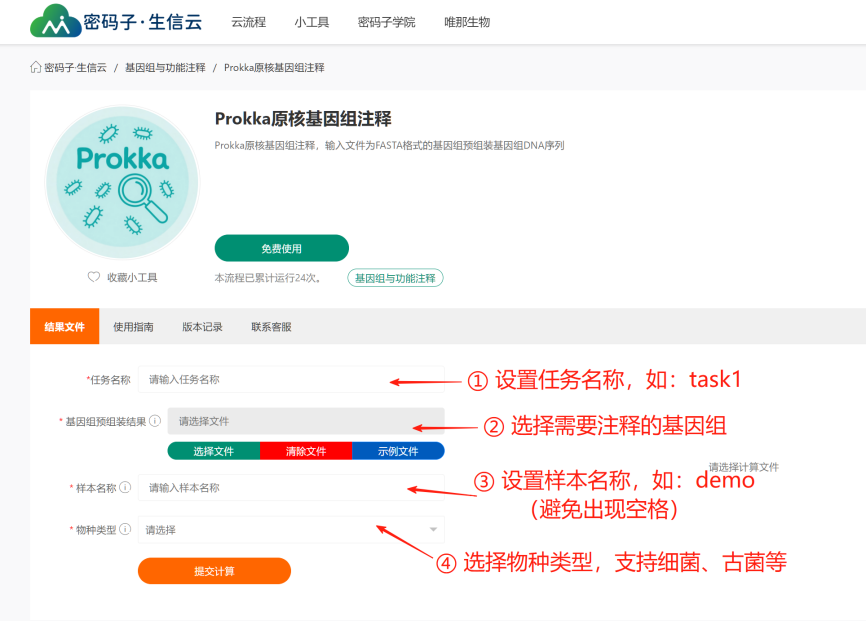
小工具链接:
https://cloud.mimazi.net/tool/article-117.html
输入文件:
基因组预组装结果/核酸序列文件

小工具结果:
*.faa:预测的所有编码序列(CDS)的蛋白质翻译产物序列文件(FASTA format of amino acids)
*.ffn: 预测的所有编码基因(CDS)的核苷酸序列文件(FASTA format of nucleotides)
*.fna:输入的基因组序列文件(FASTA format)
*.fsa:包含源序列的FASTA文件
*.gbk:GenBank 格式的文件。包含完整的注释信息以及原始基因组序列本身
*.gff:采用 GFF3 (General Feature Format version 3) 格式,包含基因组上所有预测的基因、RNA 特征(tRNA, rRNA, ncRNA等)、CDS(编码序列)以及它们的位置、功能注释(如基因名称、产物描述)、EC号(如果可预测)、注释来源等信息
*.sqn:提交到 GenBank 所需的 ASN.1 格式文件
*.tbl:Sequin 表格格式文件
*.tsv:表格格式(Tab-Separated Values)的注释摘要。包含特征(Feature)ID、类型、位置、长度、基因名称、基因座标签、产物描述、EC号等字段
*.txt:摘要统计文件,包含一些基本的统计信息
密码子生信云还提供云流程和其它各色小工具分析服务:
为细菌基因组研究量身定制的智能云流程从基因组测序原始数据(fastq)或组装序列(fasta)出发,一键化完成细菌基因组全套分析流程,包括基因预测、多功能注释和多数据库分析。多种多样的实用小工具,可以为您精准解决碎片化需求,包括序列处理、统计绘图、基因组注释等等,助力您轻松完成数据分析。

参考文献
Seemann T. Prokka: rapid prokaryotic genome annotation. Bioinformatics. 2014 Jul 15;30(14):2068-9. doi: 10.1093/bioinformatics/btu153 . Epub 2014 Mar 18. PMID: 24642063
转载本文请联系原作者获取授权,同时请注明本文来自牛祥娜科学网博客。
链接地址:https://wap.sciencenet.cn/blog-3447233-1499903.html?mobile=1
收藏

