BLAST(Basic Local Alignment Search Tool)是一款由 NCBI(美国国家生物技术信息中心)提供的生物信息学工具,用于比较基因或蛋白质序列。以下是如何设置比对结果输出为outfmt 6 时指定输出特定数据。
# 指定输出指定内容
# -outfmt "6 qseqid sseqid pident evalue bitscore qseq sseq"
# 指定输出格式为6,结果中从前到后分别为指定内容:qseqid(查询序列ID)、sseqid(目标序列ID)、pident(匹配百分比)、evalue(E值)、bitscore(比特分数)、qseq(查询序列)、sseq(目标序列)
blastn -query data/nucl_query.fasta -out data/outfmt6.tsv -db data/nucl.blastdb -num_threads 16 -evalue 1e-10 -max_target_seqs 5 -outfmt "6 qseqid sseqid pident evalue bitscore qseq sseq"
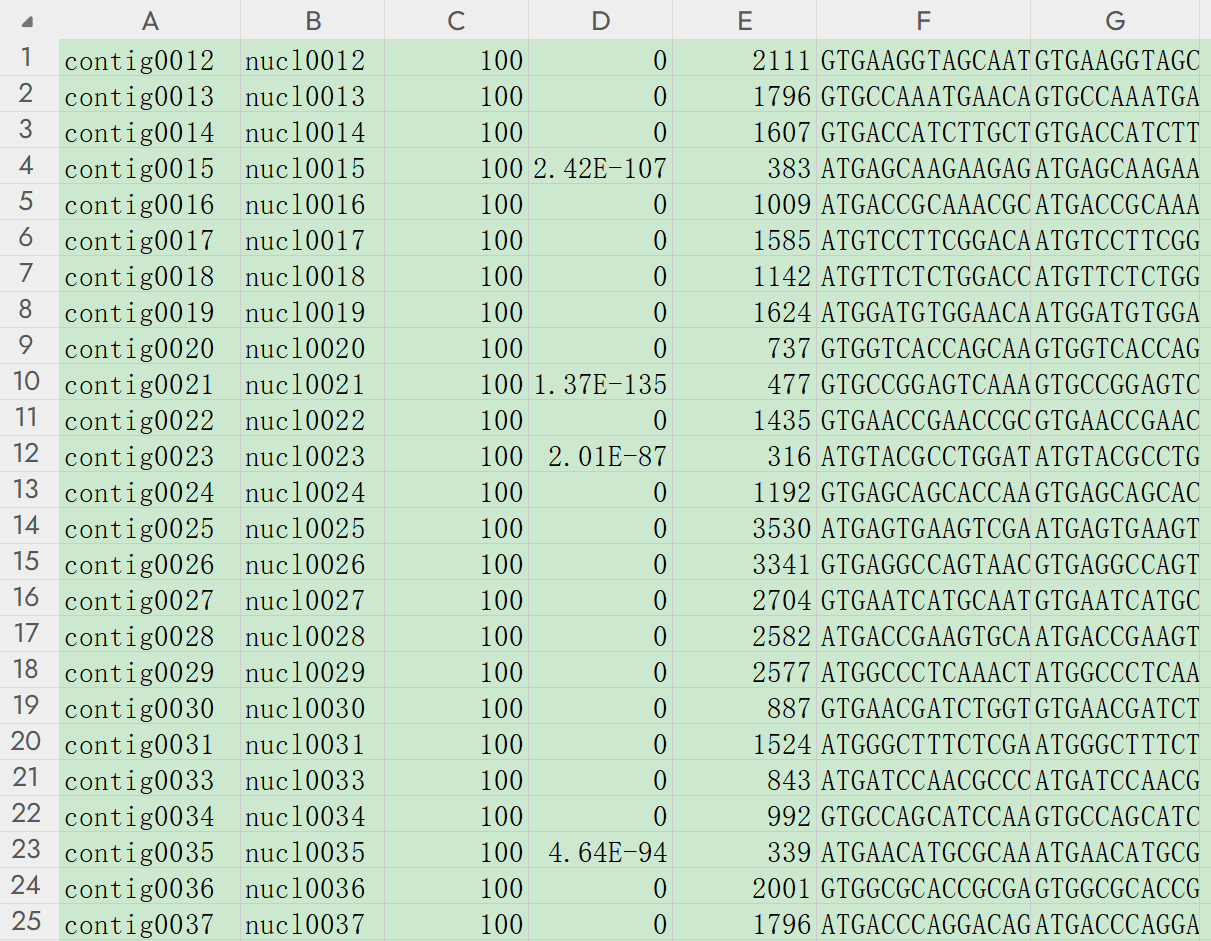
以上代码输出了输入及数据库中的序列名称、evalue以及两条序列的碱基序列等信息。
更多可指定内容请参考”https://www.ncbi.nlm.nih.gov/books/NBK279684/“的Table C1表格。
我们将持续分享微生物组学研究和生信分析相关的专业技能资料。推荐课程请搜索“密码子学院”。课程问题或个性化分析需求,请联系小唯(微信号:winnerbio01)。
转载本文请联系原作者获取授权,同时请注明本文来自牛祥娜科学网博客。
链接地址:https://wap.sciencenet.cn/blog-3447233-1489609.html?mobile=1
收藏

