博文
iTOL进化树外周条带个性化调整方法
|
引言
进化树外周条带注释的样式调整(如边线与色块)可显著提升数据可视化效果。本文通过解析文献案例(示例一至示例四),系统阐述不同注释样式的实现方法,并揭晓其设计逻辑。文中所有示例均可基于iTOL工具,通过调整模板参数或网页界面操作完成。
如何添加外周条带注释参见往期文章《iTOL的三种进化树注释方式》。
一、边线样式解析与调整方法
外周注释的边线分为两类:外周边线(环绕条带外缘)和样本间边线(分隔相邻样本)。以下为具体调整方法:
1. 外周边线样式调整
颜色与粗细控制:
iTOL网页操作:添加外周注释后,在“Datasets”界面选择“Border style”,可自定义边线颜色(条带1-3的白色、黑色或同色)及粗细。
模板调整:修改tol_color_strip模板中的BORDER_COLOR与BORDER_WIDTH参数,上传至iTOL即可生效。
2. 样本间边线添加
需在iTOL网页中将“Complete border”选项设为“Yes”(条带4-5)。
注意:iTOL模板仅支持外周边线调整,样本间边线需通过网页界面设置。
图示说明:
条带1-3展示了不同外周边线效果(白、黑、同色);条带4-5在1和3基础上添加了样本间边线。

图1 边线样式解析
二、色块样式解析与调整方法
色块用于区分数据类别(如基因型、表型),也可表示“有无”关系(如基因存在与否)。
1. 常规色块设置
iTOL网页操作:在“Datasets”界面的“Raw data”中直接编辑颜色代码,按分组赋予不同色值。
模板调整:通过tol_color_strip模板的“data”字段定义颜色,并上传至iTOL。
2. “有无”关系表示
统一底色法:在“Raw data”中将“absence”设为白色或灰色,或在模板中指定相同颜色代码(如#ffffff)(图1)。
同色系渐变法:在“Raw data”中使用主色的浅色调表示“absence”,或在模板中单独定义浅色色值(图2)。
图示说明:
图2条带1-5是在图1统一底色的基础上调整为同色系渐变的结果。

图2 色块样式解析
三、文献案例解析
以下为示例一至示例四的注释样式实现逻辑,可结合上述方法实现。
1. 示例一(doi:10.3390/ijms242116017)

设计:深色/浅色表示基因突变有无,启用样本间边线。
方法:使用tol_color_strip模板定义双色,网页端开启“Complete border”。
2. 示例二(doi:10.1016/j.scitotenv.2023.165203)
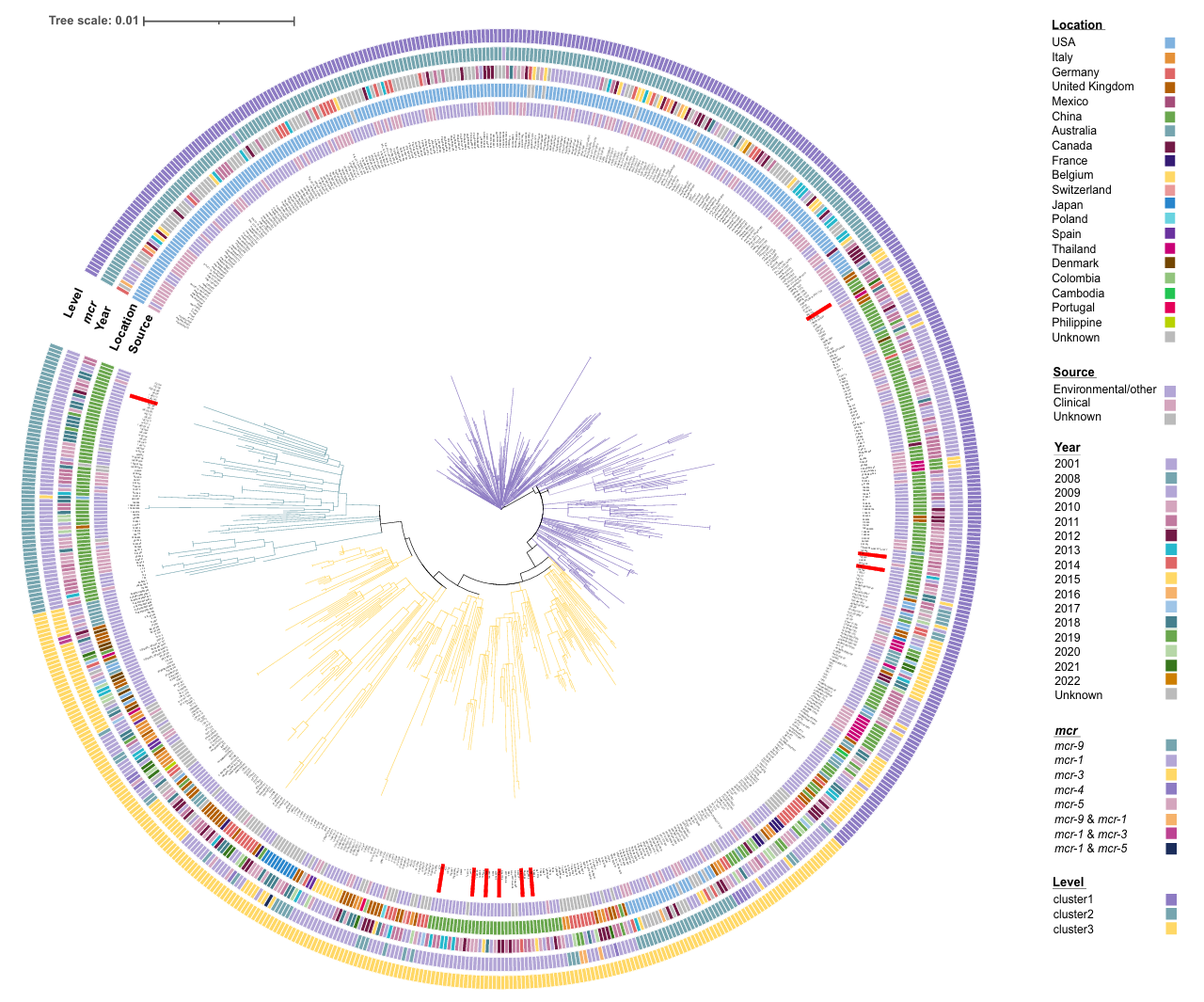
设计:多色区分样本信息、基因分类及cluster,添加样本间边线。
方法:通过tol_color_strip模板定义多组色块,网页端设置黑色外周边线。
3. 示例三(doi:10.1016/j.scitotenv.2023.165203)
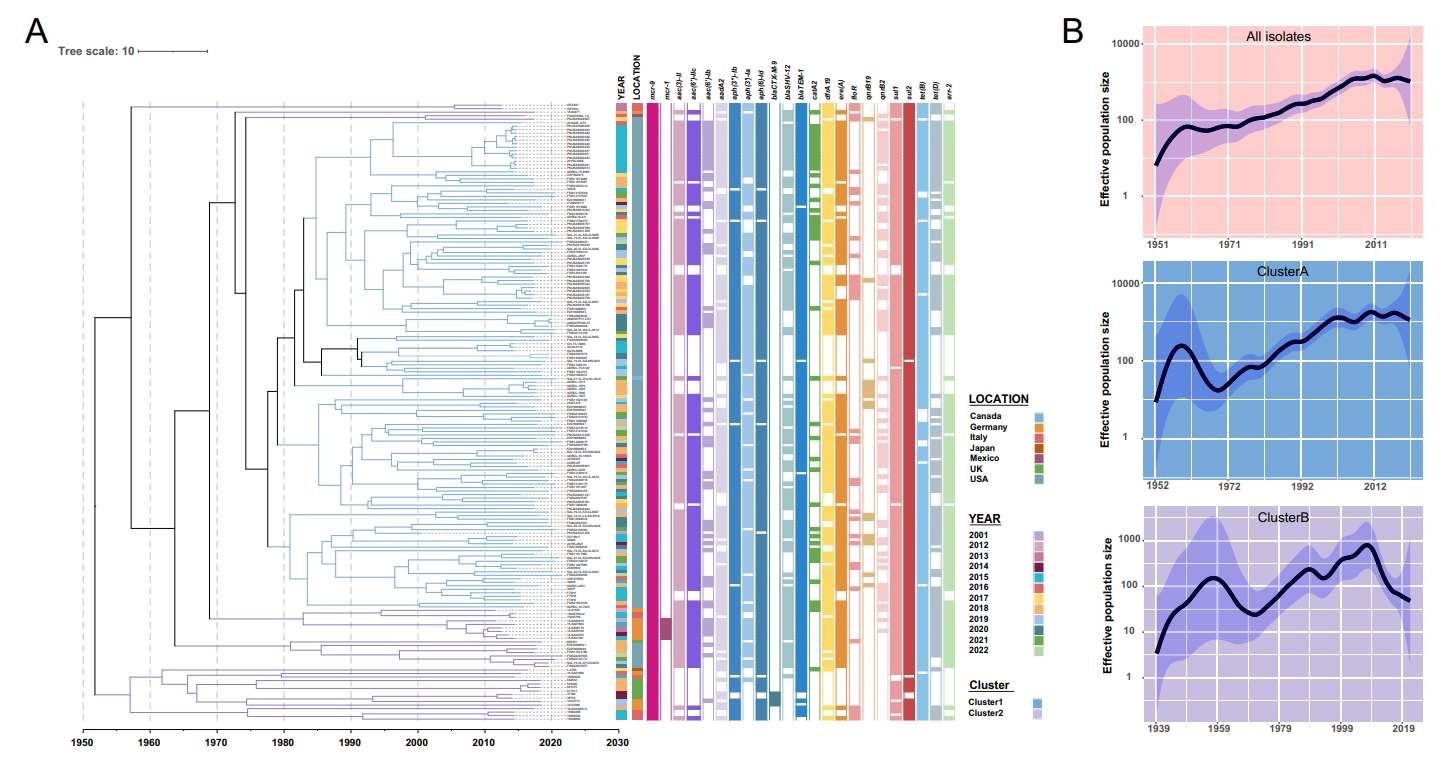
设计:彩色区分样本信息,白色表示基因缺失,采用彩色外周边线。
方法:tol_color_strip模板指定“absence”为白色,外周边线颜色与主色一致。
4. 示例四(doi:10.3389/fmicb.2023.1221428)
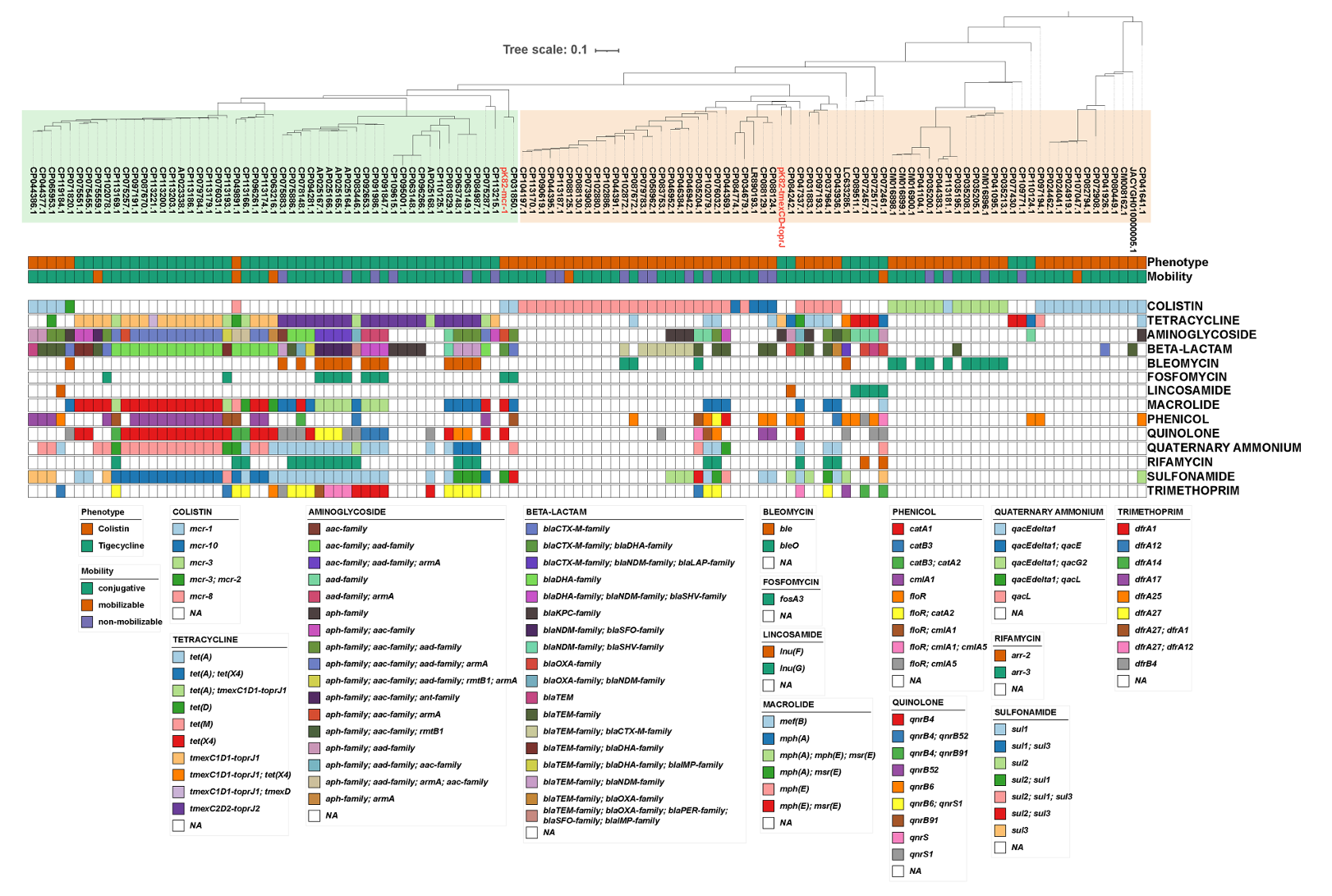
设计:彩色表示耐药基因分类,白色表示基因有无,采用黑色外周边线+样本间边线。
方法:tol_color_strip模板指定“absence”为白色,网页端启用黑色边线与样本分隔。
结语
通过灵活组合边线与色块参数,可高效实现进化树注释的多样化展示。建议优先使用iTOL网页界面进行实时调试,复杂需求则通过模板编辑批量处理。实际应用中需注意:外周边线颜色需与整体配色协调,而“有无”关系的表达应避免色彩过杂,以确保可视化结果的科学性与美观性。
更多内容请参考“密码子实验室”公众号。如有相关课程问题,请联系唯那生物技术客服小唯(微信号:winnerbio01)
https://wap.sciencenet.cn/blog-3447233-1483105.html
上一篇:微生物分型进化核心术语解析
下一篇:知识小卡片 | 如何理解菌群α多样性