微生物数据分析因其高维性、稀疏性和组合性而面临巨大挑战。最近的进展表明,整合丰度和系统发育信息是揭示稳健模式和提高微生物组研究预测性能的有效策略。然而,现有方法主要关注系统发育树的层次结构,忽略了其中嵌入的进化距离。
2024年10月15日,南昌大学徐振江通讯在Advanced Science 在线发表题为“DeepPhylo: Phylogeny-Aware Microbial Embeddings Enhanced Predictive Accuracy in Human Microbiome Data Analysis”的研究论文。该研究介绍了DeepPhylo,这是一种采用系统发育感知扩增子嵌入来有效整合丰度和系统发育信息的新方法。
DeepPhylo提高了微生物组数据分析的无监督判别能力和监督预测准确性。与现有方法相比,DeepPhylo在五个现实世界微生物组用例中表现出提供生物学相关见解的优势,包括皮肤微生物组的聚类、宿主年龄和性别的预测、15项研究中的炎症性肠病(IBD)诊断以及多标签疾病分类。

人类微生物群由细菌、古细菌、噬菌体和病毒组成,形成一个复杂的生态系统,存在于人体的各个部位。值得注意的是,微生物群的变化与宿主的生理状态有关,导致多发性硬化症、II型糖尿病和IBD等疾病。这些变化还受到年龄、生活环境和饮食等因素的影响。
16SrRNA基因靶向测序的进步通过促进细菌和古细菌的经济高效鉴定,彻底改变了微生物学。该技术专注于对核糖体RNA基因的可变区(V1-V9)进行测序,随后将其加工成(OTU)或扩增子序列变体(ASV)。由此产生的微生物丰度表以及描述分类群之间关系的系统发育树为下游分析提供了丰富的数据集。
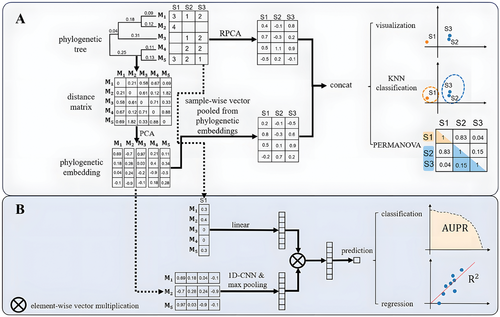
DeepPhylo的整体框架。(图源自Advanced Science )
鉴于微生物组与人体生理学之间的密切联系,使用微生物组数据进行预测建模已成为探索宿主表型和疾病诊断的关键领域。这些预测方法通常根据其对样本标签的使用情况分为无监督或监督学习算法。此外,根据它们是否整合了系统发育信息,它们可以区分为系统发育感知方法或系统发育不可知方法。在这种情况下,机器学习方法面临着数据高维性和稀疏性等挑战。通常使用Lasso和随机森林(RF)等技术,这些技术对于处理稀疏数据特别有效。最近,深度学习在捕捉微生物组数据中的复杂模式方面显示出巨大的潜力。
最近的研究试图整合丰度和系统发育信息,以便进行更有见地的微生物组数据分析。例如,PhyloRPCA将样本丰度数据从叶节点扩展到系统发育树的内部节点,以进行无监督数据分析。MDeep根据系统发育关系排列丰度数据,并采用一维卷积神经网络(CNN)来捕获不同OTU之间的系统发育信息。通过将一维丰度数据转换为二维矩阵,PopPhy-CNN利用二维CNN来捕获系统发育树各个分支之间的系统发育信息。此外,Ph-CNN通过在卷积过程中选择k个最近的OTU来增强传统的1D-CNN。然而,这些方法仅关注系统发育树的层次结构,而忽略了其中的定量距离信息,从而限制了它们全面捕获系统发育全貌的能力。
在本研究中,作者介绍了DeepPhylo,这是一种新颖的机器学习方法,旨在通过结合系统发育和丰度信息来增强无监督学习中的样本聚类并提高监督深度学习模型的预测准确性。DeepPhylo通过提取每个OTU的嵌入并将其与相应的丰度数据集成来实现这一点。此外,通过将OTU嵌入合并到神经网络中,DeepPhylo显著提高了监督深度学习模型在各种任务中的预测性能,包括回归、二元分类和多标签分类。DeepPhylo的贡献有两方面:
1)它通过合并从主成分分析(PCA)技术中得出的系统发育嵌入来增强无监督学习,从而提高样本的判别能力;
2)它引入了一种新颖的神经网络架构,可有效利用OTU嵌入来提高使用微生物组数据进行宿主生理学预测的性能。
文章链接:
https://onlinelibrary.wiley.com/doi/10.1002/advs.202404277
转载本文请联系原作者获取授权,同时请注明本文来自Wiley开放科研科学网博客。
链接地址:https://wap.sciencenet.cn/blog-3411312-1463718.html?mobile=1
收藏

