数据可以存储在塑料中吗?
诸平
Fig. 2 Credit: Angewandte Chemie, © Wiley-VCH
据威利公司(Wiley)2024年11月10日提供的消息,数据可以存储在塑料中吗?韩国首尔国立大学(Seoul National University, Seoul, Korea)的研究人员对此进行了回答,并给出其工作原理(Could Data Be Stored in Plastic? Here’s How It Works)。
一项新技术可以将数据存储在合成聚合物中,无需全序列解码即可直接访问比特,从而显著提高了存储密度和稳定性,该技术通过在聚合物中以ASCII编码大学地址来证明。
对数据存储的需求正在增长,许多类型的数据需要长期保存。合成聚合物是传统存储介质的有效替代品,因为它们可以使用更少的空间和能源存储信息。然而,传统的检索方法,如质谱法,限制了单个聚合物链的长度和存储容量。现在,据德国《应用化学国际版》(Angewandte Chemie International Edition)2024年8月30日报道——Heejeong Jang, Hyunseon Chu, Hyojoo Noh, Kyoung Taek Kim.Shotgun Sequencing of 512-mer Copolyester Allows Random Access to Stored Information. Angewandte Chemie International Edition, 30 August 2024: e202415124. DOI: 10.1002/anie.202415124.韩国首尔大学化学系(Department of Chemistry, Seoul National University, Seoul, Rep. Korea)研究人员已经开发出一种克服这一限制的新方法,可以直接访问特定的数据位,而无需读取整个链。
聚合物存储相对于DNA的优势(Advantages of Polymer Storage Over DNA)
由业务事务、流程监控、质量保证和产品跟踪而生成的数据在不断积累。将如此庞大的数据存档数十年需要大量的空间和能源。对于这种大型、很少访问的数据集的长期存储,具有定义序列的大分子,如DNA和合成聚合物,提供了一个令人信服的解决方案。
技术挑战和创新解决方案(Technical Challenges and Innovative Solutions)
合成聚合物比DNA有很多优点:合成简单,储存密度高,在恶劣条件下也很稳定。它们的缺点是编码在聚合物中的信息需要通过质谱(mass spectrometry简称MS)或串联质谱测序(tandem-mass sequencing简称MS2)来解码。
对于这些方法,必须限制分子的大小,这严重限制了每个聚合物链的存储容量。此外,完整的链必须按顺序解码,逐块构建,感兴趣的位不能直接访问。
这就像必须通读整本书,而不是打开到相关的一页。相比之下,长链DNA可以被切割成随机长度的片段,单独测序,然后通过计算重建成原始序列。
数据编码与检索的突破(Breakthrough in Data Encoding and Retrieval)
韩国首尔国立大学化学系的Kim Kyoung Taek和他的团队开发了一种新方法,可以有效地解码分子量大大超过MS和MS2分析极限的超长合成聚合物链。
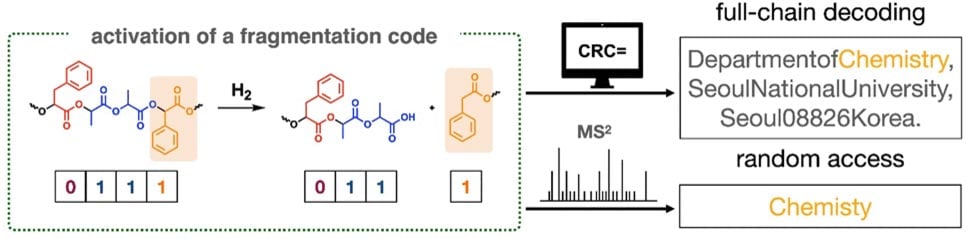
作为演示,该团队将他们的大学地址编码为ASCII,并将其与错误检测码(CRC,一种用于确保数据完整性的既定方法)一起翻译成二进制代码,即一串1和0。
这个512位序列存储在由两种不同单体组成的聚合物链中:代表1的乳酸(lactic acid)和代表0的苯乳酸(phenyllactic acid)。隔一段时间,它们还包含含有扁桃酸(mandelic acid)的片段码。当化学激活时,这些链在这些位置断裂。在他们的演示中,他们获得了18个不同大小的片段,可以通过MS2测序单独解码。
专门开发的软件最初根据片段的质量和末端基团进行鉴定,如质谱图2所示。在MS2过程中,先前测量的分子离子进一步分解,然后对这些碎片进行分析。可以根据片段的质量差对片段进行排序。该软件借助CRC错误检测码重构了整个链的序列,克服了聚合物链的长度限制。
该团队还能够解码有趣的比特,而无需对整个聚合物链进行排序(随机访问),例如代码中的“化学”(“chemistry”)一词作为地址。考虑到地址的各个部分都按照特定的顺序(部门、机构、城市、邮政编码、国家),并用逗号分隔,他们能够在链中隔离所需信息存储的位置,并仅对相关片段进行排序。
本研究得到了韩国科技信息通信部(Ministry of Science and ICT, South Korea)的资助(NRF-2020R1A5A1019631, NRF-2022R1A2C3013240/Ministry of Science and ICT, South Korea)。
上述介绍,仅供参考。欲了解更多信息,敬请注意浏览原文或者相关报道。
Digital information encoded in polymers has been exclusively decoded by mass spectrometry. However, the size limit of analytes in mass spectrometry restricts the storage capacity per chain. In addition, sequential decoding hinders random access to the bits of interest without full-chain sequencing. Here we report the shotgun sequencing of a 512-mer sequence-defined polymer whose molecular weight (57.3 kDa) far exceeds the analytical limit of mass spectrometry. A 4-bit fragmentation code was implemented at aperiodic positions during the synthetic encoding of 512-bit information without affecting storage capacity per chain. Upon activating the fragmentation code, the polymer chain splits into 18 oligomers, which could be individually decoded by tandem-mass sequencing. These sequences were computationally reconstructed into a full sequence using an error-detection method. The proposed sequencing method eliminates the storage limit of a single polymer chain and allows random access to the bits of interest without full-chain sequencing.
转载本文请联系原作者获取授权,同时请注明本文来自诸平科学网博客。
链接地址:https://wap.sciencenet.cn/blog-212210-1459743.html?mobile=1
收藏