博文
pymol中小分子的叠合 ligand align,atom align,pair_fit
||
背景:
高斯对小分子采用不同的优化条件(二面角不同11-9-1-7),从log文件中提取分子的构象,由于能量较大,想看看比较小分子构象的差异
叠合前的图片
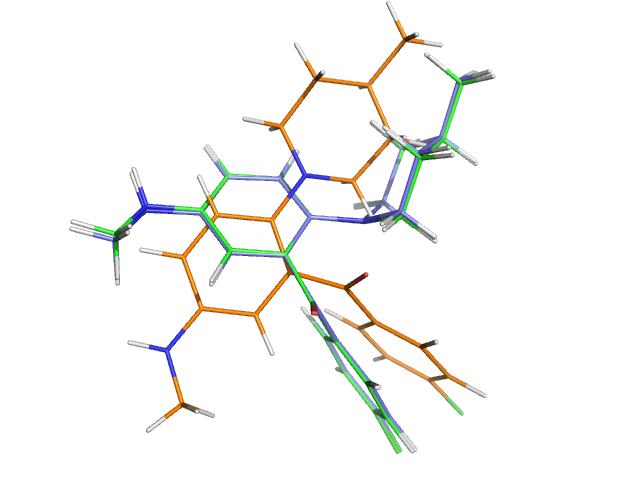
pair_fit 命令叠合相关原子
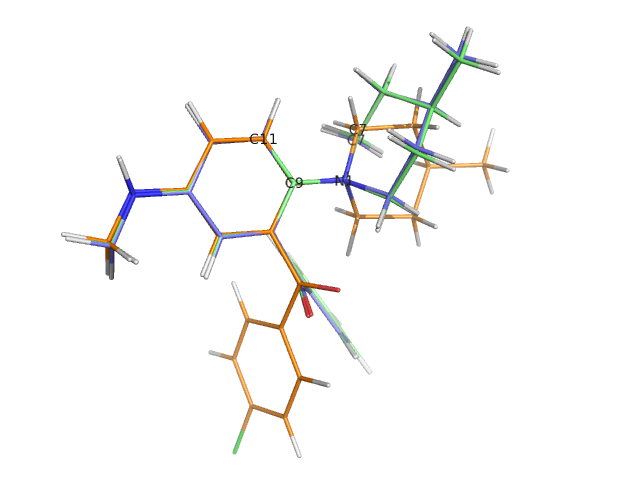
PyMOL>pair_fit phe_330log////C9+N1+C11+C10, phe_335log////C9+N1+C11+C10
ExecutiveRMS: RMS = 0.004 (4 to 4 atoms)
PyMOL>pair_fit phe_340log////C9+N1+C11+C10, phe_335log////C9+N1+C11+C10
ExecutiveRMS: RMS = 0.049 (4 to 4 atoms)
叠合后的图片
参考
http://www.pymolwiki.org/index.php/Pair_fit
多个原子叠合用+号,
不要
pair_fit ligA////C1, ligB////C8, ligA////C2, ligB////C4, ligA////C3, ligB////C10
这样简写即可
pair_fit ligA////C1+C2+C3, ligB////C8+C4+C10
https://wap.sciencenet.cn/blog-950202-787736.html
上一篇:perl hash 的拷贝深拷贝 浅拷贝 deep copy
下一篇:perl 遗传算法
扫一扫,分享此博文
全部作者的其他最新博文
- • 个人新博客
- • rdkit 绘图
- • vscode 主题设置
- • chemdraw 绘制生物体系示意图