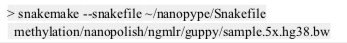博文
Nanopype: 模块化、可伸缩的 Nanopore 数据处理 pipeline
|
长读长的三代 Nanopore 测序技术使得研究者们可以解决很多之前短读长策略不能解决的问题。用户基础的快速扩大和通量的持续增加激发了很多专用软件的开发。但是,如何利用可重现及透明的(reproducible and transparent)流程高效处理 Nanopore 数据集,依然存在不足。为解决这一问题,德国学者开发了 Nanopore 数据处理 pipeline 工具 Nanopype,并于 2019 年 6 月 13 日在 Bioinformatics 杂志上发表了题为"Nanopype: A modular and scalable nanopore data processing pipeline"的论文(https://doi.org/10.1093/bioinformatics/btz461)。
※※※
Nanopype 是一个整合了多个基础的生物信息学软件并维持输出格式的一致性和标准化的 Nanopore 数据处理 pipeline。Nanopype 可以无缝整合到计算机集群环境中,使得这一框架适用于高通量的应用场景。总之,Nanopype 可以增加 Nanopore 数据分析流程的可比较性并强化生物学观点的可重现性。
※※※
Nanopype 是一套基于 Snakemake 和 Python 语言的 pipeline 工具,源代码地址为:https://github.com/giesselmann/nanopype。
※※※
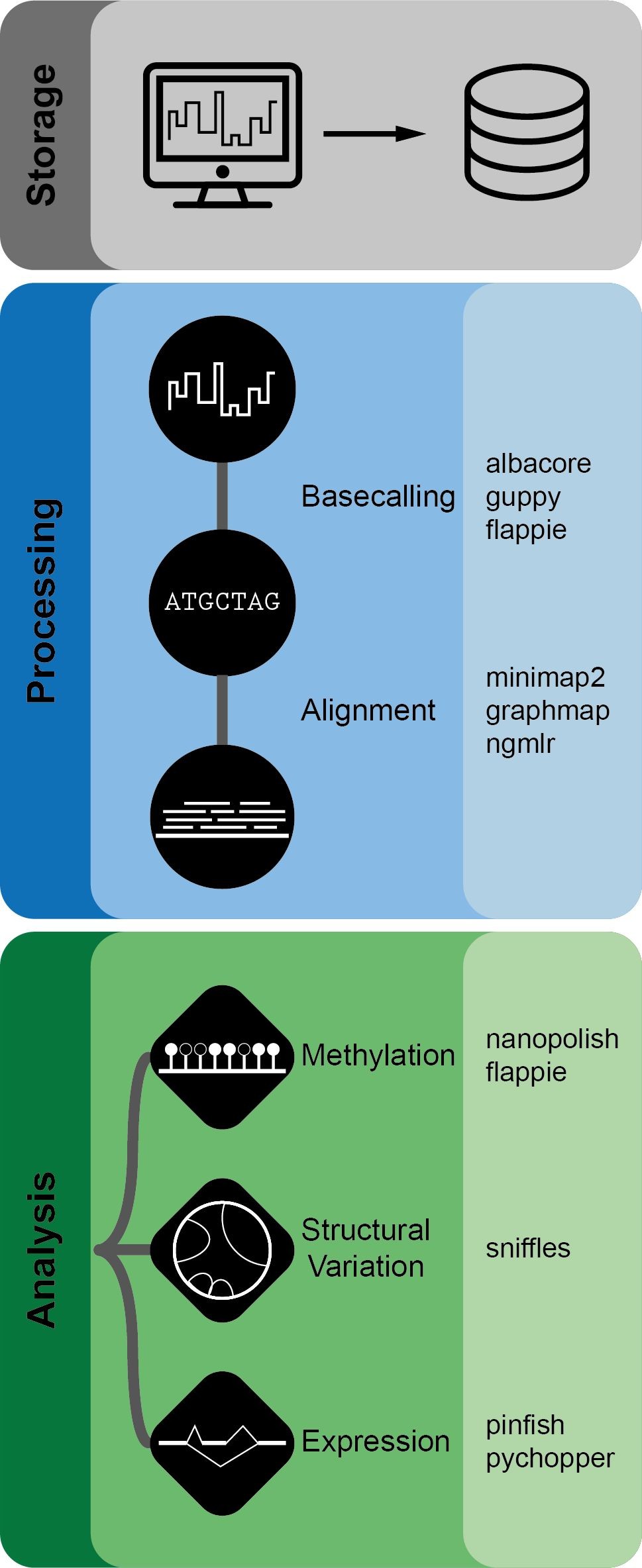
Nanopype 的工作流程如下,包含 Basecalling, Alignment, Methylation, Structural Variation 和 Expresion 几个模块,每一个模块分别整合了一个或多个软件工具。
※※※
Nanopype 的基本用法为:
详细的文档参见 https://nanopype.readthedocs.io。

https://wap.sciencenet.cn/blog-656335-1185930.html
上一篇:植物基因组(plantgenomes)微信公众号 2017 年推文列表
下一篇:PyPore: Nanopore 测序数据处理工具