博文
单细胞脑类器官筛选识别自闭症发育缺陷
||
单细胞脑类器官筛选识别自闭症发育缺陷
人类皮层的发育涉及独特而复杂的过程。随着神经管的形成,端脑内的神经上皮细胞增殖、扩张并产生放射状胶质祖细胞、中间祖细胞和外放射状胶质祖细胞。在背侧区域,这些祖细胞产生层状兴奋性神经元。在腹侧端脑,它们产生中间神经元,这些中间神经元迁移到背侧皮层,与兴奋性神经元结合。这些过程是由精确的、高度协调的遗传和分子程序控制的,其中许多程序仍然是难以捉摸的。对神经发育障碍(NDD)的研究促进了我们对人类大脑发育的理解,并有助于揭示它是如何出错的。然而,许多NDD,如自闭症谱系障碍(ASD),只有在出生后大脑发育几乎完成时才被诊断出来。在人类背景下分析与ASD相关的发育和细胞类型特异性缺陷是至关重要的,但通常仅限于神经影像学和死后组织研究。此外,ASD基因的共表达网络分析表明,与ASD相关的发育缺陷可能出现在胎儿阶段,而这一阶段很难研究。
研究NDD的遗传病因增强了我们对疾病机制的理解,但它通常需要了解人类大脑的发育过程。脑类器官概括了早期大脑发育,并产生了体内发现的多种细胞类型。虽然类器官已被用于研究疾病相关基因,但它们受到表型变异性和低通量的限制。最近的研究将CRISPR筛选技术与类器官相结合,揭示了这种策略在发现新基因功能方面的力量。然而,这种筛选受到低含量读数的限制,当受到遗传干扰时,通常使用指导RNA (gRNA)计数来评估增殖表型。尽管CRISPR筛选与单细胞转录组读数相结合为表型表征提供了前所未有的分辨率,但此类方法尚未在类器官中得到充分探索。单细胞微扰筛选在长期分化的异质组织中的可行性细胞类型尚不清楚。
在这里,Li等人描述的CHOOSE系统(图1),结合平行遗传扰动与单细胞转录组读出脑类器官。他们将条形码对gRNA作为一个汇集的慢病毒文库传递给干细胞,并生成端脑类器官,以在细胞类型和分子途径水平上鉴定36个高风险ASD基因的功能丧失表型。利用单细胞多组学数据,他们构建了脑类器官的发育基因调控网络(GRN),并确定了与基因失调相关的ASD富集调控枢纽。在这36个基因中,在ARID1B的背景下发现了细胞类型组成最显著的变化之一。具体来说,ARID1B的干扰扩大了腹侧放射状胶质细胞,并增加了它们向早期少突胶质细胞前体细胞(OPCs)的过渡,他们在ARID1B患者来源的诱导多能干细胞(iPS细胞)系产生的脑类器官中证实了这一表型。
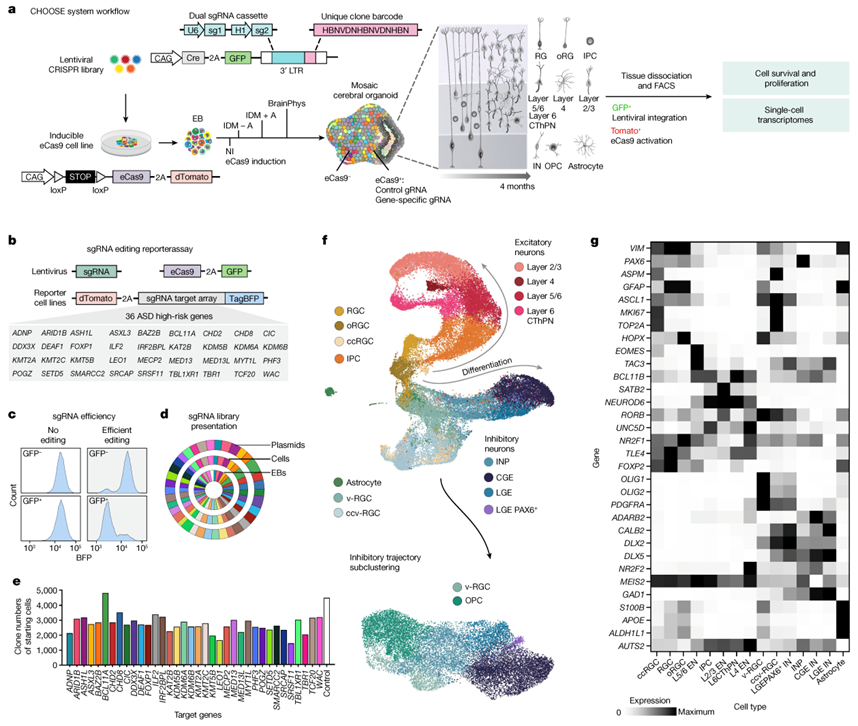
图1 人脑类器官中ASD风险基因的多重筛选的CHOOSE系统。a,CHOOSE系统概述。位于慢病毒3'LTR内的条形码双sgRNA盒。b,报告基因法检测36个ASD风险基因的gRNA效率。c,流式细胞术检测的gRNA编辑效率。图表显示了没有或有效编辑的gRNA示例。d, sgRNA序列读取第5天ASD质粒文库、慢病毒感染的hES细胞和胚状体中测序的gRNA分布。e,每次扰动产生的初始hES细胞克隆数量,用于生成脑类器官。f,包含背侧和腹侧端脑轨迹的单细胞RNA转录组数据集的UMAP嵌入。排除星形胶质细胞和ccv-RGCs的腹侧端脑轨迹的亚聚类和UMAP嵌入来注释OPC簇。g,不同细胞类型中标记基因表达的热图。ccRGC,循环放射状胶质细胞;CThPN,丘脑皮质投射神经元;oRGC,外放射状胶质细胞;IDM,改良分化培养基;NI,神经感应
本文发现的32个ASD高风险基因(ADNP,DDX3X,KMT2A,POGZ,ARID1B,DEAF1,KMT2C,SETD5,ASH1L,FOXP1,KMT5B,SMARCC2,ASXL3,ILF2,LEO1,SRCAP,BAZ2B,IRF2BPL,MECP2,SRSF11,BCL11A,KAT2B,MED13,TBL1XR1,CHD2,KDM5B,MED13L,TBR1,CHD8,KDM6A,MYT1L,TCF20,CIC,KDM6B,PHF3,WAC)可以作为实验验证数据,进行下游验证分析。具体分析结果,感兴趣可参考文献[1]。
参考文献
[1] Li C, Fleck JS, Martins-Costa C, Burkard TR, Themann J, Stuempflen M, Peer AM, Vertesy Á, Littleboy JB, Esk C, Elling U, Kasprian G, Corsini NS, Treutlein B, Knoblich JA. Single-cell brain organoid screening identifies developmental defects in autism. Nature. 2023 Sep;621(7978):373-380. doi: 10.1038/s41586-023-06473-y.
以往推荐如下:
5. EMT标记物数据库:EMTome
8. RNA与疾病关系数据库:RNADisease v4.0
9. RNA修饰关联的读出、擦除、写入蛋白靶标数据库:RM2Target
13. 利用药物转录组图谱探索中药药理活性成分平台:ITCM
19. 基因组、药物基因组和免疫基因组水平基因集癌症分析平台:GSCA
22. 研究资源识别门户:RRID
24. HMDD 4.0:miRNA-疾病实验验证关系数据库
25. LncRNADisease v3.0:lncRNA-疾病关系数据库更新版

https://wap.sciencenet.cn/blog-571917-1416529.html
上一篇:SEMgraph:用结构方程模型对高通数据进行因果网络推理的R包
下一篇:CellAnn:一个全面、超快、用户友好的单细胞注释web服务器