博文
R和Bioconductor的单细胞多模态基准数据集
||
R和Bioconductor的单细胞多模态基准数据集
了解分子与生理之间的定量关系推动了定量分析技术的发展,特别是单细胞测序。单细胞多模态组学技术将单细胞RNA测序与其他分子图谱(如DNA序列、甲基化、染色质可及性、细胞表面蛋白和空间信息)同时结合在同一个细胞中。例如,对来自同一细胞的多个分子测量进行综合分析,可以通过使用CITE-Seq和ECCITE-Seq(通过测序对转录组和表位进行细胞索引,扩展的CRISPR CITE-Seq)定义基于表面标记的亚群来发现罕见的细胞类型,使用scNMT-seq(单细胞核小体、甲基化和转录组测序)发现表观遗传调控和细胞分化谱系,利用10X Multiomics获得单细胞染色质可及性的高分辨率商业版本,利用seq-FISH了解基因表达的空间模式,以及利用G&T-seq(平行基因组和转录组测序)研究健康和疾病状态下基因型-表型的相关性。由于技术限制,其他单细胞多模态数据集从单独的细胞中进行测量,如基于质谱的蛋白质组学方法,包括SCoPE2(单细胞蛋白质质谱分析)。
在单细胞水平上捕获和整合一系列不同的分子信号提出了新的分析挑战。单细胞多模态实验产生多维和大容量的数据集,需要不同的信息和统计方法来存储、处理和分析数据。由于单细胞分析的数据容器和分析工具包的可用性,整合不同的分子层以提供生物学上有意义的见解是R/Bioconductor的一个活跃发展领域。R/Bioconductor是一个开放开发和开源平台,用于分析生物医学和基因组数据数据结构,如单细胞数据的SingleCellExperiment类和多组学数据的MultiAssayExperiment类。两者都是基于SummarizedExexperiment类(用于存储、操作和分析高通量定量组学数据的Bioconductor数据结构)设计的。相对于R编程语言内外的分析平台(如GATK、Seurat、mixOmics、MOFA+、CiteFuse软件包、用于CITE-Seq的ScanPy、用于SCoPE2的Conos), Bioconductor为单细胞多模态数据的统计分析和可视化提供了最广泛的可互操作数据结构和软件包。
长期以来,使用标准化数据类轻松获取公开可用的实验数据在开发用于分析新技术数据的可互操作软件包中发挥了重要作用,有助于围绕共享数据集和常用数据类(如ExpressionSet,然后是SummarizedExexperiment和SingleCellExperiment)将开发工作结合起来。因此,Eckenrode等人引入了一套单细胞多模态基准数据集,通过Bioconductor内ExperimentHub软件包SingleCellMultiModal对多模态分析方法进行基准测试(图1A)。该软件包的范围是为Bioconductor生态系统内的方法开发和基准测试提供对精选的、预集成的、公开可用的基准数据集的有效访问。此外,作者们使用本体术语为每个称为ontomap的实验数据库添加了细胞类型标签。细胞标签元数据帮助用户建立一个共同的基础事实。用户可以通过MultiAssayExperiment获得多模态的综合表示,MultiAssayExperiment是数十个多模态数据分析软件包所依赖的通用核心Bioconductor数据结构。每个数据集都进行了质量控制,要么是原作者在发表期间,要么是实施了质量控制,预先过滤了高质量的细胞。SingleCellMultiModal使用Bioconductor的ExperimentHub包和服务来托管、协调和管理来自云的数据。作者们计划在新的数据集和技术可用时更新该软件包,并且欢迎社区研究人员的贡献。文献《Curated single cell multimodal landmark datasets for R/Bioconductor》作为这些技术的基本方面的审查,适用于生物信息学和统计软件的开发人员,并作为SingleCellMultiModal数据包的描述。
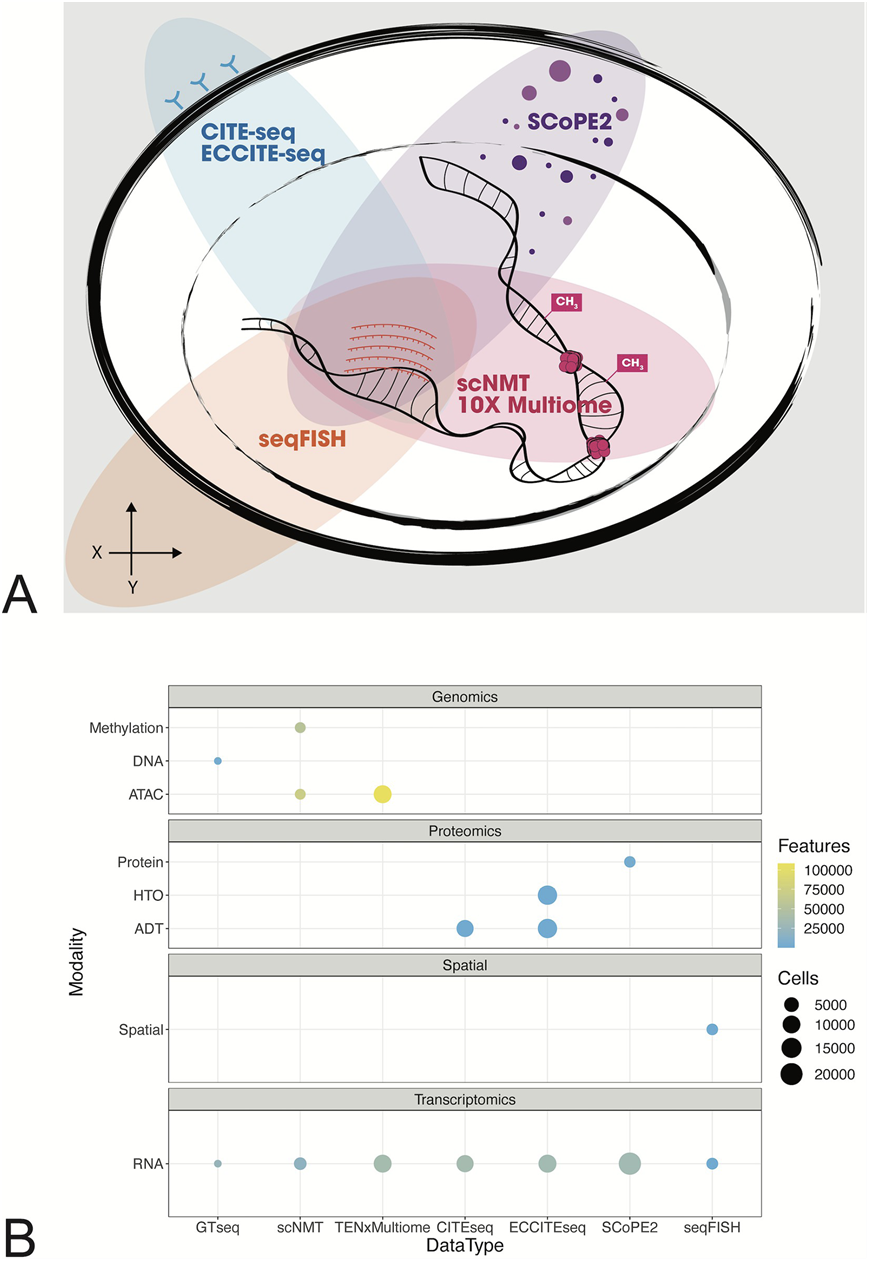
图1 SingleCellMultiModal包中包含的模态表示。(A)每种不同技术收集的模式的维恩图表示,包括:RNA(中),表面蛋白(左上),空间信息(左下),甲基化和开放染色质(右下)和肽(右上)。(B)每种技术为每种数据模式收集的特征和单元数
参考文献
[1] Eckenrode KB, Righelli D, Ramos M, Argelaguet R, Vanderaa C, Geistlinger L, Culhane AC, Gatto L, Carey V, Morgan M, Risso D, Waldron L. Curated single cell multimodal landmark datasets for R/Bioconductor. PLoS Comput Biol. 2023 Aug 25;19(8):e1011324. doi: 10.1371/journal.pcbi.1011324.
以往推荐如下:
5. EMT标记物数据库:EMTome
8. RNA与疾病关系数据库:RNADisease v4.0
9. RNA修饰关联的读出、擦除、写入蛋白靶标数据库:RM2Target
13. 利用药物转录组图谱探索中药药理活性成分平台:ITCM
19. 基因组、药物基因组和免疫基因组水平基因集癌症分析平台:GSCA
22. 研究资源识别门户:RRID
24. HMDD 4.0:miRNA-疾病实验验证关系数据库

https://wap.sciencenet.cn/blog-571917-1406597.html
上一篇:SCOM:刻画ncRNA协同竞争的泛癌特征揭示潜在致癌生物标志物
下一篇:circRNA海绵:广泛分析circRNA表达及其在miRNA海绵中的作用