博文
Normi:利用非冗余互信息推断单细胞基因调控网络
||
Normi:利用非冗余互信息推断单细胞基因调控网络
每个生物体内都存在着由基因组成的各种网络,称为基因调控网络(gene regulatory networks, GRNs),可以用来解释基因之间如何相互调控。近几十年的研究证明,这些看不见的GRN是细胞周期、细胞分化等细胞功能等生物活动的基础。从基因表达谱推断GRN是研究基因调控关系和识别关键调控因子的重要而有效的方法。然而,由于测序分辨率的限制,以往的方法大多是基于大量基因表达数据,即不同类型细胞的混合物。近年来,高通量单细胞RNA测序(scRNA-seq)技术取得了巨大进步,为研究人员提供了以细胞类型特异性分辨率获取大量表达谱的途径。
一般来说,图是一种广泛的描述GRN的方法,其中节点是基因,边缘表示基因之间的调节关系。在过去的几年里,基于各种理论和技术的计算方法被开发出来,用于重建含有scRNA-seq数据的GRN。最常见和最简单的方法是利用相关系数等一些数学概念来表征两个随机变量之间的关系。PPCOR是这类算法中的一个典型。通过计算它们之间的偏相关系数和半偏相关系数来近似地测量它们的相互作用强度。由于相关系数的对称性,它只能输出与真实生物过程不一致的无向网络。LEAP引入固定的时间窗和不同的时间滞后来量化有向相关性。尽管相关系数方法易于实现和计算,但它们在学习非线性调节关系方面的能力有限。回归是恢复调节关系的另一种方法。以SINGE为例,它通过基于核的格兰杰因果回归减轻了不规则伪时间和缺失值的影响。对每组基因进行多次回归后采用改进的Borda计数法对预测结果进行汇总,从而构建GRN。或者, SINCERITIES通过转录因子(transcription factors, TFs)的表达变化预测下一个时间窗内靶基因的表达分布。此外,一些传统的GRN推理算法,如布尔网络和基于常微分方程(ODE)的方法,仍然占有中等比例。 然而,它们中的大多数都受到大规模数据的计算负担的困扰。为了平衡计算效率和捕获非线性关系的能力,互信息(MI)是另一种广泛使用的方法。PIDC使用多变量信息度量每对的部分信息分解,并使用上下文似然关联算法构建GRN。由于它对边的两个方向都有等效的测量,因此所构建的网络是无向的。
尽管提出了各种用于GRN推断的方法,但由于单细胞基因表达数据集中存在大量的缺失和伪时间信息引入的额外噪声,这项任务仍然具有挑战性。为了解决这些问题,Zeng等人最近提出了一种新的基于非冗余互信息的GRN推理方法—Normi(图1)。为了避免噪声对表达和伪时间的影响,作者们首先对scRNA-seq数据集进行预处理,将整个发育轨迹划分为几个线性片段,并使用固定大小的滑动窗口将每个片段划分为更小的切片,然后在窗口内应用平滑平均策略获得最终的代表性细胞。在使用混合KSG估计器计算两个基因之间的高阶时滞互信息之前,采用距离相关来确定不同的调控时滞。构建初步GRN后,作者们采用最大相关最小冗余算法(mRMR)去除冗余调节边,得到最终GRN。通过使用模拟数据和scRNA-seq数据集评估了Normi与其他最先进的GRN推理算法的性能,研究结果表明,Normi方法优于其他方法,并且在面对缺失值时显示出其稳定性。
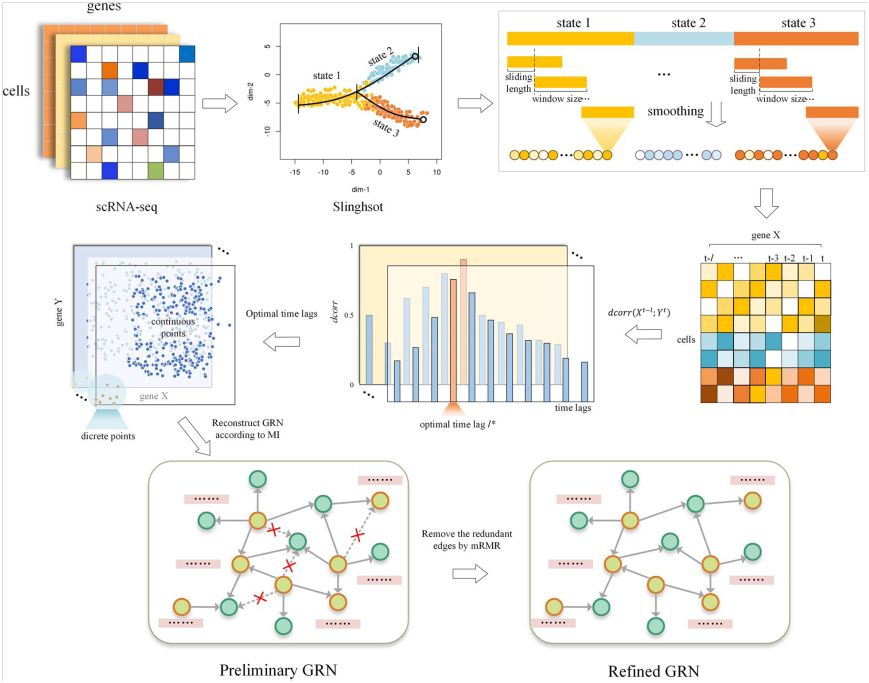
图1 Normi的工作流程。首先对输入的scRNA-seq进行预处理,将整个轨迹划分为多个片段,然后对每个片段应用滑动窗口和平均策略,得到光滑的代表性细胞。接下来,计算每个基因对的距离相关,以确定最佳的时间延迟。利用混合KSG估计器量化最优时滞下基因间的调控强度。最后,采用mRMR去除初始GRN中的冗余调节边,得到精细化网络
Normi使用Python实现,相关代码见https://github.com/CSUBioGroup/Normi。
参考文献
[1] Zeng Y, He Y, Zheng R, Li M. Inferring single-cell gene regulatory network by non-redundant mutual information. Brief Bioinform. 2023 Sep 20;24(5):bbad326. doi: 10.1093/bib/bbad326.
以往推荐如下:
8. RNA与疾病关系数据库:RNADisease v4.0
9. RNA修饰关联的读出、擦除、写入蛋白靶标数据库:RM2Target
13. 利用药物转录组图谱探索中药药理活性成分平台:ITCM
19. 基因组、药物基因组和免疫基因组水平基因集癌症分析平台:GSCA
22. 研究资源识别门户:RRID
24. HMDD 4.0:miRNA-疾病实验验证关系数据库

https://wap.sciencenet.cn/blog-571917-1405057.html
上一篇:生物复杂性的进化
下一篇:NetRank:基于网络的生物标志物发现方法