博文
细胞特异性基因调控网络识别方法CeSpGRN
||
细胞特异性基因调控网络识别方法CeSpGRN
单细胞基因表达谱数据通常用于发掘单个细胞之间的差异,今儿识别细胞类型和发现新的细胞类型。生物网络,尤其是基因调控网络(gene regulatory networks, GRNs)可以充当细胞的另一个特征来刻画每个单细胞的独一无二特性。虽然单细胞转录组数据日益丰富,但是构建细胞特异性基因调控网络方法还很缺乏。
本次就介绍一种新的细胞特异性基因调控网络识别方法(Cell Specific GRN,CeSpGRN,如图1)。CeSpGRN使用一种高斯加权核(Gaussian weighted kernel)来构建细胞特异性基因调控网络。给定一个细胞,该高斯加权核能够使用该细胞以及该细胞的上游和下游细胞的基因表达谱信息进行学习。还有一个特点是,CeSpGRN能够适用于任何细胞轨迹和聚类结构的单细胞转录数据来挖掘细胞特异性基因调控网络。为了进一步提高细胞特异性基因调控网络识别精度,CeSpGRN还提供了一个监督版本CeSpGRN-TF。CeSpGRN-TF在CeSpGRN识别接触上添加了先验信息(TF-target调控信息)。

图1 发表在bioRxiv预印本平台的CeSpGRN论文
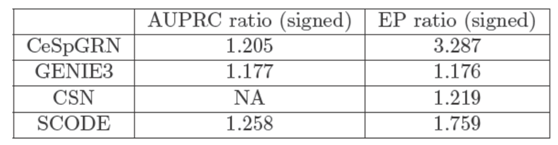
应用于模拟数据和真实数据发现,CeSpGRN及其变种CeSpGRN-TF在细胞轨迹分析和基因调控网络识别精度方面比其他方法(GENIE3、CSN和SCODE)普遍优越(如表1所示)。
表1 四种方法(CeSpGRN、GENIE3、CSN和SCODE)在群细胞水平基因调控网络精度比较
CeSpGRN在小规模基因个数的运行速率还可以,例如构建50个和100个基因单细胞转录数据之间的基因调控网络运行时间分别是182.35秒和3654.61秒(如表2所示)。按照变量个数增加时间呈指数被增加,基因在1万左右的运行时间应该要几天。
表2 不同基因大小条件下CeSpGRN运行时间

如果对CeSpGRN方法感兴趣,可以访问CeSpGRN的Python代码链接https://github.com/PeterZZQ/CeSpGRN。
参考文献
[1] Ziqi Zhang, Jongseok Han, Le Song, Xiuwei Zhang. Inferring cell-specific gene regulatory networks from single cell gene expression data. bioRxiv 2022.03.03.482887; doi: https://doi.org/10.1101/2022.03.03.482887
以往推荐如下:
2. Any RNA
3. 癌症内RNA
4. 一对搭档:计算与生物
5. miRNA组学
7. 细胞通讯推理框架
10. 环状RNAs与衰老
11. 生物网络的关系推断
12. 网络模块识别已20年
13. miRspongeR 2.0版本来了
14. 单细胞测序技术进展


https://wap.sciencenet.cn/blog-571917-1356302.html
上一篇:单细胞测序技术进展
下一篇:细胞特异性因果调控网络识别