博文
一种简单易行的方差分解方法
||
此前的博文中,给大家介绍了如何用几个圆圈来进行方差分解的方法,但事实上该方法依然有一定的复杂性,且几个圈交汇处的方差并不太容易解释。
那么有没有更简单一点的方法,能够方便的对比回归模型
Y=b0+b1*x1+b2*x2+…
中x1, x2, x3…对Y贡献的相对大小,以及不同X所属的因素类别(如生物因素,非生物因素)对Y的贡献大小呢?
当然有,本文就试图介绍这样一种方法,供大家在实际应用中参考。
要深入理解这种方法,首先要理解回归方程的本质意义:即代表了X变化一个单位时,Y会相应的变化多少。
理解了这一点后,我们引入标准化这个概念,所谓数据的标准化,不过是把原始数据的尺度进行转换,将其范围锁定在一个区间之内(如0-1之间)。通常我们用到的标准化是把某一变量的均值转变为0, SD转变为1. 比如在一个最简单的模型中:
Y=b0+b1*x1
如果我们对x1进行了标准化(mean=0, SD=1),我们记为standard_x,那么这时模型变为:
Y=b0+b1*standard_x
这时,由于x1的变化而造成Y发生的变化幅度该如何衡量呢?我们可以直接用SD来表示这种变化,因为b0为常数,所以SD(b0)=0, 所以这一项不予考虑。那么Y的变异只来自于b1*standard_x这一项。则:
SD(Y) = SD(b1*standard_x) = b1*SD(standard_x)
前面我们提到了,standard_x是标准化后的x, 所以,
SD(standard_x) = 1,
那么非常简单:上述公式变成了
SD(Y) = SD(b1*standard_x) = b1*SD(standard_x) = b1*1 = b1
这时候,X对Y的影响的大小就变成X变化一个SD, 对应的Y会变化多少, 所以这种方法消除了量纲,不同X的回归系数具有了直接可比性。比如以下模型中如果x1, x2, x3…都是标准化后的值,
Y=b0+b1*x1+b2*x2+b3*x3+…
那么b1, b2, b3直接表示的就是各个x变化一个SD, Y对应的变化多少。
所以这时候,我们便可以直接用b1, b2, b3…的大小作为不同x对Y贡献大小的依据,且其值具备了直接可比性(消除了X的量纲),这就是依据这种方法进行方差分解的核心原理。到这里,大家体会到了对X进行标准化的妙处了吧(当然,标准化的妙处不止这一点)。
但是,还是要强调一下,这种方法的应用的前提是,不同的X之间的相关性必须较低,否则首先还是要处理共线性问题。
好了,原理讲完,通过一个案例来演示一下这种方法的具体应用。案例来源(Gross et al. 2017, PNAS),其具体研究内容不做赘述,我们看他的问题:
分析24个变量对Y(logM5)的影响,并判定这24个变量对Y的影响的相对大小(即对R2)的贡献程度。
这24个变量是:LAT, SINLONG, COSLONG, ARIDITY, SLO, SAND, PH, I(PH^2), ELEVATION, CWM_logSLA, I(CWM_logSLA^2), CWV_logSLA, I(CWV_logSLA^2), CWS_logSLA, CWK_logSLA I(CWK_logSLA^2), CWM_logH, I(CWM_logH^2), CWV_logH I(CWV_logH^2), CWS_logH, CWK_logH, I(CWK_logH^2) , SR。
而这24个变量, 又可以归为Rrichness, Abiotic, Geo等五大类。其最终结果如下图所示:
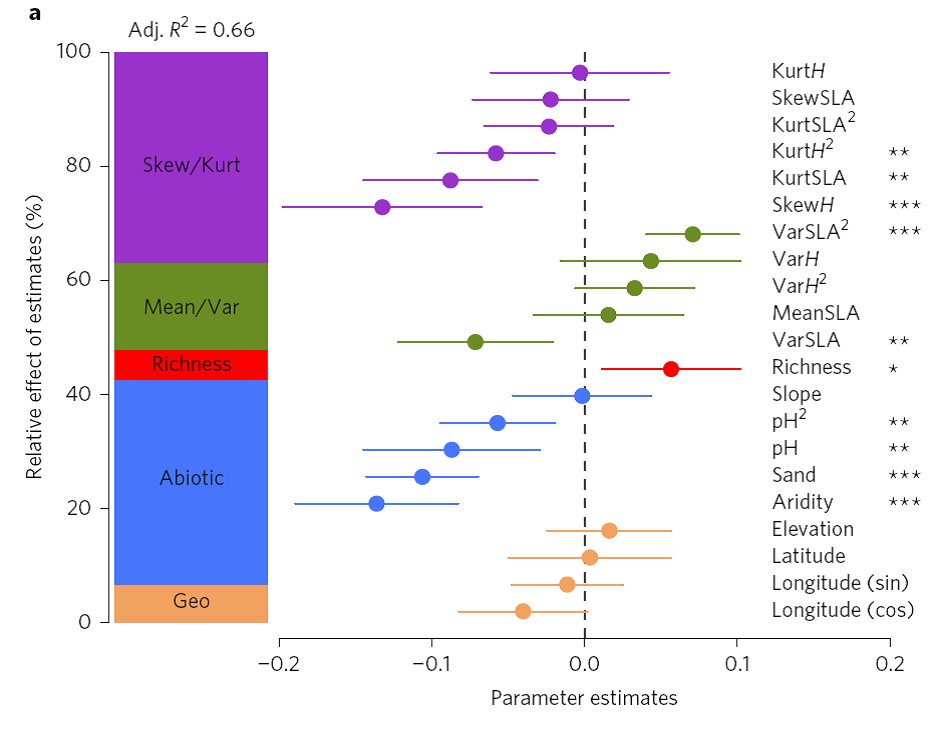
案例来源(Gross et al. 2017, PNAS)
那么这个结果是如何得到的呢?
首先我们判定这些X对Y的总体贡献是多少。
其实很简单,构造一个fullmodel, 包括所有的变量(当然要把共线性高的因素先去除掉)
mod12<-lm(logM5 ~ LAT + SINLONG + COSLONG +
ARIDITY + SLO + SAND + PH + I(PH^2) + ELEVATION+
CWM_logSLA + I(CWM_logSLA^2)+ CWV_logSLA + I(CWV_logSLA^2) + CWS_logSLA + CWK_logSLA + I(CWK_logSLA^2) +CWM_logH + I(CWM_logH^2)+ CWV_logH + I(CWV_logH^2) + CWS_logH + CWK_logH + I(CWK_logH^2) +
SR , data=datatotal)
此全模型的r2即为论文中fig4a(即上图)中报道的r2, 即0.66。
但是细心的读者会发现, 上图中x也不够24个, 其他的弄哪儿了?是我搞错了么?别着急, 我们先做一个模型选择, 把那些跟Y的关系比较弱的X通通删除(模型选择也是个技术活, 感兴趣的我们以后慢慢道来)。一定要注意, 这里的每个X都是标准化之后的X。
模型选择的核心代码如下:
########模型选择
dd12<-dredge(mod12, subset = ~ LAT & SINLONG & COSLONG & ARIDITY & SLO & SAND & PH &SR & ELEVATION & dc(CWM_logSLA, I(CWM_logSLA^2)) & dc(CWV_logSLA, I(CWV_logSLA^2)) & dc(CWK_logSLA, I(CWK_logSLA^2)) & dc(CWM_logH, I(CWM_logH^2)) & dc(CWV_logH, I(CWV_logH^2)) & dc(CWK_logH, I(CWK_logH^2)), options(na.action = "na.fail"))
######提取最优模型集, 以AIC值差值小于2为临界点
subset(dd12, delta<2)
求模型平均后的参数估计值, 即figure 4a右边的参数估计图
de12<-model.avg(dd12, subset = delta < 2)
summary(de12)
即得到以下结果:
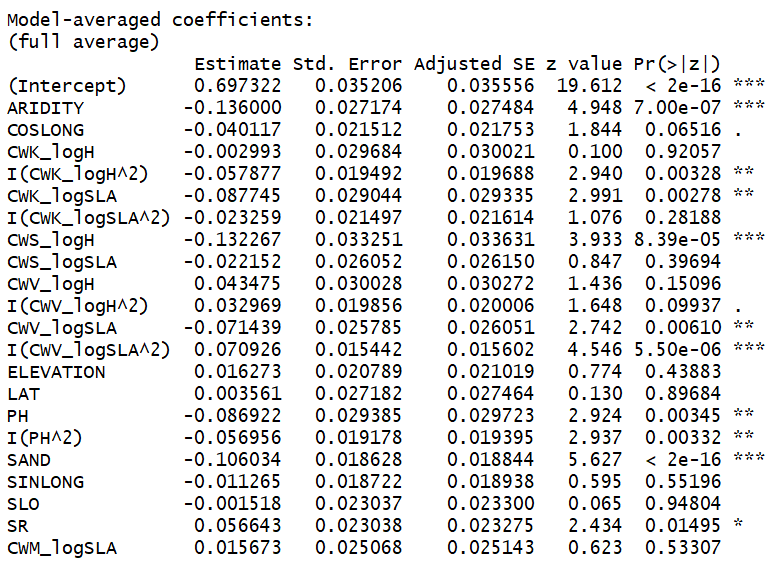
我们提取这些参数估计值, 进行对应的作图:
就得到了跟文献中一模一样的结果, 这里的X跟文章图中的X数量就相同了吧!
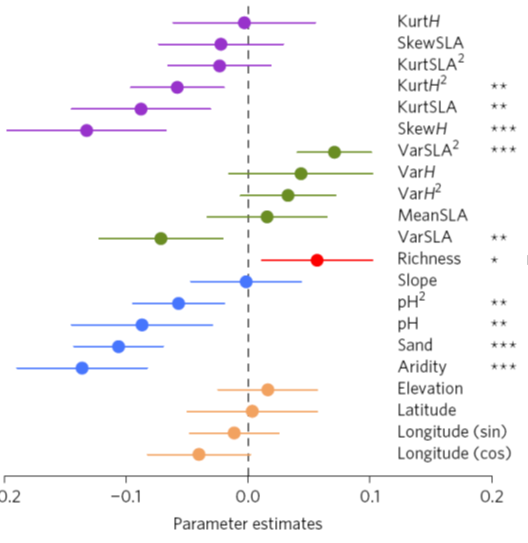
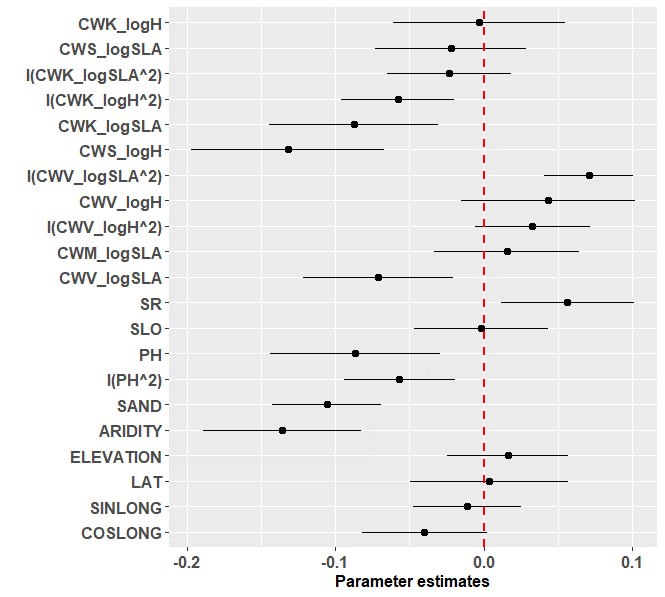
原图结果 重现结果
100%结果完全一样(细节大家自己调吧, 我要哄娃, 就不费这劲了)!
那么, 最核心的方差分解, 也就是下图这个结果如何得到呢?
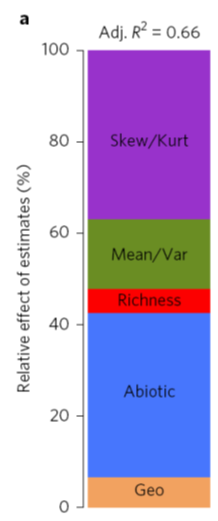
首先, R2=0.66这个值, 我们刚才已经通过fullmodel得到了, 剩下的就是如何分析五类因素各自的贡献了, 那么怎么做呢?
非常简单, 把以上模型平均后的结果中属于每一类的X的回归系数相加, 除以所有X的回归系数之和就可以了。
在R中,可以:
########求各类型参数贡献的百分比
d1<-summary(de12)
d2<-d1$coefficients
d3<-d2[1, ]
d4<-d3[c(2:22)]
d5<-abs(d4)
sum(d5)
如我们要求LAT + SINLONG + COSLONG+ELEVATION这四个因素一起在总R2中所占的比率(也就是图中的geo这一类因子), 那么:

即为geo这个因素在模型解释的总R2中所占的比重为6.6%,
Richness的贡献为:

即richness这个因素在模型解释的总R2中所占的比重为5.2%。
所以用各类回归系数的和除以所有回归系数的和, 就得到了各类因素对的贡献比率, 而所有的这五类因素的贡献加到一起, 当然就是100%, 这个案例里, 他们一起贡献了Y的变异的66%。
这种方法简单易行, 应用也较为常见, 如以下三篇PNAS论文都采用了这一方法。
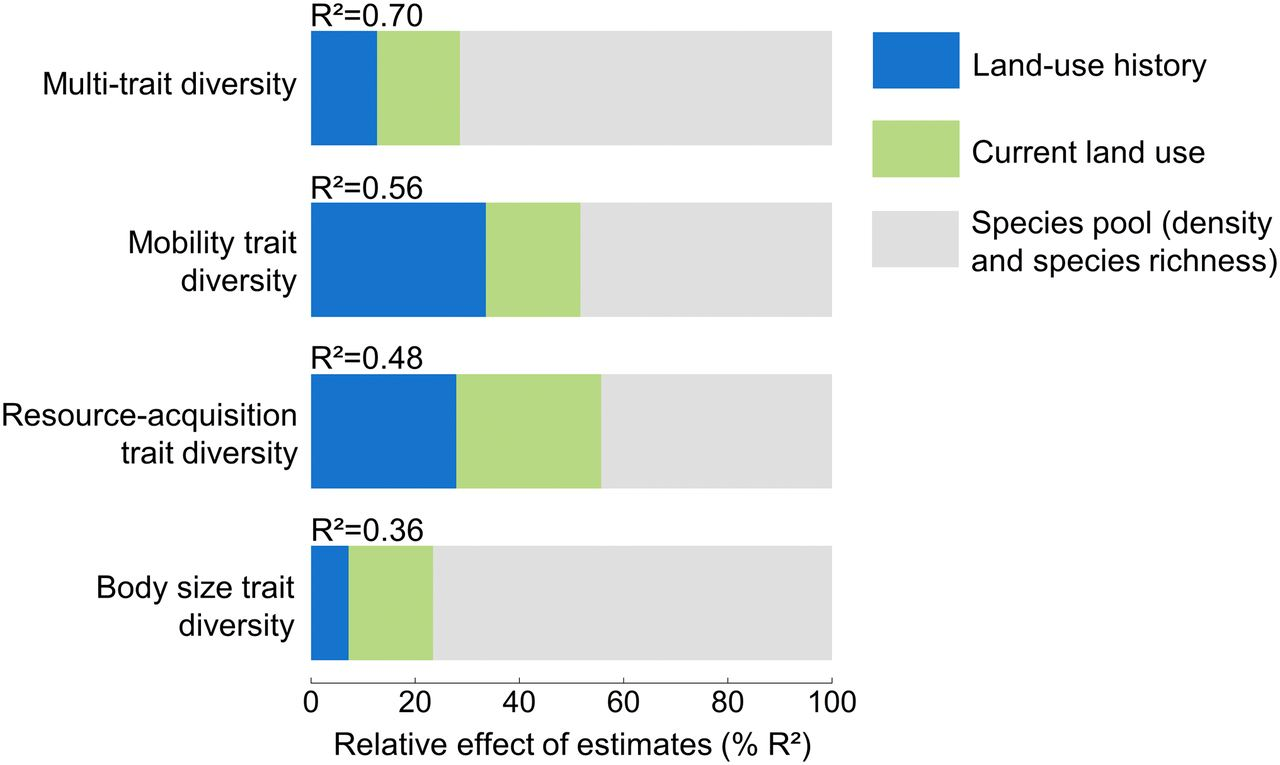
案例来源:(Le Provost et al. 2020, PNAS)
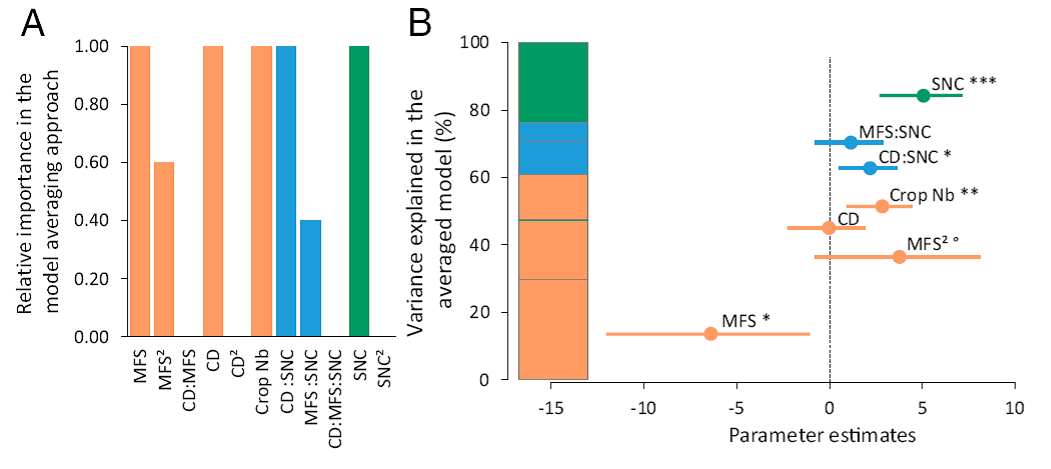
案例来源(Sirami et al. 2019, PNAS)
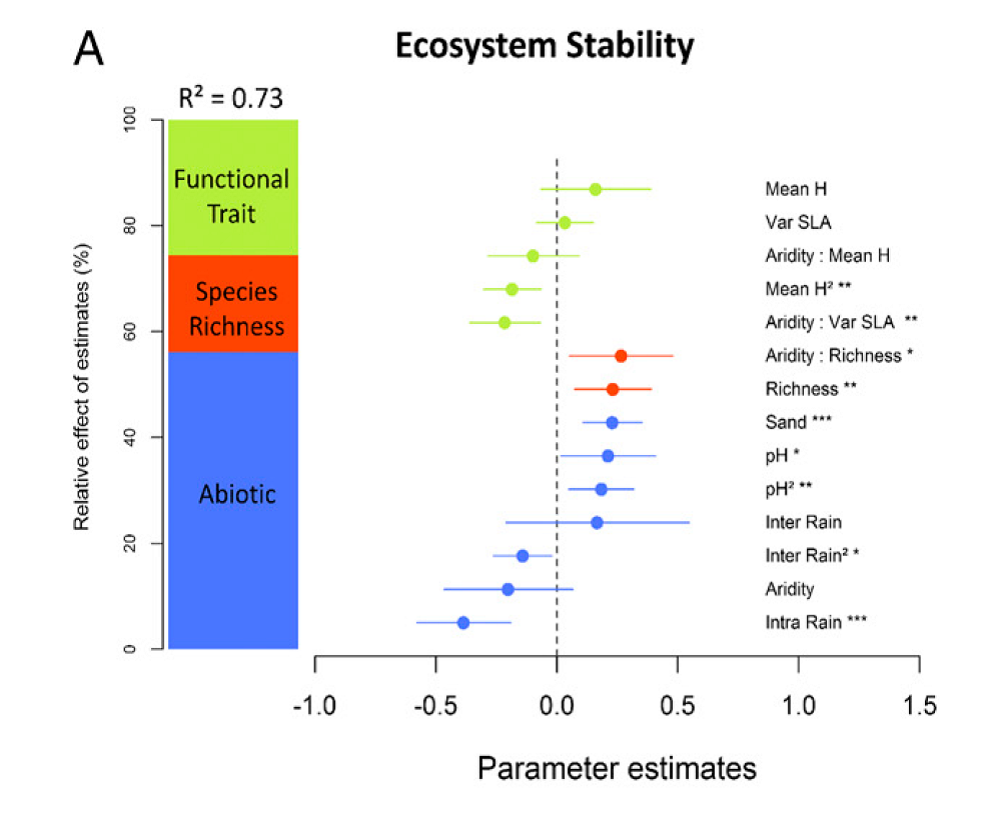
案例来源:(García-Palacios et al. 2018, PNAS)
此外做个小广告, 鄙人将于今年10月底和11月底, 分别做一期meta分析和混合效应模型的线上培训课程(其他老师还有结构方程模型, R统计制图与地图制作, Invest模型等), 敬请关注, 详情请扫描以下二维码

参考文献:
1) García-Palacios, P., N. Gross, J. Gaitán, and F. T. Maestre. 2018. Climate mediates the biodiversity–ecosystem stability relationship globally. Proceedings of the National Academy of Sciences.
2) Gross, N., Y. L. Bagousse-Pinguet, P. Liancourt, M. Berdugo, N. J. Gotelli, and F. T. Maestre. 2017. Functional trait diversity maximizes ecosystem multifunctionality. Nature Ecology &Amp; Evolution 1:0132.
3) Le Provost, G., I. Badenhausser, Y. Le Bagousse-Pinguet, Y. Clough, L. Henckel, C. Violle, V. Bretagnolle, M. Roncoroni, P. Manning, and N. Gross. 2020. Land-use history impacts functional diversity across multiple trophic groups. Proceedings of the National Academy of Sciences 117:1573-1579.
4) Sirami, C., N. Gross, A. B. Baillod, C. Bertrand, R. Carrié, A. Hass, L. Henckel, P. Miguet, C. Vuillot, A. Alignier, J. Girard, P. Batáry, Y. Clough, C. Violle, D. Giralt, G. Bota, I. Badenhausser, G. Lefebvre, B. Gauffre, A. Vialatte, F. Calatayud, A. Gil-Tena, L. Tischendorf, S. Mitchell, K. Lindsay, R. Georges, S. Hilaire, J. Recasens, X. O. Solé-Senan, I. Robleño, J. Bosch, J. A. Barrientos, A. Ricarte, M. á. Marcos-Garcia, J. Miñano, R. Mathevet, A. Gibon, J. Baudry, G. Balent, B. Poulin, F. Burel, T. Tscharntke, V. Bretagnolle, G. Siriwardena, A. Ouin, L. Brotons, J.-L. Martin, and L. Fahrig. 2019. Increasing crop heterogeneity enhances multitrophic diversity across agricultural regions. Proceedings of the National Academy of Sciences 116:16442-16447.
关注以下公众号,发送“方差分解数据及代码”,可获取本文案例数据及详细代码

https://wap.sciencenet.cn/blog-3442043-1253775.html
上一篇:关于Meta分析,你需要了解的十个技术问题
下一篇:PPT分享:Meta分析的原理与要点