博文
教你怎么查基因启动子和转录因子结合位点
|
想做Luciferase、点突变、截短、ChIP,不知如何入门,转录因子与promoter结合位点不知道怎么找?看完这篇文章轻松拿下~
基础知识:
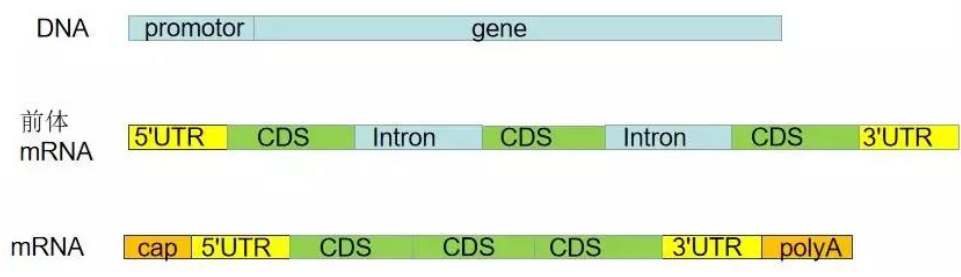
启动子(promoter):与RNA聚合酶结合并能起始mRNA合成的序列。一般选择CDS区上游2 kb。
UTR(Untranslated Regions):即非翻译区,是信使RNA(mRNA)分子编码区(CDS)两端的非编码片段。
5'UTR: 从mRNA起点的甲基化鸟苷酸帽延伸至AUG起始密码子,3'UTR: 从编码区末端的终止密码子(UAG, UAA, UGA)延伸至PolyA尾。

一、查找基因的启动子区域
1. 打开NCBI:https://www.ncbi.nlm.nih.gov/
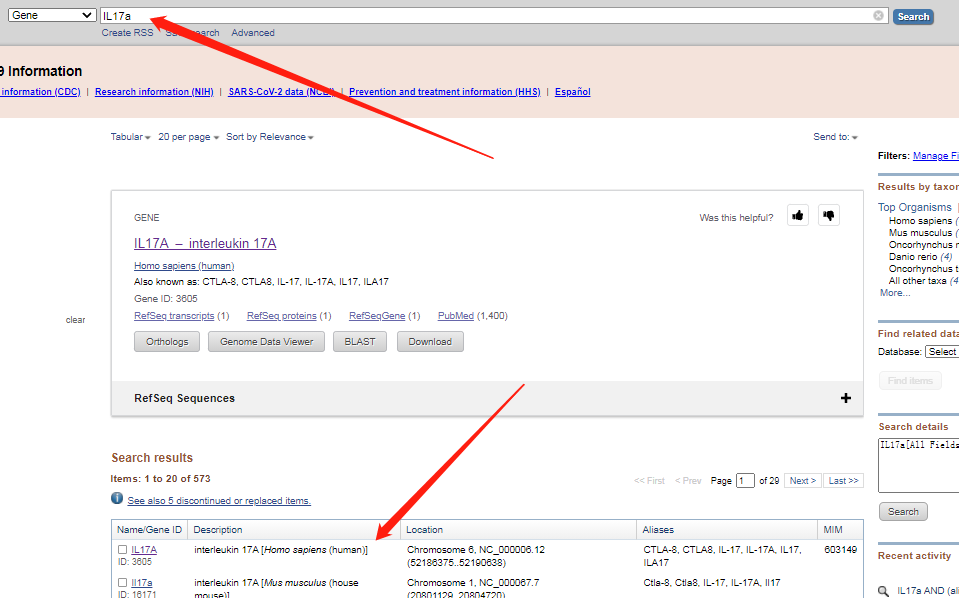
2.选取【Gene】,在旁边输入IL17A,点search,根据物种进行选择,我们以人源[Homo sapiens (human)]为例
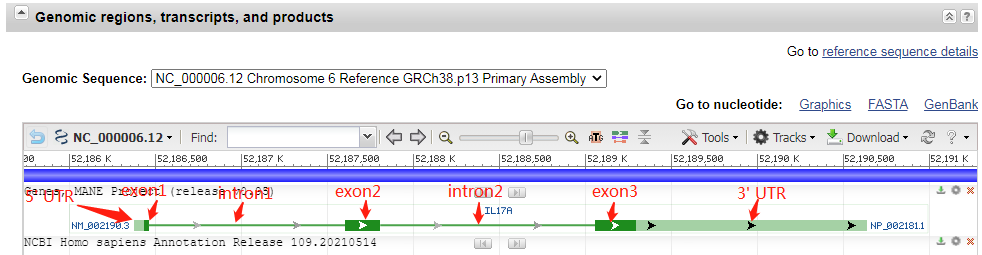
3. 下拉至下图位置,可以看到这个基因的信息:
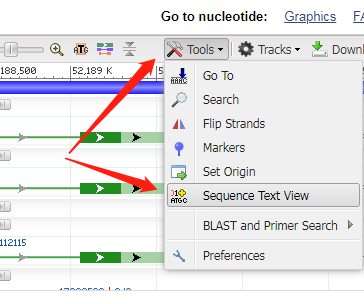
点Tools,选择Sequence Text View,还可以看到序列信息:
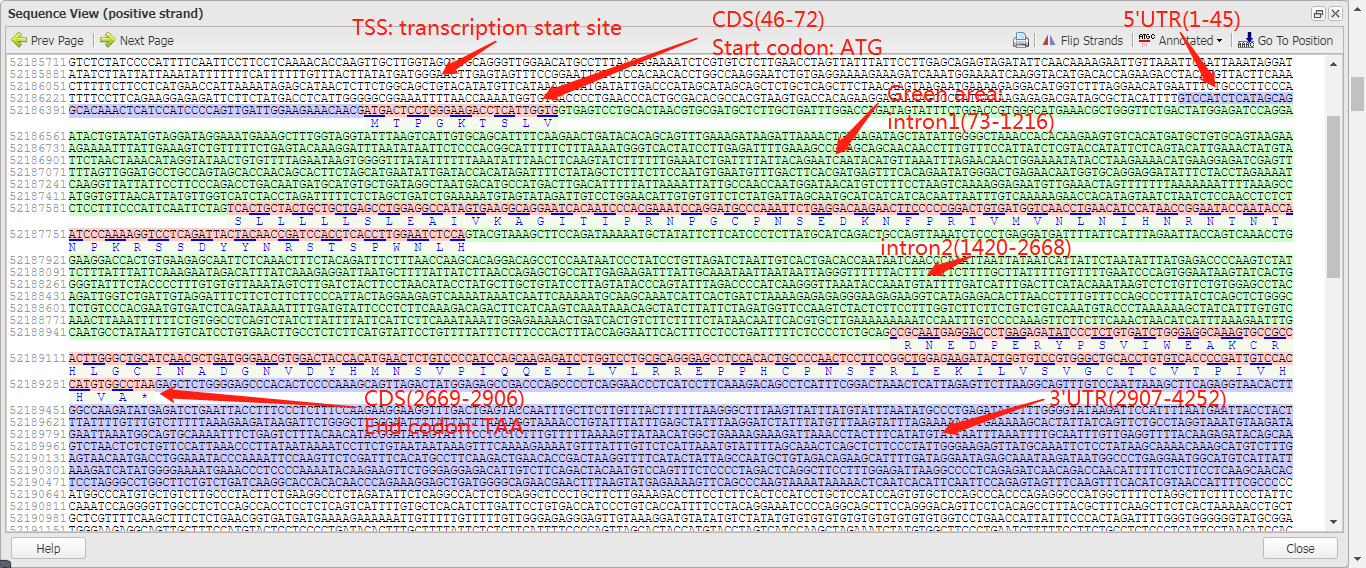
4. 上面的部分只是该基因的一些信息,可以用来查找相应的UTR等区域,接下来我们进入正题,寻找promoter区域。把网页拉到如下图位置,点击FASTA:
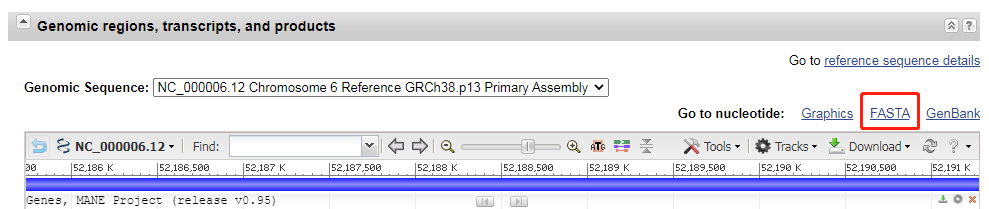
5. 基因位置信息如下图:

6. 一般认为:基因上游2 kb区域为该基因的promoter区域,所以我们将基因上游的2 kb序列先调出来:

复制上述序列,这就是基因的启动子序列了。
二、转录因子结合位点的预测
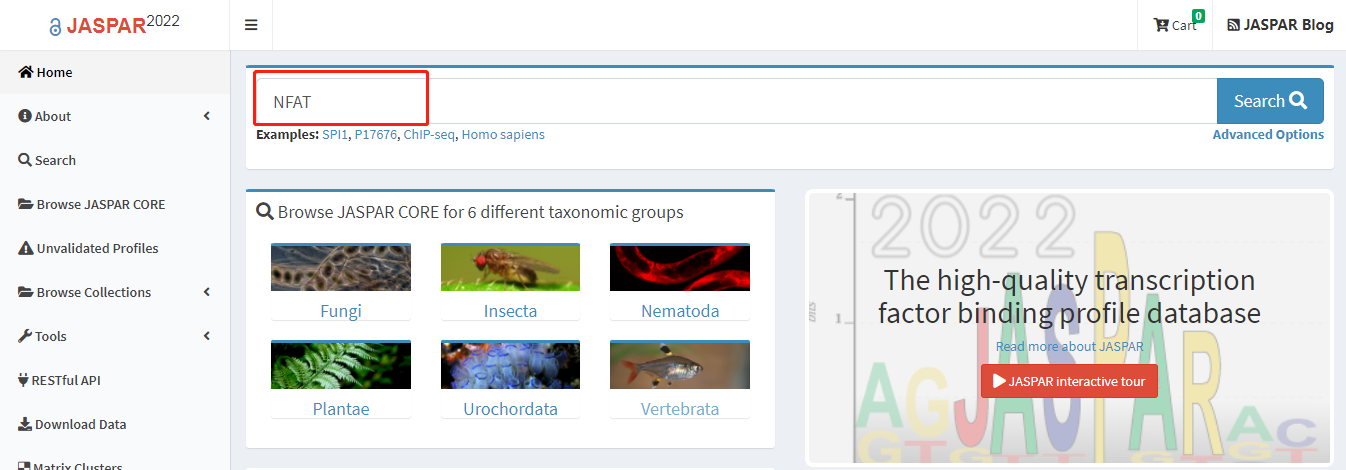
1.打开网址:https://jaspar.genereg.net/,在搜索框里输入转录因子NFAT,点击Search:
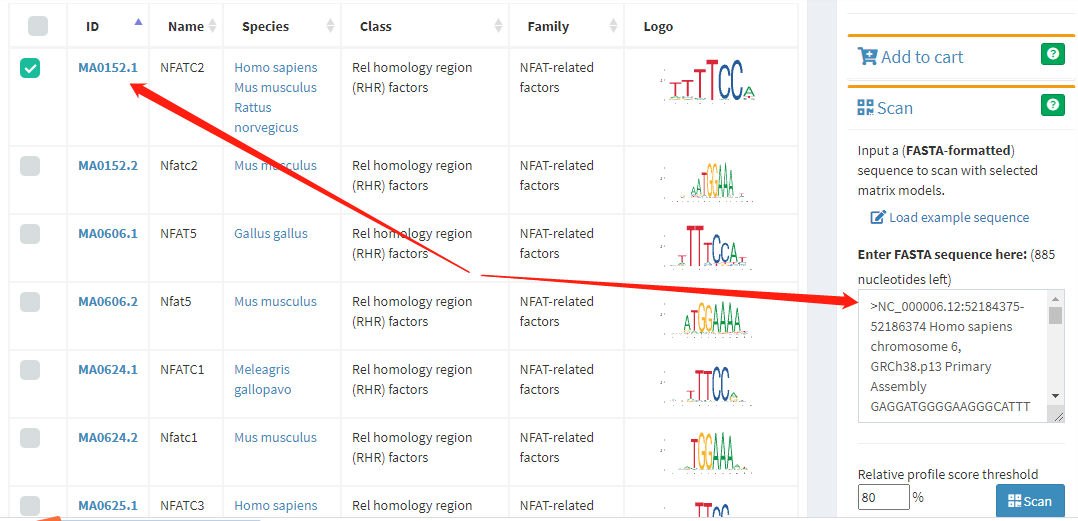
2.选择一个或多个ID文件,在SCAN序列框中输入FASTA格式文件(注意包括>后面的信息)
3. 得到27条转录因子NFAT与IL17A的结合位点,Strand -没有特殊意义,选择Strand +即可。JASPAR数据库检索出的结合位点来源于已发表的ChIP实验等,主要特点是开源、非冗余和高质量。
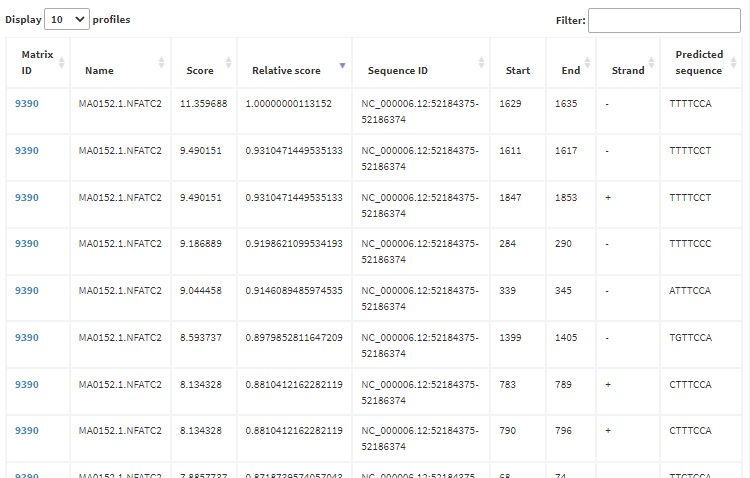
4. 好了,转录因子与promoter结合位点已经有了,接下来就是愉快的实验验证了! Luciferase、点突变、截短、ChIP等统统拉上来就可以了!
参考https://www.sohu.com/a/372441469_120325361
https://wap.sciencenet.cn/blog-3431904-1309566.html
上一篇:[转载]DESeq2原理不简单
下一篇:怎样自主进行基因GO/KEGG富集分析?