博文
用Pymol和VMD制作Gromacs分子动力学模拟后的轨迹动画(蛋白配体复合物)
||
软件准备:
1,VMD安装和制作动画
按照系统版本从http://www.ks.uiuc.edu/Research/vmd/下载对应安装包。
Ubuntu下安装要注意需要修改 bin目录下 vmd.csh的代码。因为Ubuntu不识别-b,所以要把-b的命令删掉才能安装。
$./configure LILUXAMD64 $cd src $make install
打开VMD,载入坐标文件。
打开Graphice-colors调整背景为白色
Display-Axies设置off,关闭坐标显示。
打开Graphice---Representions
通过Create Rep,可以分别对蛋白和配体设置不同的展示方式,其中在Selected Atoms中输入Protein可以只显示受体蛋白,
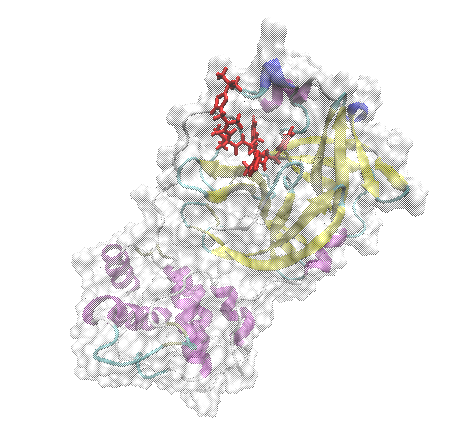
蛋白用两种显示方式,
第一种设置Coloring Method为Secondary Structure.Drawing Method为NewCartoon。
第二种设置Coloring Method为ColorID纯色显示,颜色根据个人喜欢可以设置灰色,Material设置Transparent透明,Drawing Method为Surf.注意Surf这个模式不能用来显示轨迹,因为太慢了。
配体的显示也是通过Selected Atoms来设置,可以特别制定某个氨基酸,命令为resname XXX。XXX为你坐标文件中的氨基酸名称。
导入轨迹,选中复合物然后 File>Load Date into Molecule。
设置Renderer为Snapshot,Movie Settings设置Trajectory,Format设置为MPEG-1.然后点击Make Movie开始制作。原理就是通过连续截图然后用VideoMach连接成video(如果电脑没有装VideoMach的需要安装并且向VMD指名videomach.exe的路径)。
动画制作完成!
2,Pymol 2.3安装和制作动画
之前直接安装的Pymol 1.8和Pymol 2.3试用版,里面的水印通过调整可以处理掉,最近看到李继存老师给的开源版Pymol2.3的安装教程,也就顺便装了开源版,步骤记录如下(win10):
下载Anaconda(https://www.anaconda.com/),安装的时候把环境变量打勾。但实际上卡在了安装过程中,唯一的解释就是这个Anaconda版本和目前的系统冲突。
需要换一个版本,从https://repo.anaconda.com/archive/ 下载2019年的版本
顺利安装。
然后下载pymol安装文件(https://www.lfd.uci.edu/~gohlke/pythonlibs/#pymol-open-source)
numpy+mkl,Pmw和pymol
下载好以后放在Anaconda的安装目录下Scripts文件夹中。
打开CMD,cd到Scripts文件夹
C:\ProgramData\Anaconda3\Scripts> pip install numpy-1.18.3+mkl-cp38-cp38-win_amd64.whl
如果报错
ERROR: numpy-1.18.3+mkl-cp38-cp38-win_amd64.whl is not a supported wheel on this platform.
原因是python的版本和numpy不一致,安装的是python3.8的numpy,而本地是3.7。调整版本对应。
检查一下本机的版本
pip --version
确保安装文件版本一致
重新安装
PS C:\ProgramData\Anaconda3\Scripts> pip install numpy-1.18.3+mkl-cp37-cp37m-win_amd64.whl PS C:\ProgramData\Anaconda3\Scripts> pip install Pmw-2.0.1-py3-none-any.whl PS C:\ProgramData\Anaconda3\Scripts> pip install pymol-2.3.0-cp37-cp37m-win_amd64.whl PS C:\ProgramData\Anaconda3\Scripts> pymol
OK,可以看到了pymol的界面

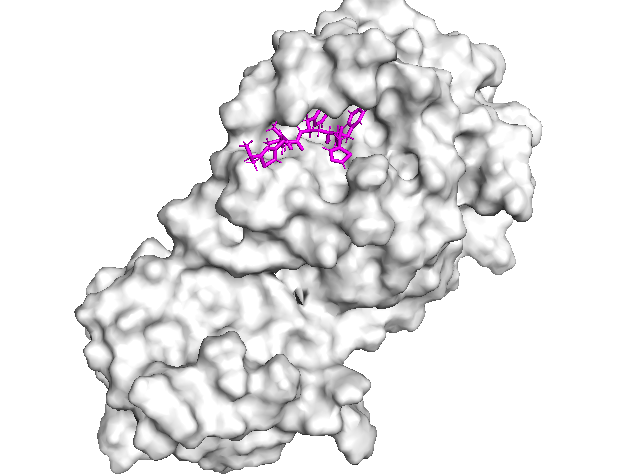
Pymol分别显示受体蛋白和配体的方法比较复杂(如果显示方式为Surface的话),因为不显示配体会在Surface模式下在蛋白上留下一个空洞。如果用其他方式显示的话就无所谓,但是Surface显示需要导入两个不同文件名的坐标文件,并分别导入轨迹。
>load_traj xxx.xtc,start=1,stop=10000000,interval=20
然后在两个坐标的序列中分别删除受体蛋白和配体,这样同时显示两个坐标即可用不同模式显示蛋白和配体(图中为SARS-Cov-2的主蛋白酶和配体的复合物)。
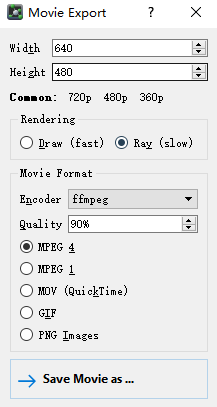
VMD制作动画需要VideoMach,而Pymol制作动画需要FFmpeg支持。可以通过pip或conda安装,或者干脆把
ffmpeg.exe
ffplay.exe
ffprobe.exe
这三个可执行文件放在环境变量里,我放在了Anaconda所在的Scripts文件夹中,它本身就在环境变量中。
之后就可以直接制作动画了
Pymol制作动画最大的好处是可以用Surface来显示,大多数情况下显示活性口袋用Surface比较直观。
制作完成!
https://wap.sciencenet.cn/blog-3341717-1229634.html
上一篇:谷歌浏览器上传图片卡死未响应
下一篇:nmon查看linux下CPU使用率并指定CPU