博文
GWAS计算BLUE值1--计算最小二乘均值(lsmeans)
||
本节,介绍如何使用R语言的lm拟合一般线性模型,计算最小二乘均值(lsmeans)
1. 试验数据
❝数据来源: Isik F , Holland J , Maltecca C . Genetic Data Analysis for Plant and Animal Breeding. Springer International Publishing, 2017.
❞
「数据及代码下载,请关注公众号:育种数据分析之放飞自我,进入知识星球进行相关下载和学习」
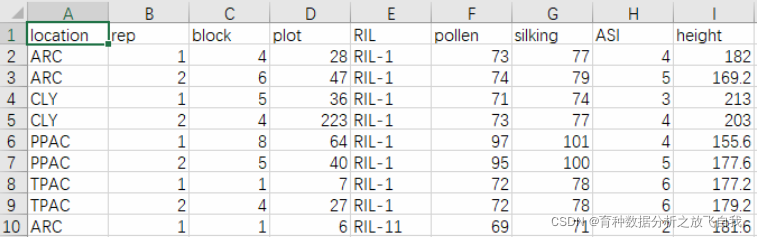
该数据有62个重组自交系(RIL),在4个地点进行试验,随机区组,每个地点2个重复,每个小区种植20株,随机选择5株的表型平均值作为观测值。
2. 用R语言进行一般线性模型拟合
拟合模型:
m1 = lm(height ~ location*RIL + location:rep,data=dat) summary(m1) anova(m1)
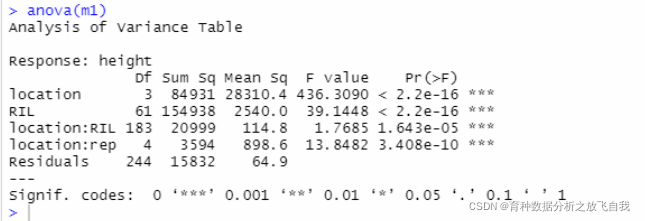 上面是方差分析的结果。
上面是方差分析的结果。
系数的结果是: 注意,这里的值是系数,不是最小二乘均值。这里,如果我们要计算第一个品种RIL1的lsmeans(最小二乘均值),我们需要:
注意,这里的值是系数,不是最小二乘均值。这里,如果我们要计算第一个品种RIL1的lsmeans(最小二乘均值),我们需要:
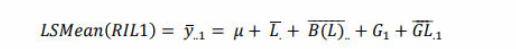 即我们需要整体均值 + 品种RIL1的回归系数 + 地点的效应平均值 + 地点内区组效应品均值 + 品种RIL1和地点互作的效应品均值
即我们需要整体均值 + 品种RIL1的回归系数 + 地点的效应平均值 + 地点内区组效应品均值 + 品种RIL1和地点互作的效应品均值
3. 手动计算RIL11的最小二乘均值
这里我们要计算RIL-11这个品种,
「整体均值」
coef = summary(m1)$coefficients coef["(Intercept)",1]
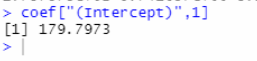 可以看到整体均值为179.7973
可以看到整体均值为179.7973
「地点效应品均值」注意,这里共四个地点,但是只有三个效应值,因为有一个强制为0了,我们再计算平均值时,需要3个地点效应值的和除以4才可以。
coef[c("locationCLY","locationPPAC","locationTPAC"),1]
sum(coef[c("locationCLY","locationPPAC","locationTPAC"),1])/4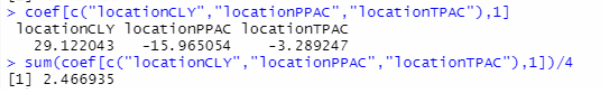 可以看到,效应值为2.466935
可以看到,效应值为2.466935
「地点内区组效应值」
注意,这里共有4个效应值(4*(2-1)),但是分母应该是4*2=8.
coef[c("locationARC:rep2","locationPPAC:rep2","locationCLY:rep2","locationTPAC:rep2"),1]
sum(coef[c("locationARC:rep2","locationPPAC:rep2","locationCLY:rep2","locationTPAC:rep2"),1])/8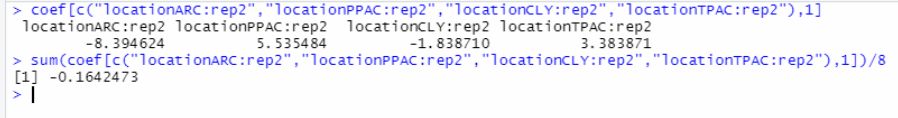 可以看到,效应值为-1642473
可以看到,效应值为-1642473
「品种与地点互作的效应」
coef[c("locationCLY:RILRIL-11","locationPPAC:RILRIL-11","locationTPAC:RILRIL-11"),1]
sum(coef[c("locationCLY:RILRIL-11","locationPPAC:RILRIL-11","locationTPAC:RILRIL-11"),1])/4
loc_ril11 = sum(coef[c("locationCLY:RILRIL-11","locationPPAC:RILRIL-11","locationTPAC:RILRIL-11"),1])/4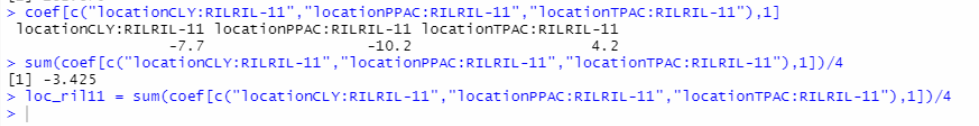 可以看到,效应值为-3.425
可以看到,效应值为-3.425
「RIL-11的效应值为:」
coef["RILRIL-11",1] ril11 = coef["RILRIL-11",1]
 RIL-11的效应值为4.2
RIL-11的效应值为4.2
「最后,RIL-11的最小二乘均值为:182.875」
mu + loc + loc_rep + loc_ril11 + ril11
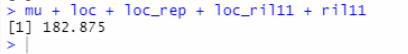
4. 使用函数计算最小二乘均值
之前都是用lsmeans这个包,现在用emmeans,可以看作是lsmeans的升级包。
但是,数据量大时,这个包也是巨慢。
library(emmeans) re1 = emmeans(m1,"RIL") %>% as.data.frame() head(re1,10)
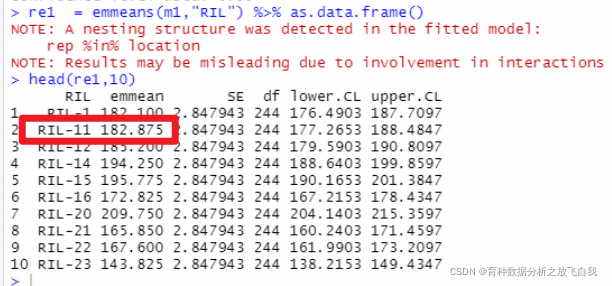 结果是一致的。
结果是一致的。
5. 用asreml进行估计
library(asreml) m2 = asreml(height ~ location*RIL + location:rep,data=dat) summary(m2)$varcomp re2 = predict(m2,classify = "RIL")$pval %>% as.data.frame() head(re2)
这里用predict函数,计算RIL的lsmeans: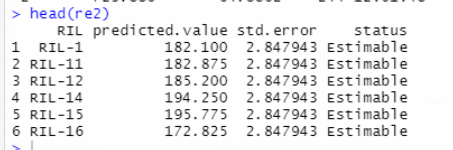 可以看到结果是一致的。
可以看到结果是一致的。
6. 总结
一般,很少用一般线性模型去估算最小二乘均值,都是用混合线性模型,将品种作为固定因子,去估计BLUE值(最佳线性无偏估计)。
用一般线性模型,演示一下如何计算lsmeans,通过手动计算和函数计算两种形式,理解计算方法。
另外,lsmeans和整体平均值不一样,它比平均值更能代表表型值。所以,如果不使用混合线性模型,使用lsmeans作为表型值,也要比平均值更好。
「数据及代码下载,请关注公众号:育种数据分析之放飞自我,进入知识星球进行相关下载和学习」

https://wap.sciencenet.cn/blog-2577109-1316217.html
上一篇:孟德尔抽样误差和基因组选择
下一篇:GWAS计算BLUE值2--LMM计算BLUE值