博文
[转载]微生物组和组学成分数据分析之ALR对数转换
||

微生物组和组学数据集,由于其生物学性质,通常是高维的,特征常以各种成分,如基因、OTU、RNA转录本等的计数为特征。这些数据统称为成分数据。
这类数据分析的中心概念是对数转换,而其中最简单的策略是ALR(Additive log ratio)方法。对于高维数据,ALR方法有一下几个特点:
(a) 次要成分都是相干的
(b) 可以解释100%的总对数方差
(c) 测量结果非常接近于等距。
最近,来自西班牙科学团队的一篇题为“Compositional Data Analysis of Microbiome and Any-Omics Datasets: A Validation of the Additive Logratio Transformation” 的文章指出:
ALR对数转换可以有效提供一组简单的变量来表示整个成分数据集,其关键节点在于选择哪个成分为参考,并使用三个高维组学数据集进行验证。
01
验证方法
通过ALR方法的理论和推导公式(这里不详述,推荐看原文),分别计算总对数方差(The total logratio variance 总结了采样点在多维空间中的分散程度),Logratio Geometry和Procrustes分析,以此找到有效的参照特征。再与其它对数转换方法对比,如CLR对数转换。
02
数据集验证
1. 兔子数据集
数据集为非零数据集,89个样本,3937个特征。
总对数方差为0.1601,Procrstes相关系数最高为0.9991,对应的基因数为856。该基因在3937个基因中的相对丰度排名第201位。
图一为所有3937个特征的Procrstes相关性直方图。为了直观地显示ALR变量接近等距的程度。
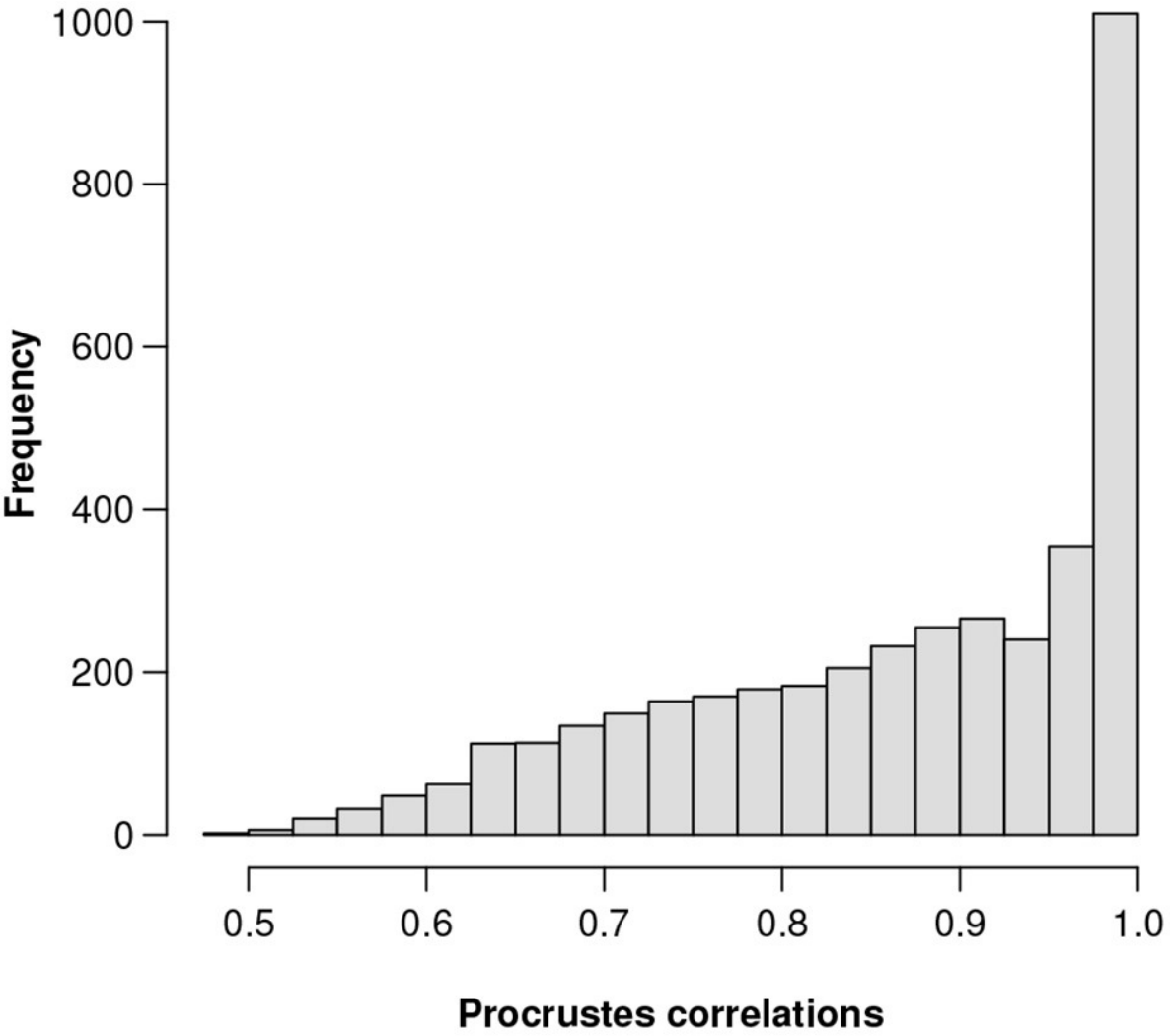
图一
图二显示了在ALR上计算的所有样本间距离,基于所有成对对数的对数距离或同等情况下的所有CLR绘制相应的精确对数距离。
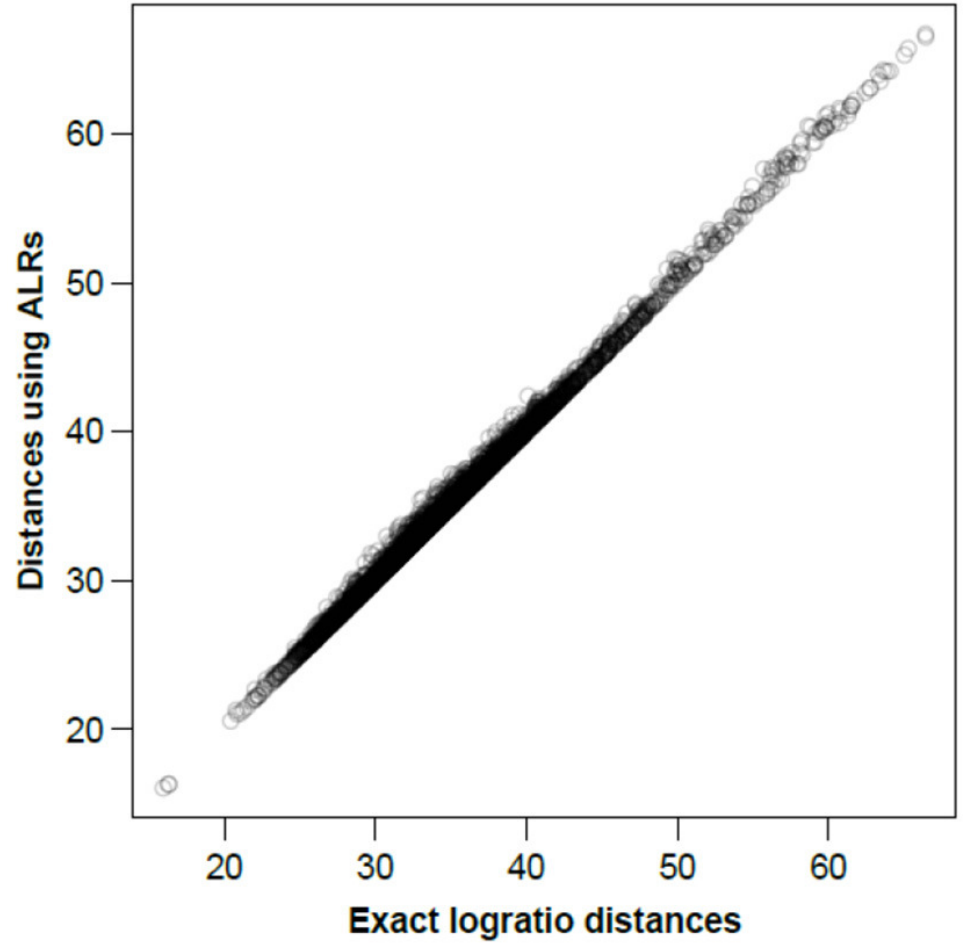
图二
图三为对于数据集的89个样本,参考基因编号856的计数与计数总和之间成正比。
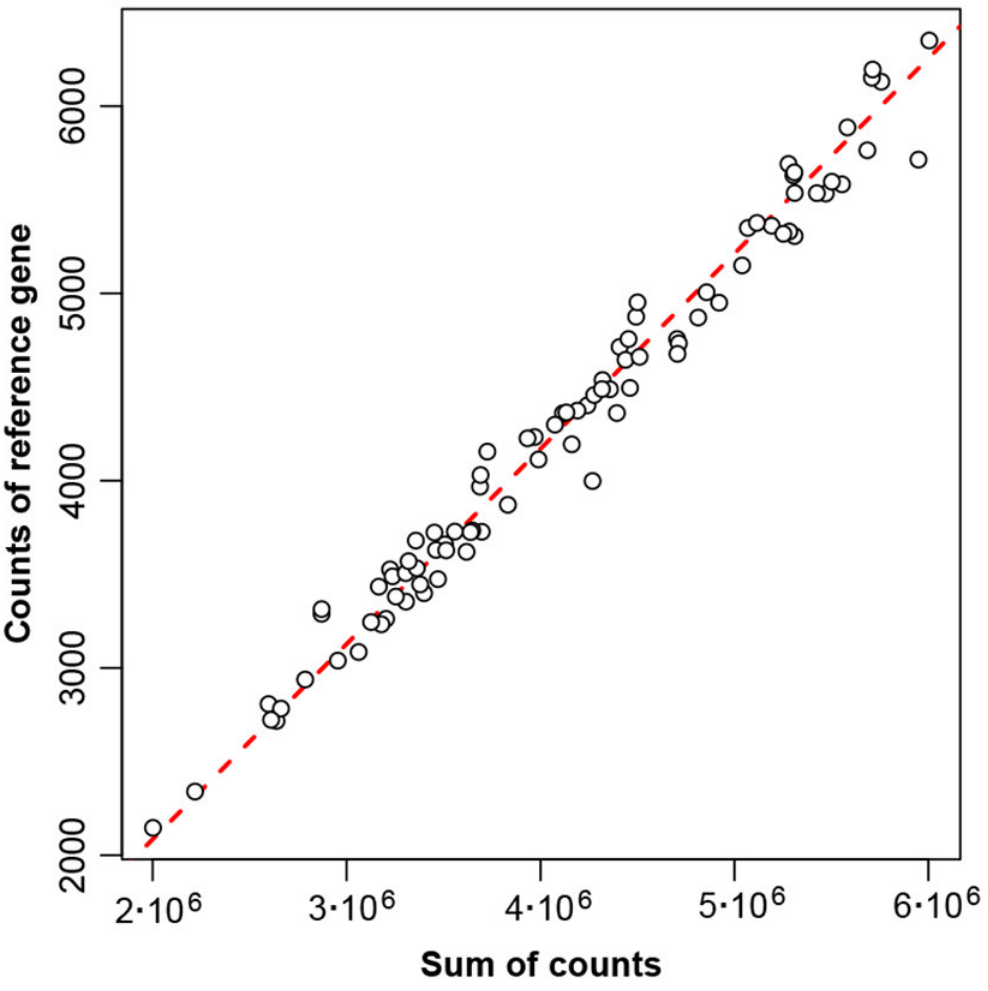
图三
下图四展示了整个数据集的LRA(是所有成对对数的主成分分析(PCA),相当于所有CLR的主成分分析,以加权或非加权的形式)。
而图五中展示了具有参考基因856的ALR的对应PCA。主成分分析与参考成分微生物基因编号为856时,其几何形状实际上与确切的直线几何形状相同(Procrstes相关=0.9991)。字母S和F代表进行测序的两个实验室,显示出明显的分离。
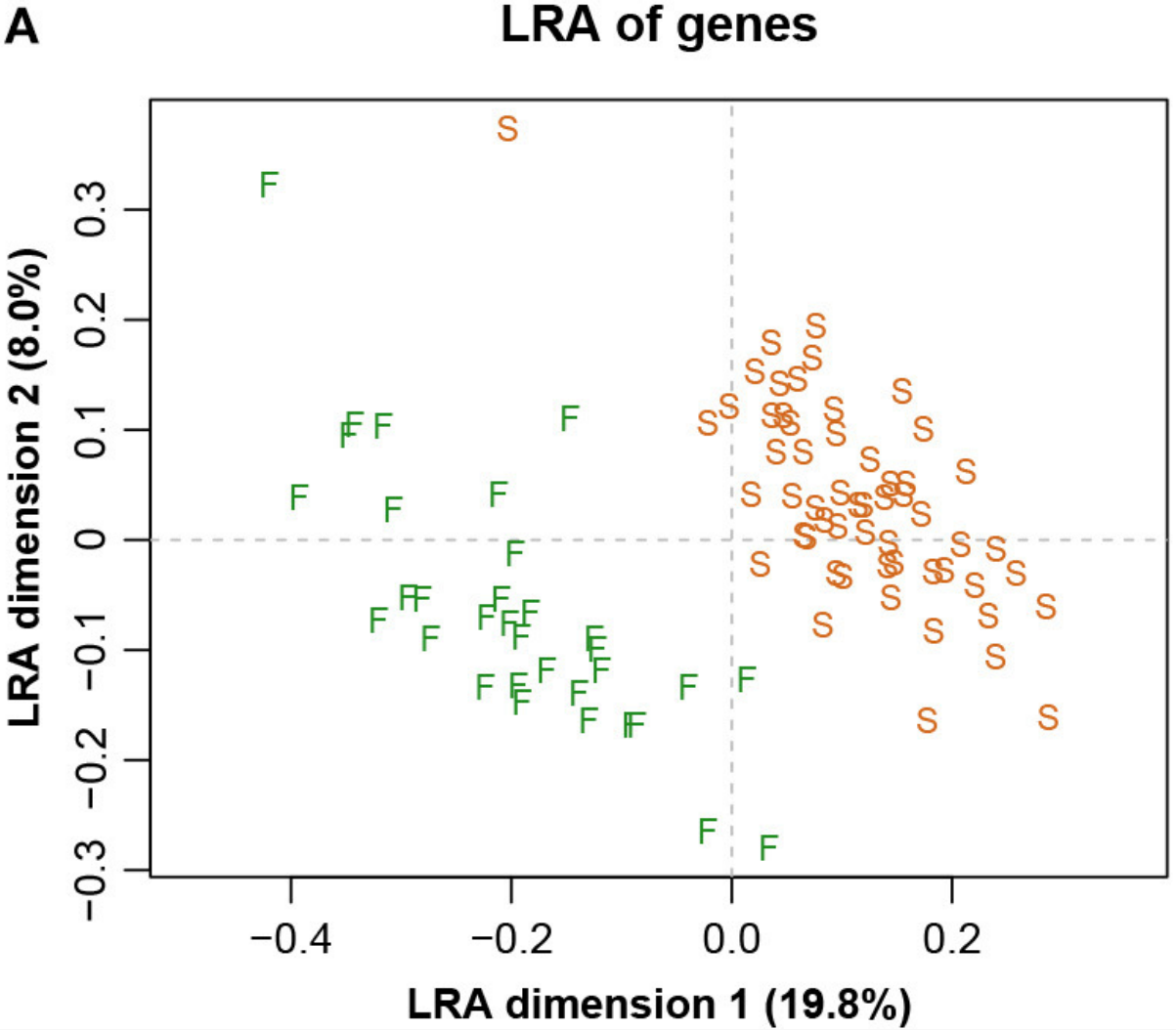
图四
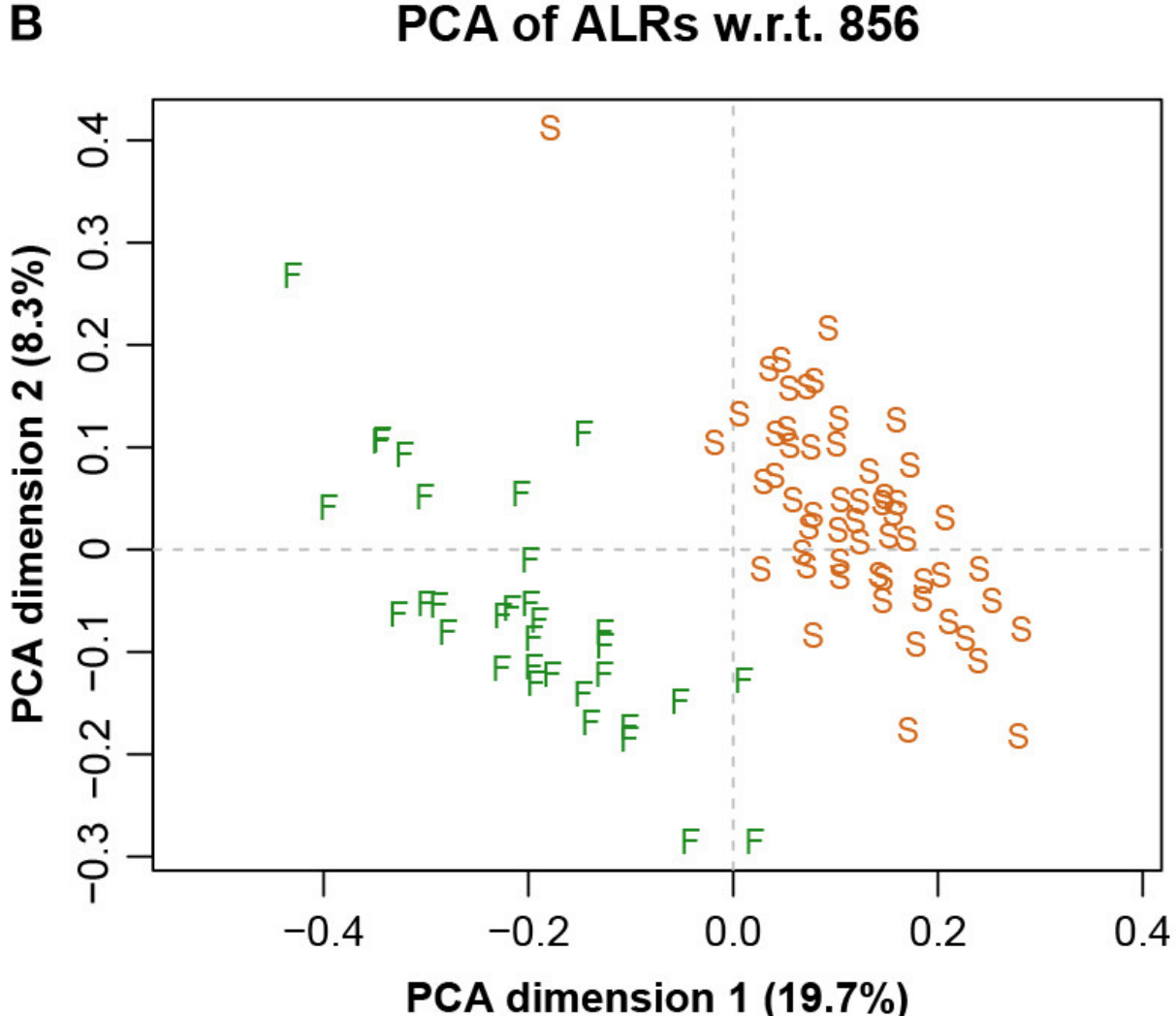
图五
2. 小鼠数据集
数据集大小,28个样本,3147个特征。此数据集中有34个零,使用R包zComposition中的函数cmultReplin替换。
总对数方差 0.2099,Procrustes相关系数最高为0.9977,对应转录本编号1318,其中转录本编号1179的Procrustes相关系数也与其相似。
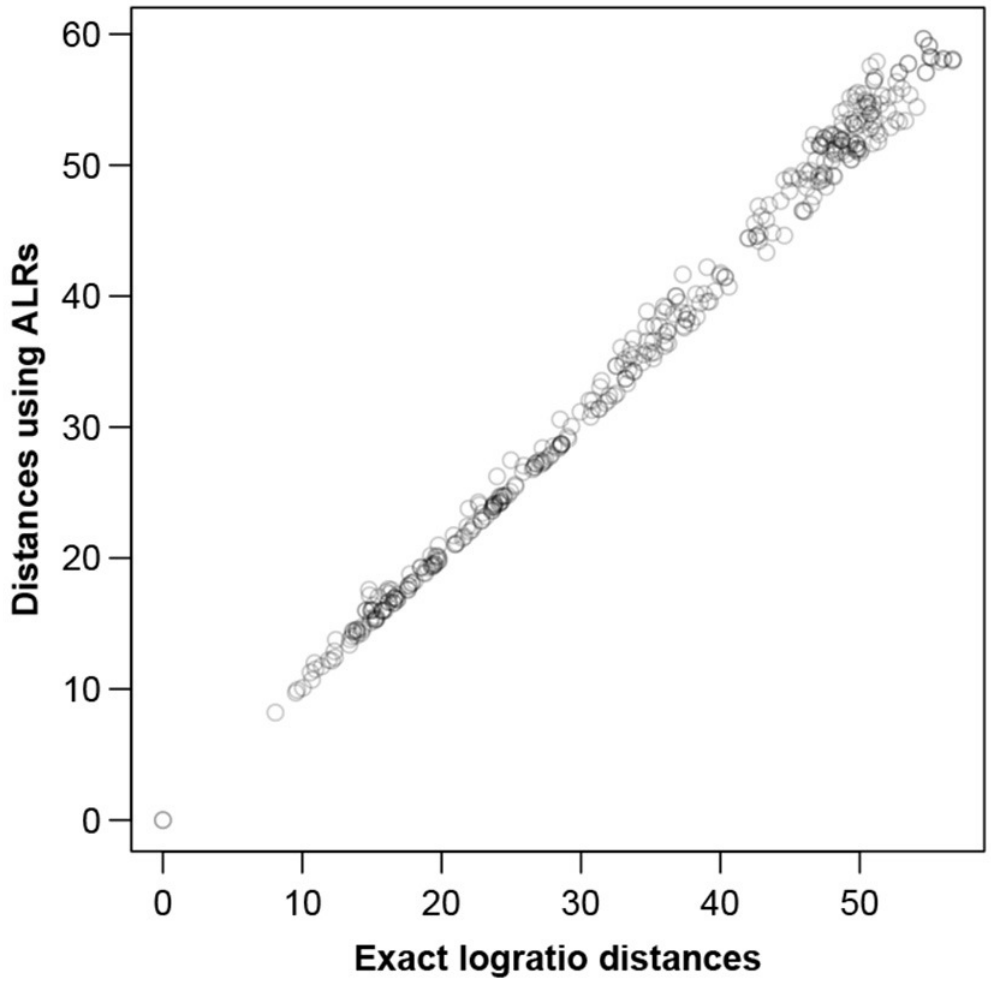
图六
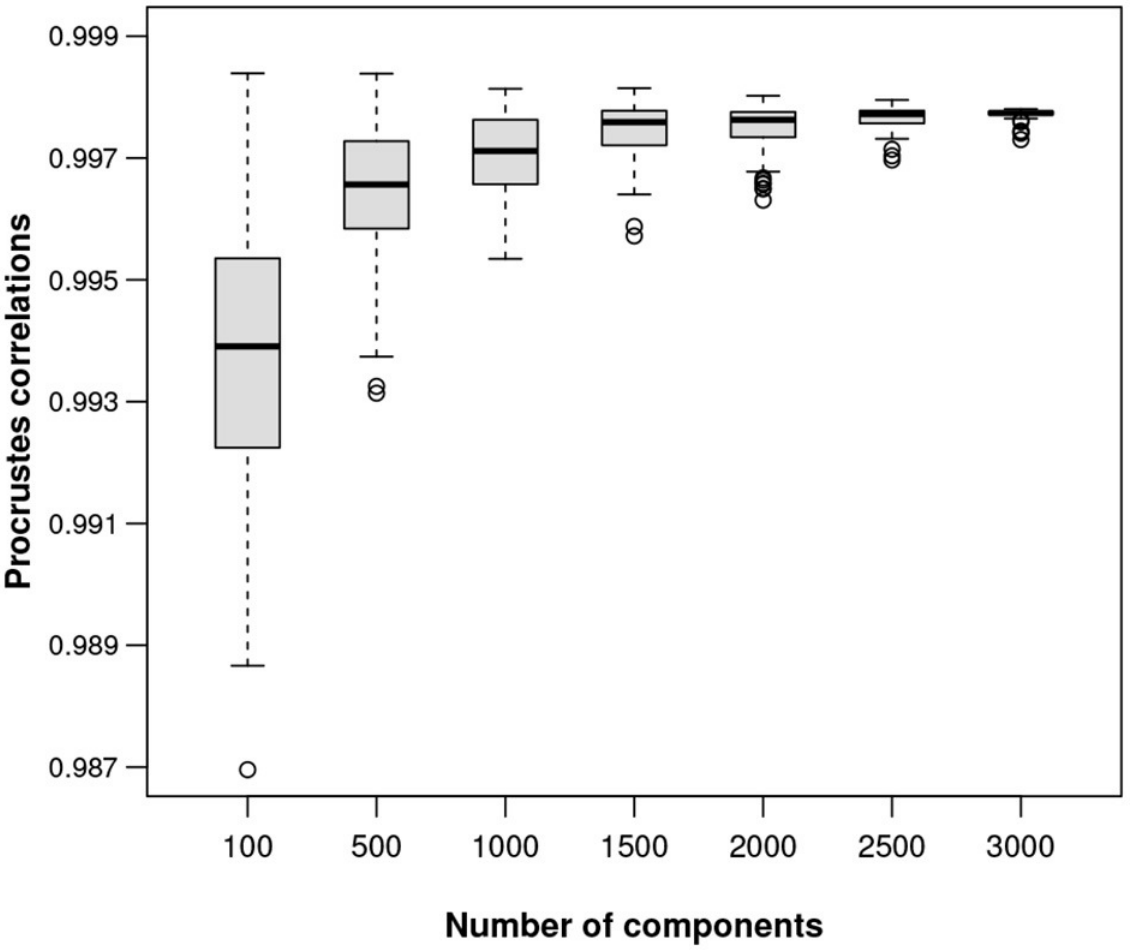
图七
图六显示了在ALR上计算的样本间距离。为了显示任意大小数据集的ALR变换的质量,对MICE数据进行了模拟研究,从数据中随机抽取不同大小的样本,将每个样本作为独好的立的样本,并为该特定数据集的ALR变换找到最佳参考。
对于100、500、1,000、1,500、2,000、2,500、3,000和3,500个转录本的子集,以及每个子集的100个随机样本,绘制最佳的Procrstes相关性,如图七展示。ALR变换的等距质量随着可能的参考成分特征数量的增加而提高。
图八展示完整数据集的LRA,图九展示了参考转录本编号1179的ALR的PCA。它们实际上是相同的,只是有很小的差异,而在这之前的Procrstes相关系数结果就已经指示出了。标签代表两种不同的处理(L和M)和7种不同的时间(0、1、2、4、6、9和12h)。
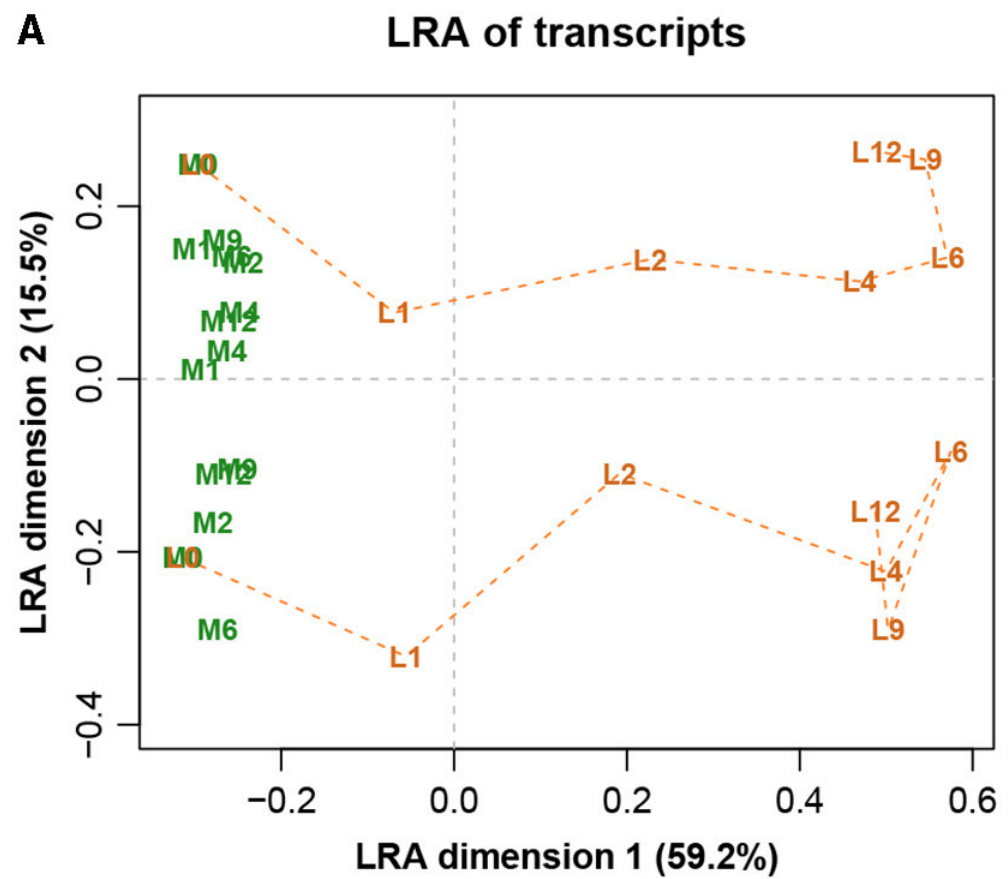
图八
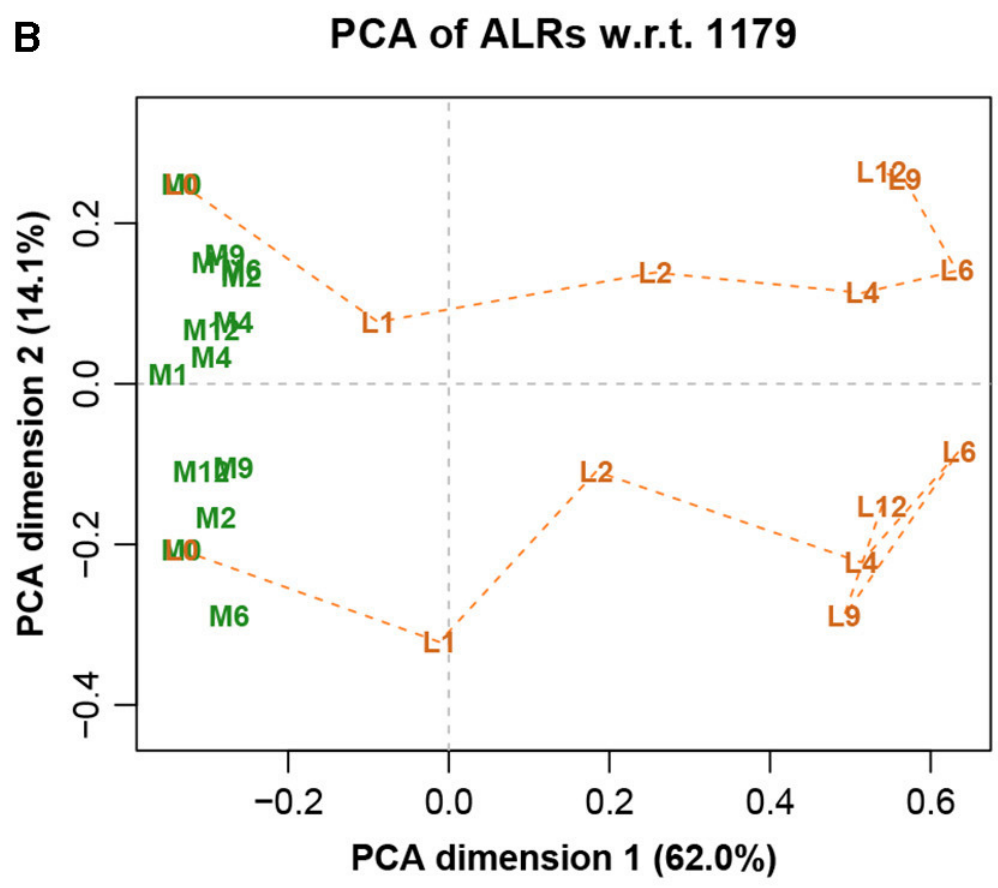
图九
3. 奶牛数据集
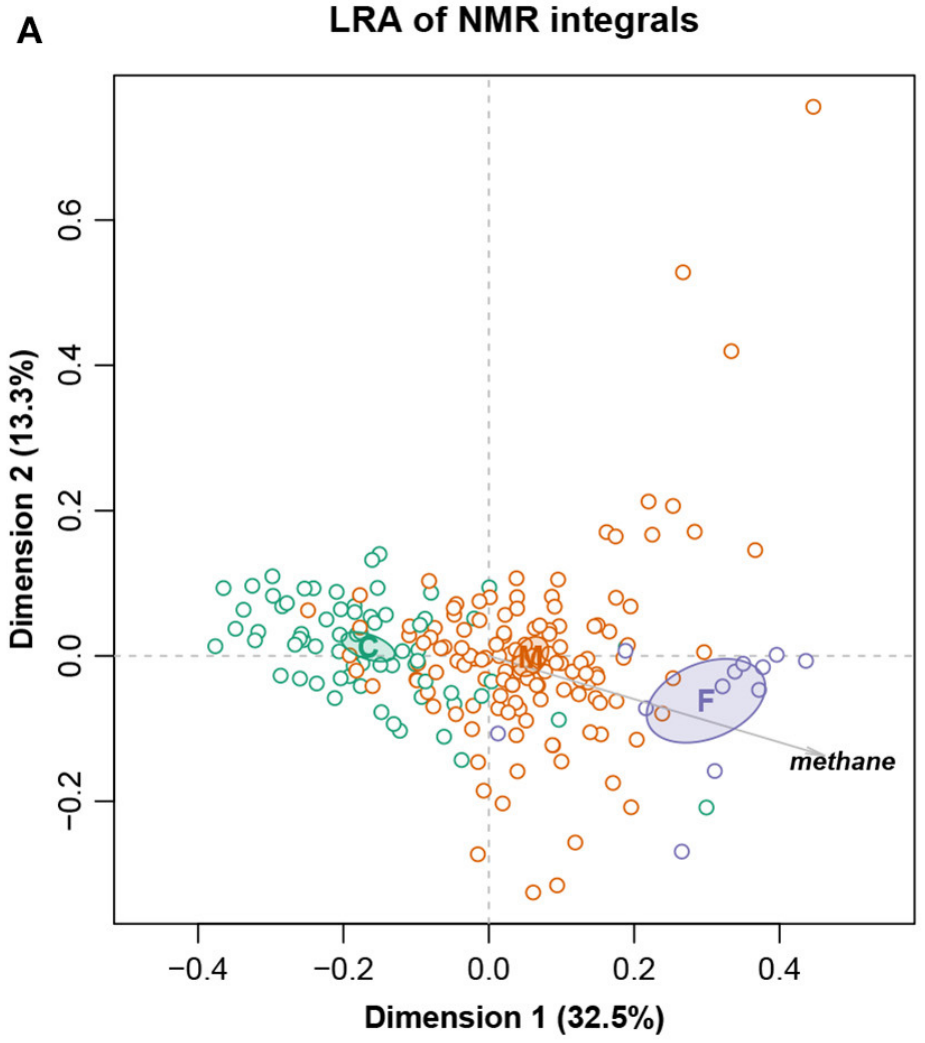
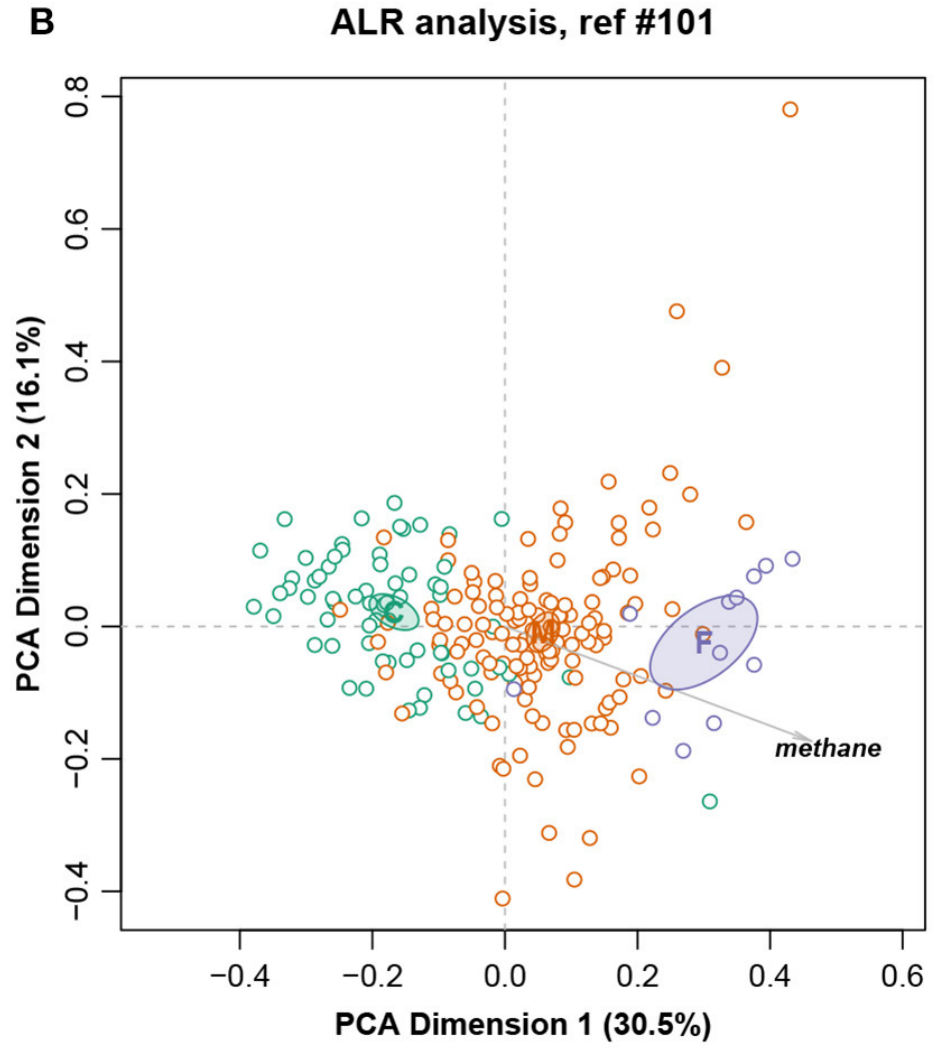
这是一个大小为211个样,127个特征的核磁共振强度数据集。样本被分成三个饮食组:精料组、混合组和饲草组,还测量了甲烷产量。
图十
图十一
总对数方差0.09128,Procrustes相关系数最高为0.9902,对应于编号101。图十展示完整数据集的LRA,图十一展示了编号101的ALR的PCA。标签C(精料)、M(混合)和F(饲料)。
03
结论
从以上三个数据集的验证分析不难看出,对于高维数据,使用ALR对数转换也能得到对全部特征使用CLR对数转换方法的结果,关键在于找到有效的参考特征(成分)。
文章中作者建议将其作为此类高维数据成分数据分析的第一步。作者公开了部分数据集的存放地址,以及用于数据处理的部分代码。可以自己尝试看看是否适用。
扩展:数据集位置及实用脚本
兔子数据集:
https://www.ebi.ac.uk/ena/browser/view/PRJEB46755
小鼠数据集:http://doi.org/10.5281/zenodo.3270954
其它数据集及脚本:https://github.com/michaelgreenacre/CODAinPractice
在这个github中有详细列出文中所使用的用于数据处理的各个R源码,以及目前这些数据处理的相关函数。
而这些脚本现已被整合为R包,easyCODA,可以从CRAN中直接下载。在Rstudio中调用“install.packages("easyCODA")”。
Tips
在对成分数据(composition data)进行分析时,通常会对原始数据进行矫正,也可以理解为一种标准化方法。比较常用的对数转换方法是CLR(Centered Log-Ratio),其次是ALR(Additive Log-Ratio,也就是文章主要推荐的方法)和ILR(Isometric Log-Ratio)。
每种方法都有优缺点,对于后续统计分析的适用程度,CLR>ALR>ILR。个人建议先使用CLR和ALR先对数据进行转换,然后使用PCA或其他降维分析方法查看其类群分布,搭配adonis查看其统计显著性水平。只要能达到预期结果就都能使用。如果CLR和ALR数据转换后结果差异不大,那推荐使用CLR。
参考文献:
Greenacre M, Martínez-álvaro M, Blasco A. Compositional Data Analysis of Microbiome and Any-Omics Datasets: A Validation of the Additive Logratio Transformation. Front Microbiol. 2021 Oct 11;12:727398. doi: 10.3389/fmicb.2021.727398.
本文转自:谷禾健康
https://wap.sciencenet.cn/blog-2040048-1312947.html
上一篇:[转载]你的焦虑可能与食品添加剂有关,警惕食品添加剂引起的微生物群变化
下一篇:[转载]存疑 | 《CELL》最新研究:自闭症与肠道菌群