博文
PacBio平台助力454和HiSeq测序数据组装获得斯氏按蚊基因组
|||
2014年9月23日,斯氏按蚊(Anopheles stephensi)基因组序列发表在Genome Biology上。文章结合Roche 454、illumina HiSeq 2000以及PacBio平台综合测序、组装的策略。
一、取材
印度斯氏按蚊(Anopheles stephensi)实验室内自然繁殖多代。取五十多只雄性斯氏按蚊和雌性斯氏按蚊提取基因组DNA和总RNA。gDNA提取试剂盒为Qiagen (Hilden, Germany) DNeasy Blood and tissue kit;总RNA提取试剂盒为mirVana RNA isolation kit (Life Technologies, Carlsbad, CA)。
二、基因组测序策略
1)454 FLX Titanium测序:1个shotgun文库SE测序;3Kb、8Kb、20Kb mate pair文库测序;测序深度为19.4×;
2)Illumina HiSeq 2000 100PE:雄性gDNA,1个~200bp shotgun文库测序1个lane;测序深度为86.4×;
3)PacBio v1测序:雄性gDNA,测序10个cell;测序深度为5.2×;
4)Sanger测序:7,263 BAC-ends。
三、基因组组装
1)454 reads结合Newbler单独组装;
2)454 reads和illumina raw reads结合Newbler进行组装:this resulted in a worse assembly than 454 alone;但是运用这个策略组装Solenopsis invicta基因组效果很成功;
3)先组装illumina reads,再将454 reads进行拼接:
a.先结合Celera assembler组装illumina reads获得Illumina pseudo-454 reads assembly:组装获得41,213 contigs spanning 212.8 Mb,该组装结果contig N50为16.8kb;
b.再将该结果通过Newbler 2.8和454 reads进行组装:组装获得23,595 scaffolds spanned 221 Mb,scaffold N50为1.34 Mb;
c.结合PacBio reads填补gaps:首先通过Celera pacBioToCa pipeline结合454 reads对PacBio raw reads进行错误校正,获得0.88 Gb校正好的PacBio reads;再结合Pbjelly填补gaps。一共补洞1310个,长度达5.4Mb。
d.结合BAC-ends进一步scaffolding:(BAC文库大小约120kb±70kb),结合Bambus scaffolder进一步将3,527 BAC-end pairs序列进行scaffolding,共获得46 links定位22 scaffolds, 将scaffold N50从1,378 kb增长至1,572 kb。
四、验证组装结果
1)结合CEGMA对组装结果中存在的真核生物核基因(Core Eukaryotic Genes)来验证组装结果:248个真核生物核基因中有96.37%是包含在该组装结果中的;97.89%显示为是部分被包含其中的。
2)BAC-ends验证:结合NUCMER将BAC-ends比对回组装结果进行验证,仅唯一比对、99% identity、 >95% coverage的才用于后续统计中,比对率为21.6%。
3)ESTs序列验证:ESTs序列是从NCBI和VectorBase中下载的,去除ESTs中的载体序列等污染序列后再结合GMAP比对至组装结果上,35,367 of 36,064 ESTs比对上,其中26,638至少覆盖95%(>98%identity)。

五、构建遗传图谱
1)A physical map of the An.stephensi genome was created from FISH on polytene chromosomes;
2)227 probes对86个scaffolds进行定位;被定位的86个scaffolds包含137.14Mb组装子序列信息,覆盖比例为62%。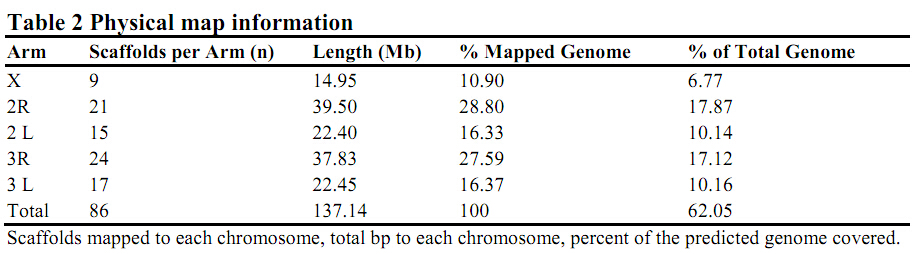
六、基因注释
通过同源比对注释和从头注释获得11,789各蛋白编码基因。
七、转录组测序
共11个总RNA样本开展转录组测序:0–1, 2–4, 4–8, and 8–12 hour post-egg deposition embryos, larvae, pupae, adult males, adult females, non-blood-fed ovaries, blood-
fed ovaries, and 24 hours post-blood-fed female carcasses without ovaries。
重点分析了与免疫、唾腺分泌有关的功能基因。
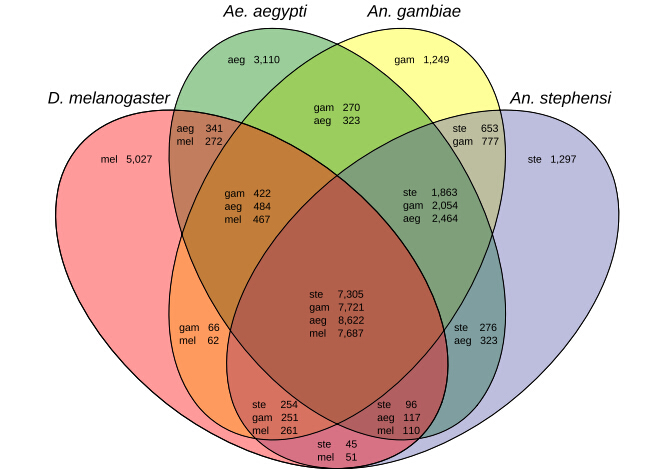
八、基因家族比较分析:An. stephensi and An. gambiae。
九、重复序列分析

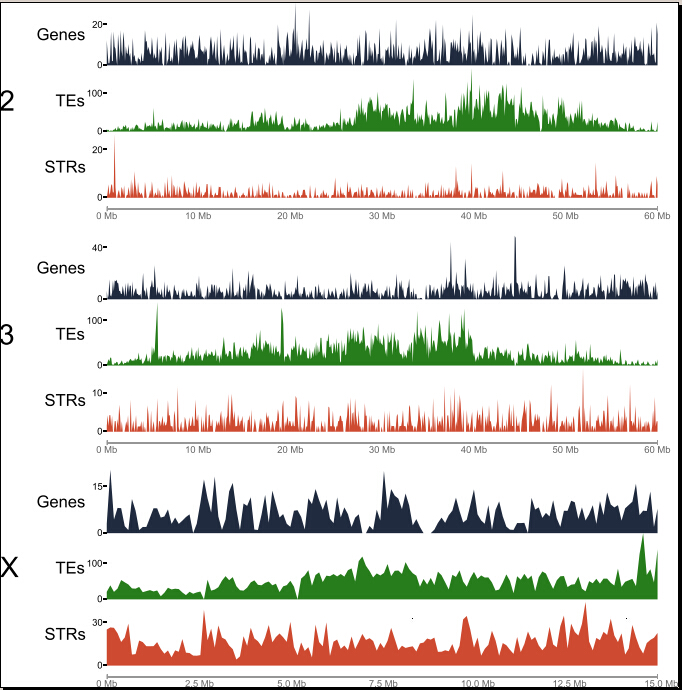
十、基因组图谱:包括基因、TEs、短串联重复序列基于遗传图谱在每条染色体上的分布情况
十一、染色体臂上异染色质的分布情况
十二、Y染色体分析
十三、基因共线性和进化分析
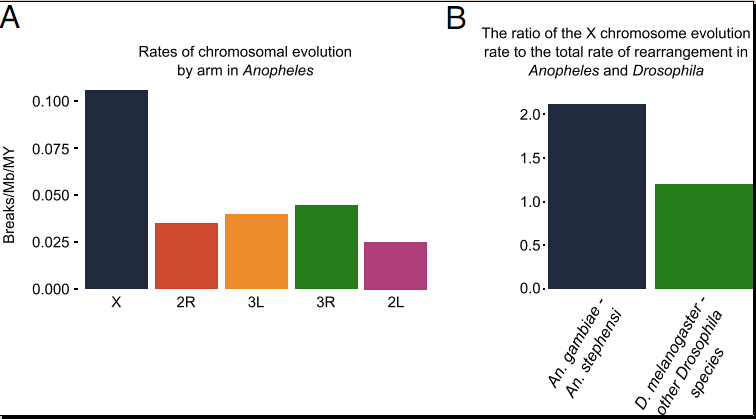
十四、比较果蝇和按蚊染色体的进化速率
十五、基因组遗传多样性分析
文献下载:Genome analysis of a major urban malaria vector mosquito, Anopheles stephensi.pdf
文献链接:http://genomebiology.com/2014/15/9/459/abstract。
https://wap.sciencenet.cn/blog-1333578-830755.html
上一篇:Macrogen将运用PacBio平台对黄种人基因组进行denovo测序组装
下一篇:千年基因将在全球华人遗传学大会上设展台,欢迎各老师到展台交流