博文
Phenomics | 复旦大学王瑞团队开发SIMPLE超快速单核苷酸变异精准识别新方法
|
该研究开发了体外PAM全面评估系统,对AapCas12b的PAM序列及反式切割偏好性进行了分析,构建了基于RPA-Cas12b的一锅式超快速分子检测技术,可在30分钟内精准识别单核苷酸变异(SNV),在耐药菌检测和癌症早筛领域实现重大突破。
文末点击“阅读原文”可在线阅读文章。

扫描二维码 | 下载PDF原文
论文DOI链接:
https://doi.org/10.1007/s43657-025-00240-x
论文引用格式:
Guo, K., Lai, Y., Han, Y. et al. SIMPLE: A One-Pot RPA-Cas12b Method for Single Nucleotide Variations Identification with an Expanded Scope and Clinical Applications. Phenomics (2025). https://doi.org/10.1007/s43657-025-00240-x
研究背景
临床诊断和生物技术领域亟需快速、可靠的核酸检测方法。当前金标准PCR技术依赖精密仪器,难以满足即时检测(POCT)需求;等温扩增技术(如RPA、LAMP)虽简化了设备,但在单核苷酸变异(SNV)检测中面临灵敏度低、特异性不足的局限。
CRISPR系统作为新一代检测技术,其应用受到两大关键限制:
(1)原间隔序列邻近基序(PAM)限制:Cas酶需识别特定PAM序列(如TTTN),严重制约了可检测靶点范围;
(2)操作复杂性:现有CRISPR方法(如DETECTR/SHERLOCK)需分步进行扩增与检测,增加污染风险且难以实现SNV的高特异性快速检测。
此外,耐药菌感染和癌症热点突变的精准诊断需求日益迫切,亟需开发一种突破PAM限制、可一步完成SNV检测的新技术。
研究结果
本研究开发了体外PAM全面评估系统,并通过该系统全面解析AapCas12b的PAM识别特性(图1a),发现其不仅能识别经典TTN序列,还能有效识别非经典PAM(如ATN/GTN)(图1b, c)。荧光验证实验证实ATN在单管检测中性能接近TTN(图1e),显著将CRISPR可靶向序列范围扩展,为突破传统PAM限制提供基础。
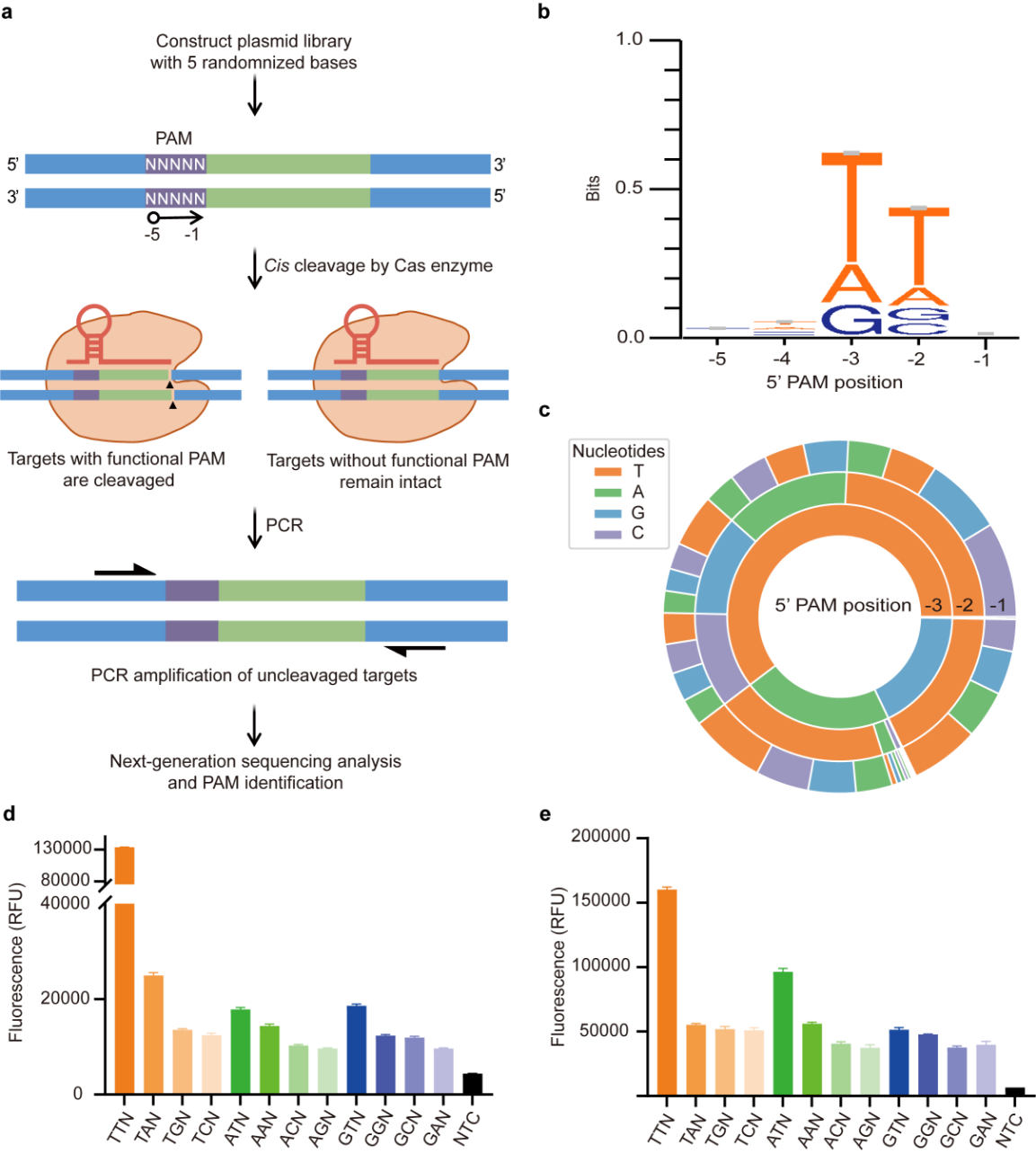
图1 Cas酶体外PAM全面评估系统建立与应用
此外,研究者系统评估crRNA间隔序列长度对单碱基识别的影响(图2a),发现17nt长度间隔序列在15-20nt范围内具有最优特异性(图2d)。其错配耐受性显著低于其他长度,可在单管反应中实现单核苷酸变异(SNV)的精准区分,为高特异性检测奠定基础。
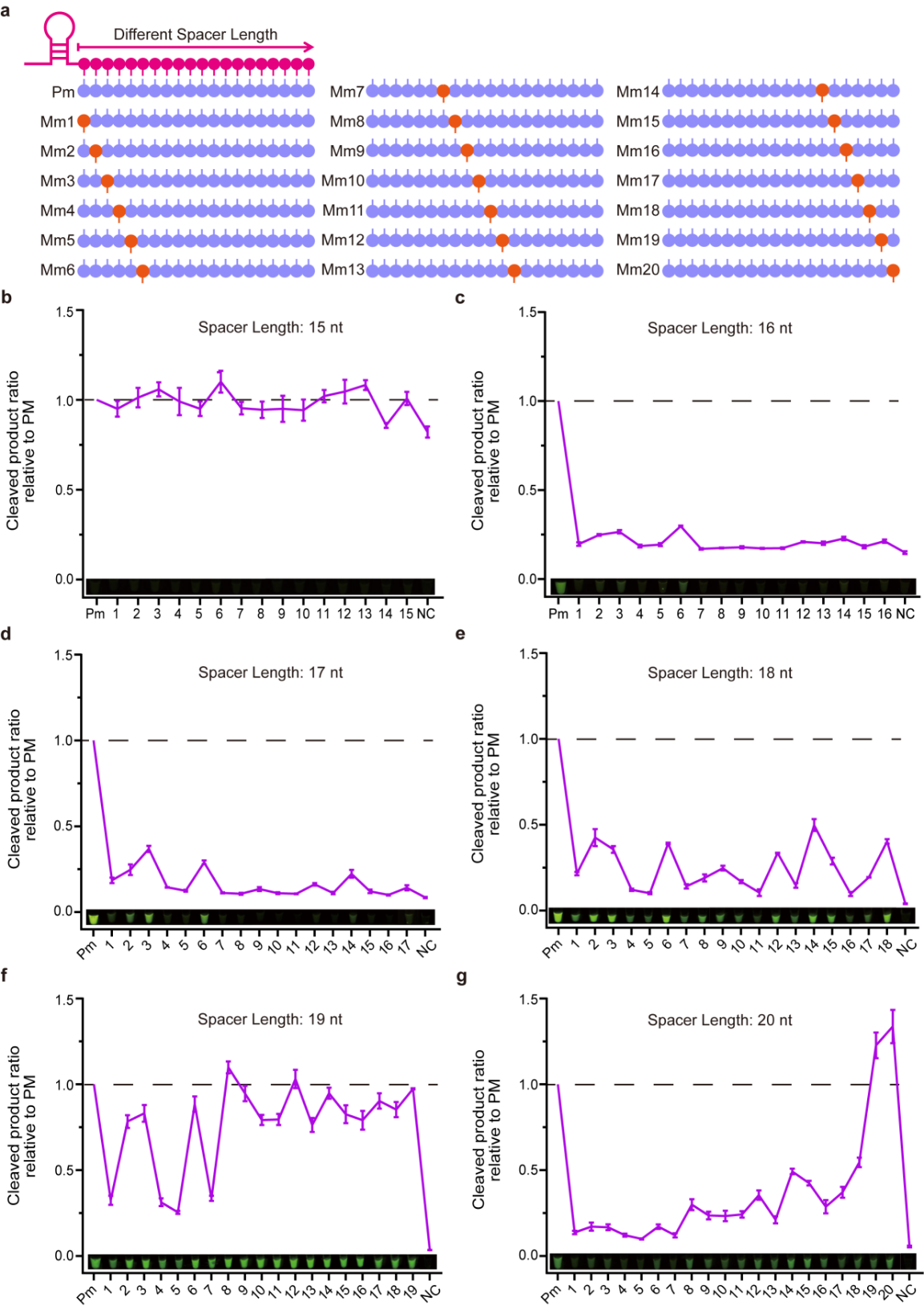
图2 全面评估Cas12b的单碱基识别特异性
进一步研究者揭示了AapCas12b对报告探针的反切偏好性:对C-rich探针的切割速率显著高于其他类型(图3c),且8nt长度探针在6-20nt长度探针范围内兼具高裂解速率与低背景信号(图3e, f)。最终选定8nt长度的 C-rich探针作为最优报告探针,确保高检测灵敏度和低信噪比。
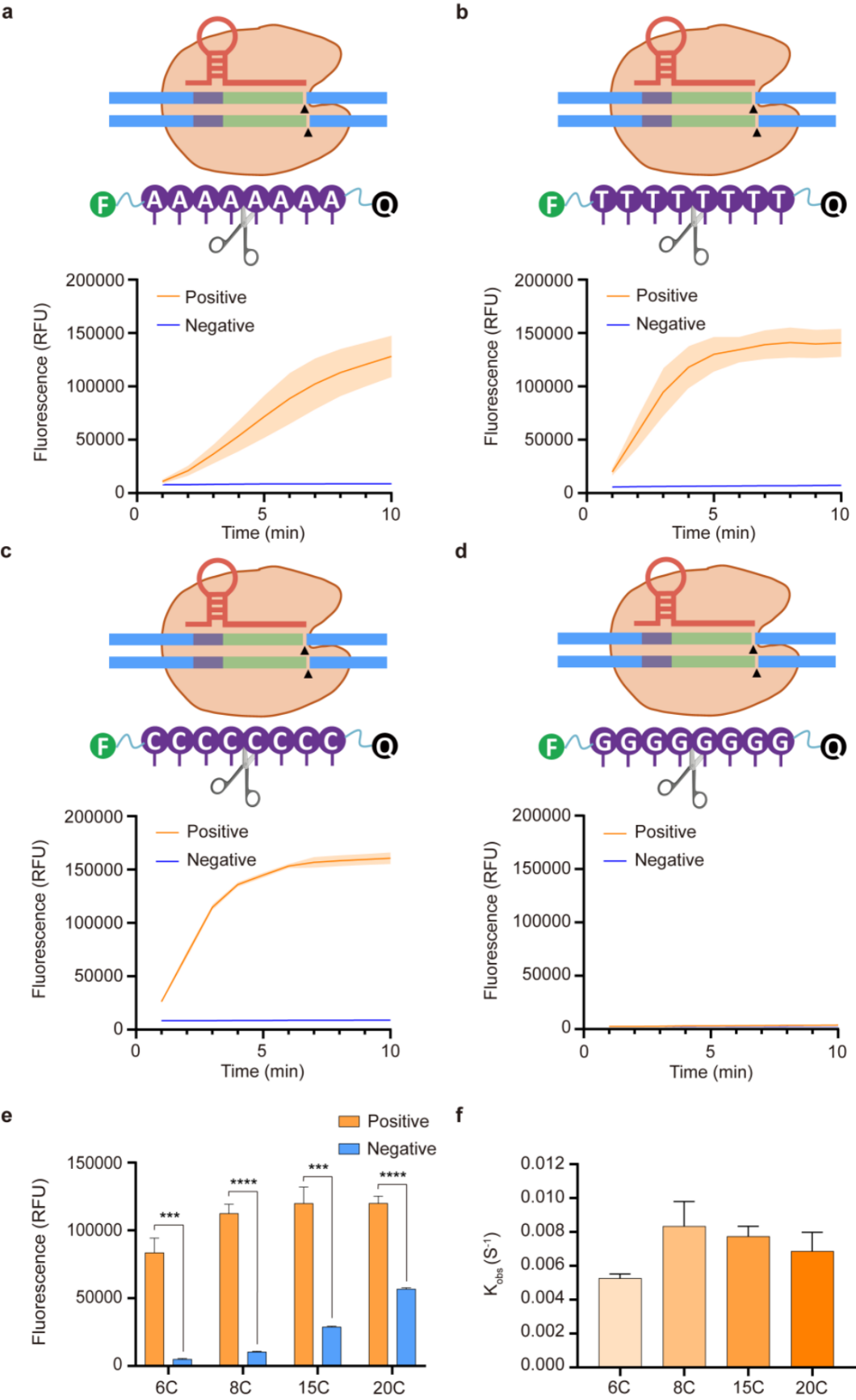
图3 AapCas12b的反式切割偏好性探究
成功建立CRISPR一体化多重检测体系(图4a):通过混合三种crRNA(靶向幽门螺杆菌A2142G/A2142C/A2143G耐药突变),实现了30分钟内对三种SNV耐药突变位点的同步检测(图4b)。检测灵敏度达10拷贝/反应(图4c),并且可精准区分野生型与其他病原体(图4d, e),为耐药菌感染与耐药鉴定提供了POCT解决方案。
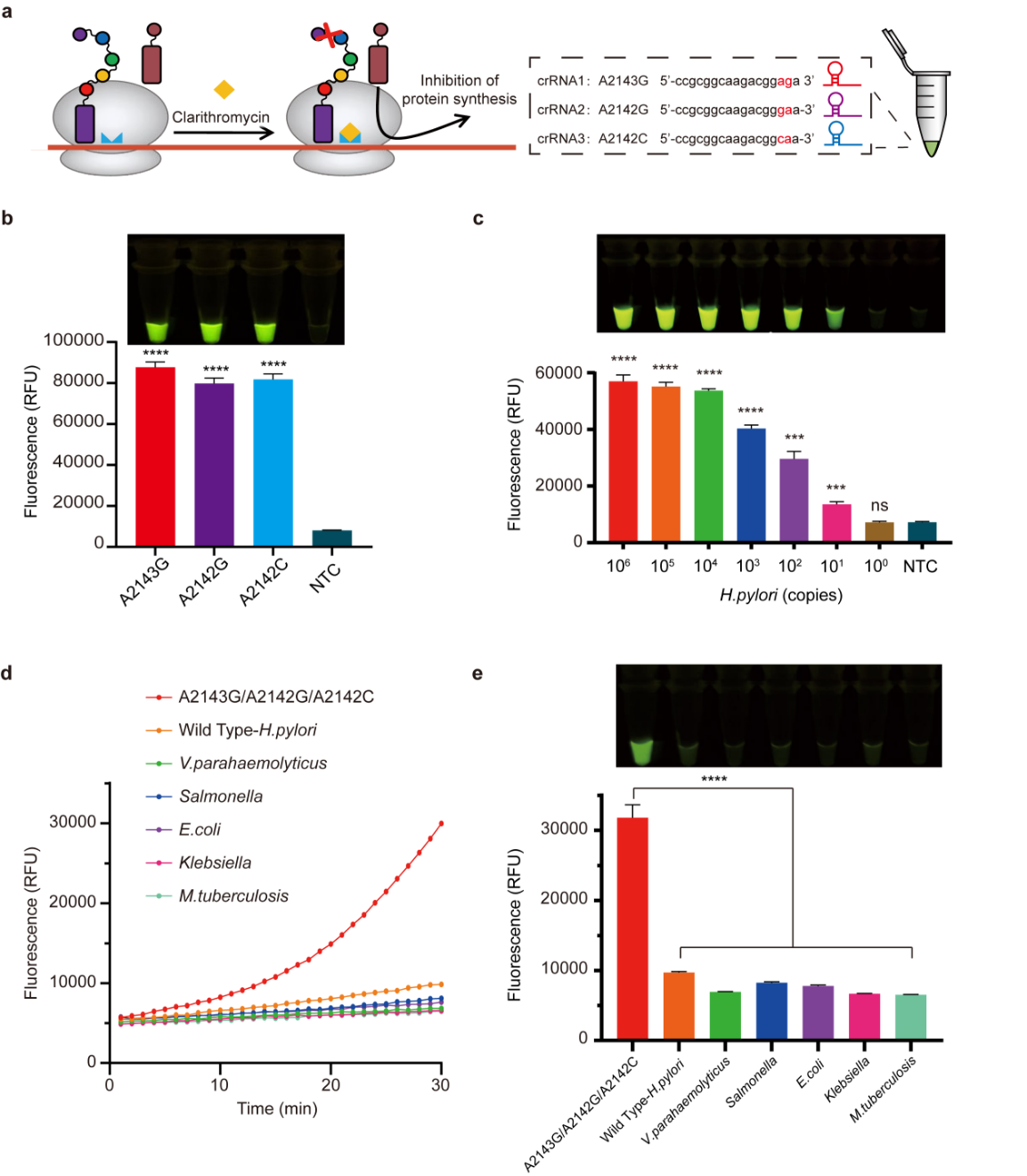
图4 CRISPR一体化方法灵敏度和特异性评估
研究者进一步将SIMPLE用于实际样本验证,对幽门螺杆菌患者粪便样本进行了检测,检测时间约30分钟(图5a)。将20例幽门螺杆菌感染患者的粪便样本进行热裂解快速处理,然后进行SIMPLE检测,检测结果与Sanger测序结果100%一致(图5b)。NGS进一步证实SIMPLE方法无假阴性,证明其在真实临床场景中检测耐药突变的可靠性。
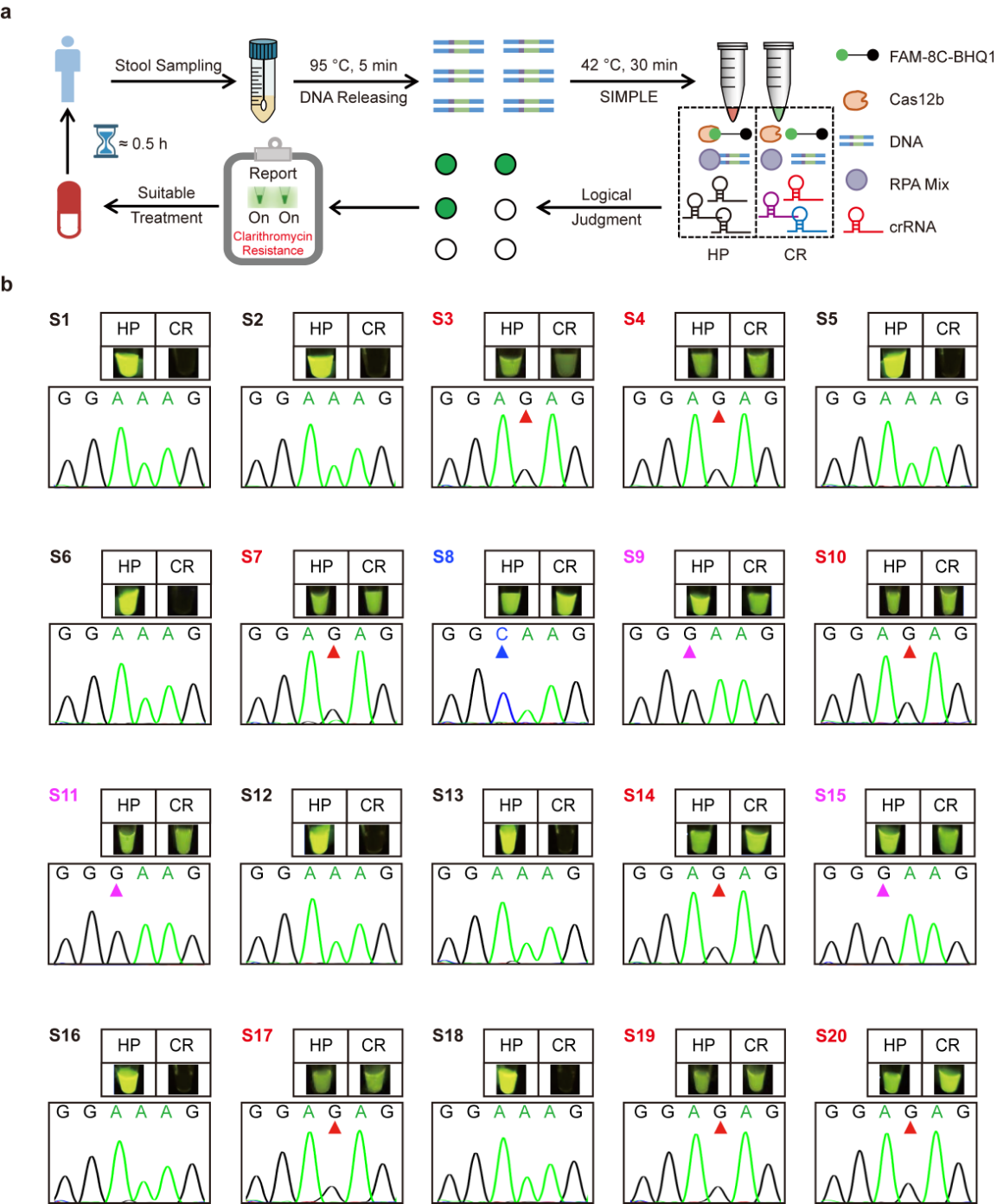
图 5 SIMPLE方法用于幽门螺杆菌耐药检测
为了拓展SIMPLE的检测范围,研究者利用非经典PAM(ATN)实现了对肿瘤热点突变的检测。针对白血病(AML)FLT3基因的 D835Y突变,检测灵敏度达到100 copies(图6b),并且实现了低至1%的低频突变检测(图6c)。采用SIMPLE对临床样本的检测结果与NGS结果100%一致(最低检出2.6%突变),并且检测灵敏度高于Sanger测序(图6d),凸显了SIMPLE方法在癌症早筛中的应用前景。
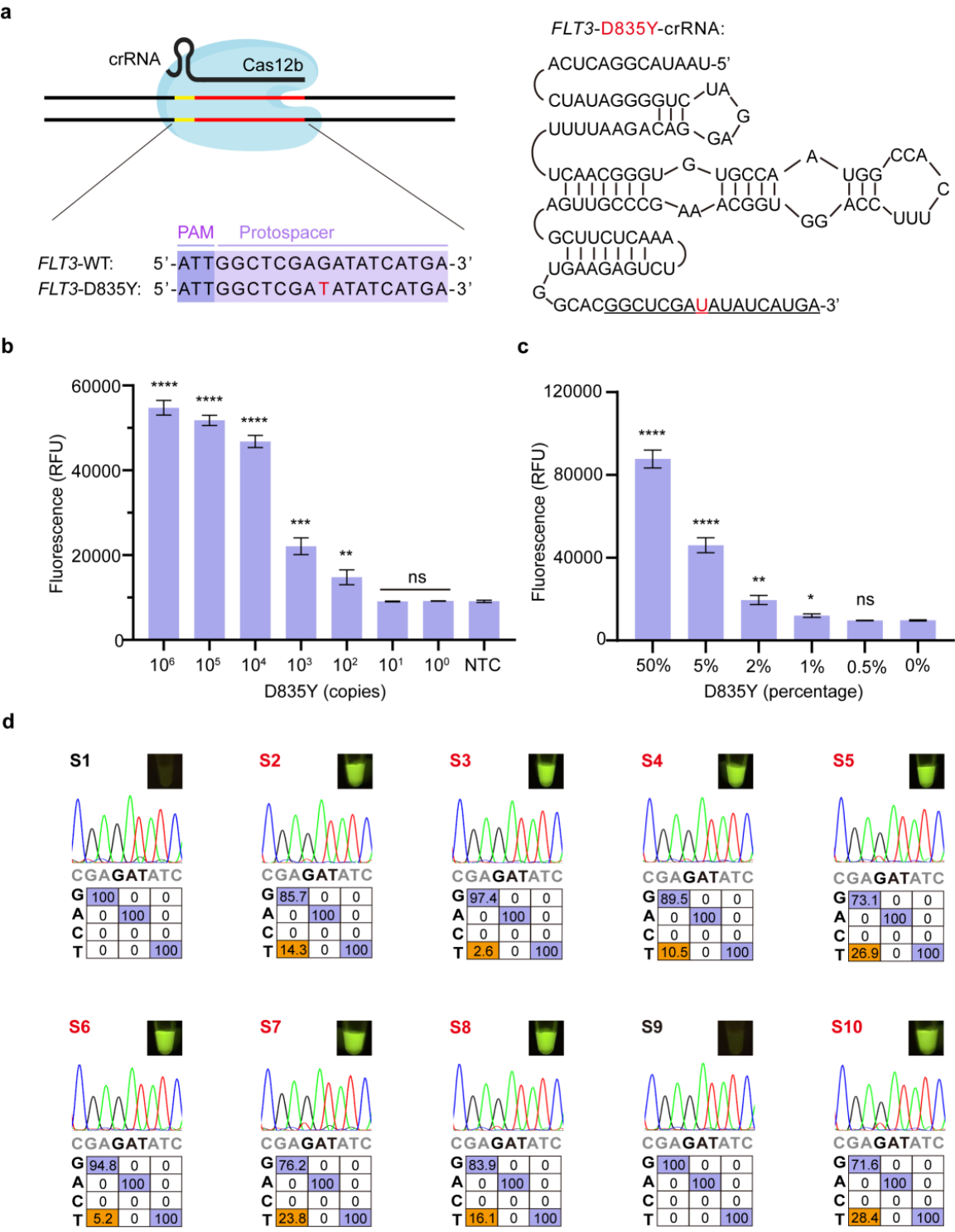
图 6 SIMPLE方法用于癌症热点突变检测
研究结论
本研究开发了体外PAM全面评估系统,并且采用此系统发现AapCas12b可高效识别非经典PAM,显著扩展了Cas酶的靶向和检测范围;此外,本研究发现Cas酶的单碱基识别特异性与crRNA长度密切相关,并且Cas酶对报告探针具有碱基偏好性。采用17nt 间隔序列长度的crRNA实现了单碱基分辨率检测,并采用8nt C-rich探针显著提升了信噪比。开发的SIMPLE方法可在30分钟内完成一步法检测。研究者成功将其用于幽门螺杆菌克拉霉素耐药检测,检测结果与二代测序具有100%一致性,并且将其拓展用于癌症热点突变检测,在白血病患者样本中能够检测低至1%的低频突变。因此,所开发的SIMPLE方法为病原体耐药鉴定和癌症早筛提供了POCT分子诊断平台,具有广阔的应用前景。
Abstract
Rapid and reliable nucleic acid detection methods are essential in clinical diagnostics and biotechnology. The clustered regularly interspaced short palindromic repeats (CRISPR) system is emerging as a next-generation nucleic acid detection technology, offering versatility, convenience and rapid detection. However, CRISPR methods are significantly limited by the protospacer adjacent motif (PAM) sequence, and achieving a one-pot reaction for detecting single nucleotide variations (SNVs) within a short time still remains challenging. Here, we developed a comprehensive method for screening PAM sequences, which significantly expands the CRISPR detection scope. Additionally, we also proposed a one-pot CRISPR method, termed "SIMPLE," capable of identifying SNVs within 30 min. We applied the SIMPLE method to the clinical diagnostics of drug-resistant bacteria and the screening of cancer hotspot mutations. The SIMPLE method successfully detected drug-resistant bacteria mediated by canonical PAM TTN sequence with a sensitivity of 10 copies per reaction and achieved 100% consistency with next-generation sequencing results. Furthermore, the SIMPLE method proved effective in detecting hotspot mutations in cancer, even at a low mutation rate of one percent in the presence of high background interference mediated by non-canonical PAM ATN sequence. Therefore, the SIMPLE method not only expands the CRISPR detection scope but also offers a one-pot reaction with high specificity for SNVs identification, making it a promising tool for next-generation molecular diagnostics.
作者简介
通讯作者

王瑞,上海市“启明星”人才,复旦大学青年副研究员。主要研究领域为:新型分子诊断方法开发和智能生物传感器构建。已主持国家级和省部级项目8项,包括:国自然面上项目,国自然青年项目,上海市“启明星”人才项目,上海市“科技创新行动计划”等。已经在本领域Top期刊Cell Discovery,Biosensors and Bioelectronics,TrAC Trends in Analytical Chemistry,Analytical Chemistry 等期刊上发表SCI论文46篇,其中以第一作者或通讯作者(含共同)发表SCI论文27篇,他引2000余次,2篇被评为“ESI高被引文章”,已申请发明专利16项。
第一作者

郭凯明,复旦大学人类表型组研究院硕士研究生。主要研究领域为:基于CRISPR/Cas系统的生物传感以及基因编辑工具的开发。在Biosensors and Bioelectronics,Theranostics,Analytical Chemistry ,Phenomics等期刊上发表SCI论文6篇,其中以第一作者发表SCI论文2篇,已申请发明专利3项。
https://wap.sciencenet.cn/blog-3558836-1491829.html
上一篇:Phenomics | 南京中医药大学高梓珊团队揭示针刺特定穴位调节无氧糖酵解和线粒体功能从而缓解偏头痛的经穴特异性代谢
下一篇:Phenomics | 中南大学湘雅医院陈泽宇教授团队综述:新型成像技术在生物医学中的应用
全部作者的其他最新博文
- • Phenomics | 中山大学肿瘤防治中心曾木圣院士团队发表综述:肿瘤诊疗新靶点,整合素α6靶向技术引领精准医疗新时代
- • Phenomics | 上海交通大学医学院张孝勇教授团队开发磁共振成像降噪新方法,有潜力改善脑小血管病的诊断效能
- • Phenomics | 复旦大学倪挺教授团队揭示基因内含子多聚腺苷酸化调控细胞衰老新机制
- • Phenomics | 加拿大阿尔伯塔大学和复旦大学联合开发基于文本、音频和视频的多模态抑郁症检测与评估方法
- • Phenomics | 翟振国/蒋太交/张鹏团队合作开发自适应多基因风险评估模型提升汉族人群静脉血栓栓塞症的风险预测能力
- • 祝贺 | 《表型组学(英文)》成功入选2025年“中国科技核心期刊”